A TaqMan RT-PCR Method for Detecting Porcine Circovirus 3
-
摘要:目的 建立检测猪圆环病毒3型(Porcine circovirus 3, PCV3)感染的TaqMan实时荧光定量PCR方法。方法 通过分析明确PCV3复制相关蛋白Rep基因特征,设计针对Rep基因的特异性引物和探针,经条件优化后建立检测PCV3感染的TaqMan实时荧光定量PCR方法。结果 建立的TaqMan实时荧光定量PCR方法敏感性好,最低检测限为42.2拷贝·μL−1;特异性强,对常见猪群传染病均无交叉反应;重复性好,组内变异系数和组间变异系数均在1.48%以内。对福建省2014年至2018年保存的193份组织样品进行检测发现,PCV3在福建省猪群中存在较高的阳性感染率(65.80%),且和PCV2混合感染率较高(52.85%)。结论 本方法的建立为开展Rep基因在PCV3复制和感染过程中的作用机制提供检测方法。
-
关键词:
- 猪圆环病毒3型 /
- Rep基因 /
- TaqMan实时荧光定量PCR方法 /
- 检测
Abstract:Objective A TaqMan RT-PCR method was established for detecting porcine circovirus 3 (PCV3) infection in swine.Method Specific primers and probe were designed by Oligo 7 software targeting the Rep gene after genetic comparison.Result The established TaqMan RT-PCR method could detect 42.2 copies·uL−1 with no positive signal on common porcine infectious diseases indicating high specificity of the methodology. On the constructed positive plasmids, the coefficients of variation for the intra- and inter-assays were less than 1.48% showing a high detection reproducibility. From the 193 clinical specimens collected in Fujian from 2014 to 2018, the newly developed method showed a high prevalence of PCV3 at 65.80% and high co-infection with PCV2 at 52.85%.Conclusion The established TaqMan RT-PCR method was made available for studying the function of Rep in PCV3.-
Keywords:
- Porcine circovirus 3 /
- Rep gene /
- TaqMan RT-PCR method /
- detection
-
0. 引言
【研究意义】截至目前,猪群中发现存在3种圆环病毒:猪圆环病毒1型(Porcine circovirus 1, PCV1)[1]、猪圆环病毒2型(Porcine circovirus 2, PCV2)[2]和近年来新发的猪圆环病毒3型(Porcine circovirus 3, PCV3)[3-4]。根据国际病毒分类委员会的最新分类,PCV1、PCV2和PCV3均属于圆环病毒科(Circoviridae)圆环病毒属(Circovirus)成员。PCV3在美国、中国、日本、韩国、瑞典、俄罗斯、泰国、巴西、丹麦、意大利、西班牙等猪群中均有报道[5]。此外,流行病学调查还发现猪群中存在多种其他病原和PCV3混合感染,且PCV3在野猪中也广泛流行[6-7]。一般认为,PCV1对猪群不致病,而PCV2是当前猪群中最为重要的致病病原之一;但是关于PCV3是否存在致病性,目前还存在一定的争议,这主要和当前缺乏对PCV3的有效培养有关[8-10]。因此,亟待开发一种特异、敏感、快速的检测方法用于开展PCV3流行病学调查和致病机制研究。【前人研究进展】目前,PCV3的检测已见有SYBR I实时荧光定量PCR方法[11]、重组酶扩增方法(Recombinase polymerase amplification, RPA)[12]、环介导等温扩增(Loop-mediated isothermal amplification, LAMP)[13-14]和酶联免疫吸附测定法(Indirect enzyme-linked immunosorbent assay, ELISA)[15]、数字PCR方法(Droplet Digital PCR)[16]等。对GenBank中PCV3序列进行分析发现,PCV3基因组全长在1 999~2 001 bp,编码有两个大的开放阅读框(Open reading frame, ORF):位于基因组左侧的ORF1编码病毒复制相关蛋白(Rep)和位于病毒基因组右侧的ORF2编码病毒抗原相关蛋白(Cap)[17-18]。序列分析比较发现, 不同PCV3来源Rep蛋白和圆环病毒属其他病原该基因同源性均较低,和蝙蝠源圆环病毒(Bat circovirus)最高,约为54%~55%,和PCV2的氨基酸同源性仅为48%左右。研究还进一步发现,PCV3的Rep蛋白上有3个和病毒滚环复制相关的结构域:FTINN、HLQG和YCKK[3-4]。【本研究切入点】以PCV3中的Rep基因建立特异性检测方法具有一定实用性和可行性。【拟解决的关键问题】本研究通过分析PCV3福建株Rep基因特征,建立特异性检测PCV3核酸感染的TaqMan实时荧光定量PCR方法,为后续开展Rep基因在PCV3复制和感染过程中的作用机制提供研究手段。
1. 材料与方法
1.1 试验毒株
PCV1、PCV2、猪细小病毒(Porcine parvovirus,PPV)、猪伪狂犬病毒(Porcine pseudorabies virus, PRV)、变异型猪伪狂犬病毒(Porcine pseudorabies virus variants, v-PRV)[19]、猪巨细胞病毒(Porcine cytomegalovirus, PCMV)[20]由福建省农业科学院畜牧兽医研究所猪病研究室分离、鉴定和保存。
PCV3(FJ2017株)、猪博卡病毒(Porcine bocavirus, PBoV)[21]、猪细环病毒1a型(Porcine Torque teno sus virus type 1a, PTTSuV 1a)和猪细环病毒1b型(Porcine Torque teno sus virus type 1b, PTTSuV 1b) [22-23]阳性病料均由福建省农业科学院畜牧兽医研究所猪病研究室鉴定和保存。
1.2 主要仪器和试剂
实时荧光定量试剂FastFire qPCR PreMix(Probe)(FP208)、PCR扩增试剂2×Taq PCR MasterMix Ⅱ、病毒基因组DNA/RNA提取试剂盒、普通琼脂糖凝胶DNA回收试剂盒、快速质粒小提试剂盒均购自天根生化科技(北京)有限公司;pMD 18-T、DL2000 Marker均购自宝生物工程大连有限公司;其他试剂均为商业公司购买的分析纯。
1.3 PCV3 Rep基因的克隆和阳性标准品的建立
按照病毒基因组DNA/RNA提取试剂盒说明书操作,提取PCV3(FJ2017株)的核酸DNA,利用针对Rep基因的特异性引物进行扩增获得PCV3完整的Rep基因全长。上游引物PCV3-Rep-F1:5′-GTCCGGA、GGGAAAGCCCGAAAC-3′和下游引物PCV3-Rep-R1:5′- TCAATAGTTTATTGGATACACTGT-3′,预期扩增长度为891 bp。PCR扩增体系配置如下(40 μL反应体系):2×Taq PCR MasterMix Ⅱ反应液20 μL、上游引物(PCV3-Rep-F1,10 μmol·mL−1)和下游引物(PCV3-Rep-R1,10 μm·mL−1)均1 μL、提取的核酸DNA模板2 μL,补充灭菌去离子水至终体积40 μL。PCR扩增反应条件:94 ℃ 3 min后进行PCR循环(共进行35个循环),条件为94 ℃ 30 s、56 ℃ 30 s、72 ℃ 55 s。PCR循环结束后,72 ℃延伸10 min。对目的片段使用普通琼脂糖凝胶DNA回收试剂盒切胶回收后克隆到pMD18-T 克隆载体上,转化感受态细胞后,随机选取8个克隆利用快速质粒小提试剂盒提取相应的质粒进行鉴定。经序列测定验证BLAST分析后,将符合预期的质粒作为本试验的阳性标准品质粒(记为PCV3-Rep)。将获得的阳性标准品用超微量紫外可见分光光度计对阳性质粒进行定量,计算拷贝数并进行10倍连续稀释(浓度分别为4.22×107~4.22×100 拷贝·μL−1),置于−20 ℃备用。
1.4 基于TaqMan探针检测PCV3实时荧光定量PCR方法的建立
1.4.1 特异性引物和探针的设计
将PCV3(FJ2017株)的Rep基因序列和GenBank中登录的PCV3其他毒株的相关基因序列分析比较,选取保守区作为特异性的引物和探针的设计区域。利用引物设计软件Oligo v7.37 进行引物和探针设计,上游引物TaqMan-PCV3-F:5′-GGTGGGATGGTTATAATG-3′、下游引物TaqMan-PCV3-R:5′-TAGCCACAAAATTAACAAAC-3′、探针引物TaqMan-PCV3-P:5′-CACCCTTAACAGGAACCCTCAGA-3′,其中5′端以FAM标记,3′端Eclipse标记。预期扩增长度为141 bp,上述引物和探针均在宝生物工程大连有限公司合成。
1.4.2 反应体系的建立和条件优化
采用FastFire qPCR PreMix(Probe)推荐的25 μL配置TaqMan实时荧光定量PCR方法的反应液。以出现最高的荧光值(ΔRn)、最小的Ct值为指标,对退火温度(54~64 ℃)、不同引物终浓度(0.1、0.2、0.4、0.6、0.8、1.0 μmol·L−1)和不同探针终浓度(0.05、0.1、0.25、0.50 μmol·L−1)进行条件优化,同时设NTC(No template control)对照。
1.4.3 标准曲线建立
以连续10倍系列稀释(浓度分别为4.22×107~4.22×100 拷贝·μL−1)的标准品质粒(PCV3-Rep)为模板,以1.4.2优化条件进行扩增,获得扩增动力学曲线。以阳性标准品(PCV3-Rep)拷贝数的常用对数为横坐标,以循环阈值(Ct值)为纵坐标,计算出建立的TaqMan实时荧光定量PCR方法的标准曲线,获得本方法的标准线性回归方程。
1.4.4 特异性检测
用病毒基因组DNA/RNA提取试剂盒提取PCV1、PCV2、PPV、PRV、v-PRV、PCMV、PBoV、PTTSuV 1a和PTTSuV 1b的基因组DNA,经超微量紫外可见分光光度计进行定量后作为特异性检测试验对照。以1.4.2优化条件分别检测PCV1、PCV2、PPV、PRV、v-PRV、PCMV、PBoV、PTTSuV 1a和PTTSuV 1b提取的核酸DNA,以标准品质粒(PCV3-Rep)为阳性对照,以灭菌去离子水为阴性对照,评价建立的TaqMan实时荧光定量PCR方法的特异性。
1.4.5 变异系数测定
将阳性标准品质粒(PCV3-Rep)设3个重复(含量分别为4.22×105、4.22×103和4.22×101拷贝·μL−1),用建立的基于TaqMan实时荧光定量PCR方法进行特异性检测,计算组内(Intra-group)变异系数。将该标准阳性标准质粒置于−20 ℃冰箱保存,每隔两周重检一次,连续检测3次,计算其组间(Inter-group)变异系数。
1.5 临床样品检测
对本研究室保存于−70 ℃冰箱的193份2014–2018年收集的来自福建省多地市的猪临床组织样品,经病毒基因组DNA/RNA提取试剂盒提取相应的核酸DNA后,利用建立的基于TaqMan实时荧光定量PCR方法进行PCV3感染的特异性检测。为明确相关样品中PCV2的感染情况,本研究同时对这193份样品,利用前期建立的PCV2特异性TaqMan实时荧光定量PCR方法进行平行检测[24]。
2. 结果与分析
2.1 Rep基因的序列分析
本研究克隆获得的PCV3(FJ2017株)Rep基因全长为891 bp,编码完整的296个氨基酸(aa),其等电点(Isolectric point)为8.752,GenBank登录号为MF072716。核苷酸同源性分析发现,不同PCV3来源Rep基因同源性均较高,在99.6%~100%。将其和GenBank中的PCV3其他代表株进行遗传进化分析,采用MEGA7软件,绘制遗传进化树采用NJ法(Neighbor-Joining Methods),计算1 000次重复(Bootstrap=1 000)。结果可见,不同地区、不同来源和不同分离时间PCV3代表株Rep基因相互之间较保守(图1)。
2.2 实时荧光定量PCR检测方法的反应条件和反应体系
优化的TaqMan实时荧光定量PCR检测条件:98 ℃ 30 s后进行循环,95 ℃ 5 s、 60 ℃ 15 s,进行40个循环。优化出的25 μL最佳反应体系为体系:2×FastFire qPCR PreMix(2×)12.5 μL、上/下游引物(10 μmol·L−1)各0.75 μL、探针(10 μmol·L−1)0.5 μL、阳性标准品稀释液2 μL、补水至终体积25 μL。
2.3 实时荧光定量PCR检测方法的扩增曲线和标准曲线
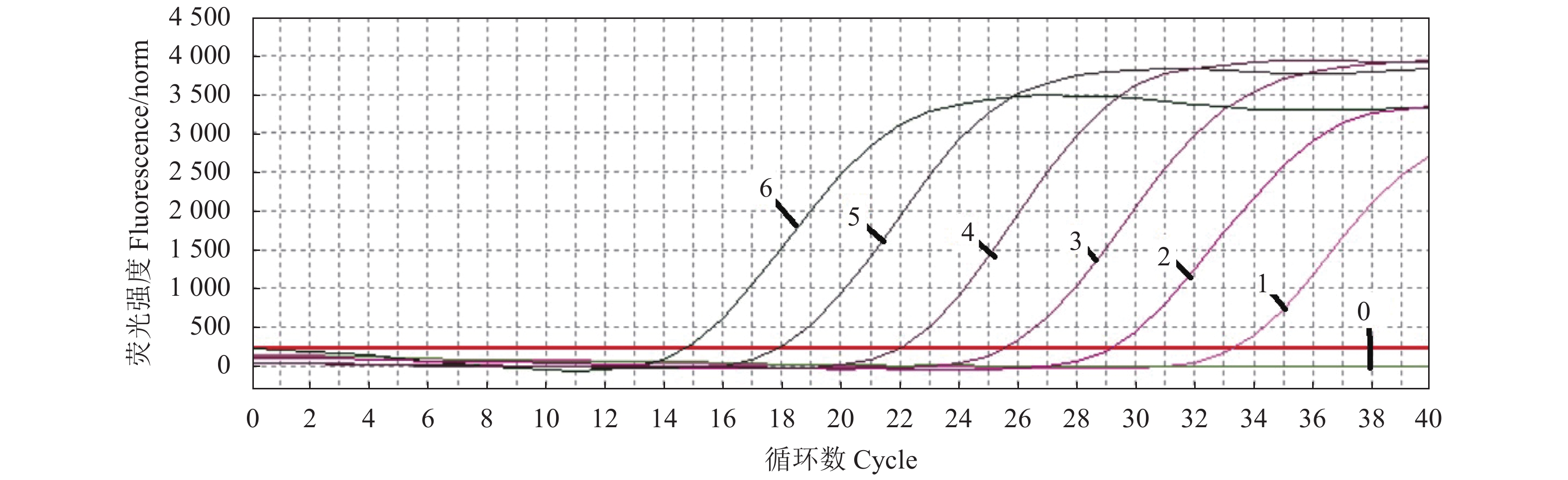
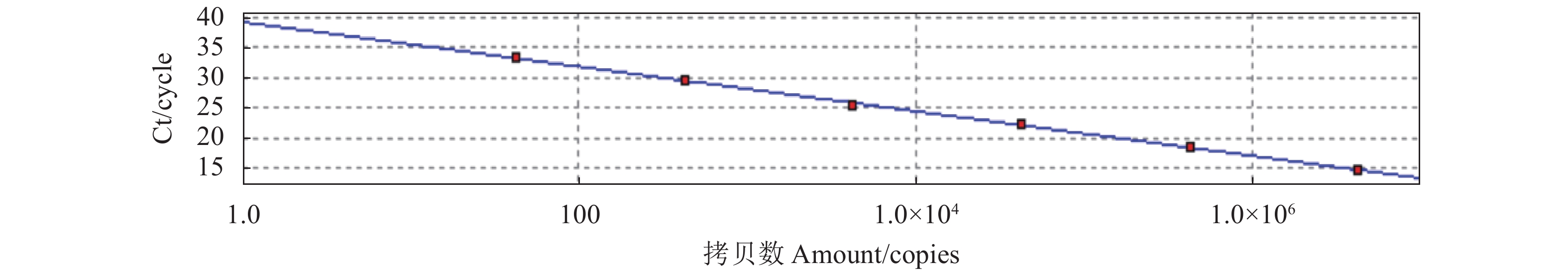
TaqMan实时荧光定量PCR方法检测Rep基因为4.22×101 ~4.22×106 拷贝·μL−1有很好的线性关系,检测的最低值为42.2拷贝·μL−1(图2)。所获得标准曲线斜率(slope)为−3.413,Y轴截距(Y-intercept)为38.39,相关系数(R2)为0.999(图3)。
2.4 实时荧光定量PCR检测方法的特异性
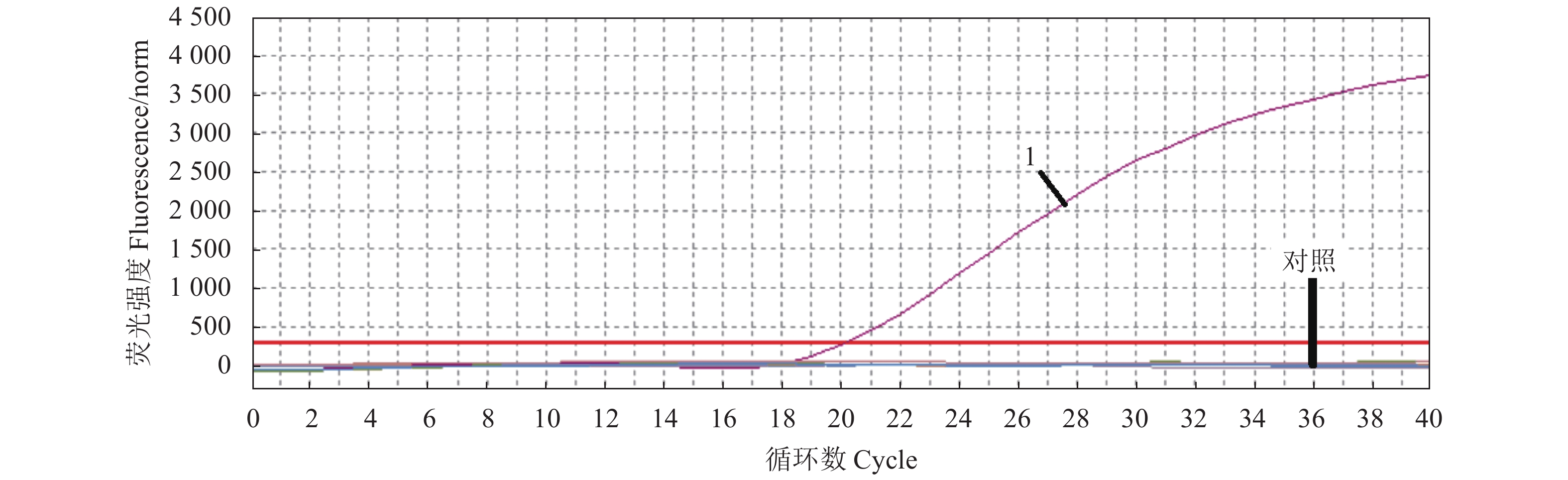
本研究建立的TaqMan实时荧光定量PCR仅对PCV3检测出现阳性扩增信号,对猪群其他常见病原(例如PCV1、PCV2、PPV、PRV、v-PRV、PCMV、PBoV、PTTSuV 1a和PTTSuV 1b)检测均为阴性(图4)。
![]() 图 4 实时荧光定量PCR检测方法的特异性注:1:PCV3阳性;对照:PCV1、PCV2、PPV、PRV、v-PRV、PCMV、PBoV、PTTSuV 1a和PTTSuV 1b,样品均为阴性,无荧光扩增信号。Figure 4. Specificity of TaqMan RT-PCR methodNote: 1: PCV3 positive control; experimental control groups: PCV1, PCV2, PPV, PRV, v-PRV, PCMV, PBoV, PTTSuV 1a, and PTTSuV 1b (all negative with no fluorescent signal).
图 4 实时荧光定量PCR检测方法的特异性注:1:PCV3阳性;对照:PCV1、PCV2、PPV、PRV、v-PRV、PCMV、PBoV、PTTSuV 1a和PTTSuV 1b,样品均为阴性,无荧光扩增信号。Figure 4. Specificity of TaqMan RT-PCR methodNote: 1: PCV3 positive control; experimental control groups: PCV1, PCV2, PPV, PRV, v-PRV, PCMV, PBoV, PTTSuV 1a, and PTTSuV 1b (all negative with no fluorescent signal).2.5 实时荧光定量PCR检测方法的变异系数
本研究建立的基于TaqMan实时荧光定量PCR方法检测PCV3组内变异系数为0.58%~1.10%;建立的方法的组间变异系数0.82%~1.48%(表1)。
表 1 实时荧光定量PCR方法的变异系数Table 1. Coefficients of variation (CV) of TaqMan RT-PCR between inter- and intra-groups序号 Number 拷贝数 Copies 组内 (Intra-group) 组间 (Inter-group) Ct值±标准差 Ct value ± SD 变异系数 CV/% Ct值±标准差 Ct value ± SD 变异系数 CV/% 1 4.22×106 14.82±0.09 0.58 14.83±0.13 0.85 2 4.22×104 22.24±0.16 0.72 22.28±0.18 0.82 3 4.22×102 29.64±0.33 1.10 29.78±0.44 1.48 2.6 临床样品检测结果
对193份样品进行PCV3和PCV2感染的实时荧光定量PCR检测(表2),其中PCV3阳性样品127份,阳性率为65.80%;PCV2阳性样品161份,阳性率为83.42%;PCV3和PCV2混合感染样品102份,阳性率为52.85%。
表 2 临床样品的检测Table 2. Detection on clinical samples by TaqMan RT-PCR method项目 Item 样品数 Number PCV3 PCV2 PCV2&PCV3 阳性样品
Positive samples阳性率
Positive ratio/%阳性样品
Positive samples阳性率
Positive ratio/%阳性样品
Positive samples阳性率
Positive ratio/%2014年 25 7 28.00 21 84.00 5 20.00 2015年 28 13 46.43 20 71.43 11 39.29 2016年 41 28 68.29 34 82.93 18 43.90 2017年 63 51 80.95 55 87.30 43 68.25 2018年 36 28 77.78 31 86.11 25 69.44 合计 Total 193 127 65.80 161 83.42 102 52.85 3. 讨论
截至目前,PCV3还未见成功分离培养的相关研究报道,这为研究PCV3的致病机理带来困扰。前期研究发现,在患有多种临床症状的猪(包括野猪)群多脏器中均可检测到较高的PCV3核酸阳性率[25-27]。但是近期研究也证实在健康猪群中也可检测到PCV3阳性,这导致PCV3对猪群的致病力和机制还存在一定争议[28]。此外,研究还发现PCV3还可在死胎、精液和初乳中均可检测到核酸阳性,上述证据表明PCV3可以垂直传播[29-30],给PCV3的科学防控带来极大挑战。
研究发现,虽然PCV3最早于2015年在美国发现,但是对早期保存的猪组织样品进行检测发现,瑞典猪群中1993年、西班牙和中国猪群中1996年组织样品中均可检测到PCV3阳性,表明PCV3在猪群中存在较长时间[31-32],本研究在保存的2014年组织样品中也检测到PCV3感染阳性,进一步丰富了PCV3在我国的流行病学信息。对于PCV3基因组进行分析发现,PCV3不同时空各毒株相互之间核苷酸同源性较高(98.0%以上),表明PCV3在遗传进化上较为保守。
前期Han等[33]建立的可同时检测PDEV和PCV3的SYBR I实时荧光定量PCR方法,其最低检测限分别为34.6拷贝·μL−1(检测PDEV)和61.2拷贝·μL−1(检测PCV3)。Kim等[34]建立的可同时检测PCV2和PCV3的SYBR I实时荧光定量PCR方法,其最低检测限均为50拷贝·μL−1。本研究利用GenBank数据库PCV3基因特点,选取其复制相关蛋白Rep基因为候选基因靶区来设计特异性的引物和探针,建立检测PCV3感染的TaqMan实时荧光定量PCR方法,该方法敏感性好,最低检测限为42.2拷贝·μL−1,比Chen等[27]建立实时荧光定量PCR方法高,与Han等[33] 和Kim等[34]等建立的方法灵敏度相当;特异性强,对常见猪群传染病均无交叉反应;建立的方法组内变异系数和组间变异系数均在1.48%以内。对福建2014–2018年保存的193份组织样品进行检测发现,PCV3在福建省猪群中存在较高的阳性感染率(65.80%)。进一步对相关样品进行PCV2检测发现,福建省猪群中存在极高的PCV3和PCV2混合感染,这提示我们需进一步开展PCV3和PCV2混合感染的致病机制和协调感染相关研究。
-
图 4 实时荧光定量PCR检测方法的特异性
注:1:PCV3阳性;对照:PCV1、PCV2、PPV、PRV、v-PRV、PCMV、PBoV、PTTSuV 1a和PTTSuV 1b,样品均为阴性,无荧光扩增信号。
Figure 4. Specificity of TaqMan RT-PCR method
Note: 1: PCV3 positive control; experimental control groups: PCV1, PCV2, PPV, PRV, v-PRV, PCMV, PBoV, PTTSuV 1a, and PTTSuV 1b (all negative with no fluorescent signal).
表 1 实时荧光定量PCR方法的变异系数
Table 1 Coefficients of variation (CV) of TaqMan RT-PCR between inter- and intra-groups
序号 Number 拷贝数 Copies 组内 (Intra-group) 组间 (Inter-group) Ct值±标准差 Ct value ± SD 变异系数 CV/% Ct值±标准差 Ct value ± SD 变异系数 CV/% 1 4.22×106 14.82±0.09 0.58 14.83±0.13 0.85 2 4.22×104 22.24±0.16 0.72 22.28±0.18 0.82 3 4.22×102 29.64±0.33 1.10 29.78±0.44 1.48 表 2 临床样品的检测
Table 2 Detection on clinical samples by TaqMan RT-PCR method
项目 Item 样品数 Number PCV3 PCV2 PCV2&PCV3 阳性样品
Positive samples阳性率
Positive ratio/%阳性样品
Positive samples阳性率
Positive ratio/%阳性样品
Positive samples阳性率
Positive ratio/%2014年 25 7 28.00 21 84.00 5 20.00 2015年 28 13 46.43 20 71.43 11 39.29 2016年 41 28 68.29 34 82.93 18 43.90 2017年 63 51 80.95 55 87.30 43 68.25 2018年 36 28 77.78 31 86.11 25 69.44 合计 Total 193 127 65.80 161 83.42 102 52.85 -
[1] KIM J, CHAE C. Differentiation of porcine Circovirus 1 and 2 in formalin-fixed, paraffin-wax-embedded tissues from pigs with postweaning multisystemic wasting syndrome by in situ hybridisation [J]. Research in Veterinary Science, 2001, 70(3): 265−269. DOI: 10.1053/rvsc.2001.0471
[2] HIRAI T, NUNOYA T, IHARA T, et al. Infectivity of porcine Circovirus 1 and Circovirus 2 in primary porcine hepatocyte and kidney cell cultures [J]. The Journal of Veterinary Medical Science, 2006, 68(2): 179−182. DOI: 10.1292/jvms.68.179
[3] PALINSKI R, PIÑEYRO P, SHANG P C, et al. A novel porcine Circovirus distantly related to known circoviruses is associated with porcine dermatitis and nephropathy syndrome and reproductive failure [J]. Journal of Virology, 2017, 91(1): e01879.
[4] PHAN T G, GIANNITTI F, ROSSOW S, et al. Detection of a novel Circovirus PCV3 in pigs with cardiac and multi-systemic inflammation [J]. Virology Journal, 2016, 13(1): 1−8. DOI: 10.1186/s12985-015-0456-4
[5] OUYANG T, NIU G Y, LIU X H, et al. Recent progress on porcine Circovirus type 3 [J]. Infection, Genetics and Evolution, 2019, 73: 227−233. DOI: 10.1016/j.meegid.2019.05.009
[6] FRANZO G, TUCCIARONE C M, DRIGO M, et al. First report of wild boar susceptibility to Porcine Circovirus type 3: High prevalence in the Colli Euganei Regional Park (Italy) in the absence of clinical signs [J]. Transboundary and Emerging Diseases, 2018, 65(4): 957−962. DOI: 10.1111/tbed.12905
[7] KLAUMANN F, DIAS-ALVES A, CABEZÓN O, et al. Porcine Circovirus 3 is highly prevalent in serum and tissues and may persistently infect wild boar (Sus scrofa scrofa) [J]. Transboundary and Emerging Diseases, 2019, 66(1): 91−101. DOI: 10.1111/tbed.12988
[8] SEGALÉS J, ALLAN G M, DOMINGO M. Porcine Circovirus diseases [J]. Animal Health Research Reviews, 2005, 6(2): 119−142. DOI: 10.1079/AHR2005106
[9] GILLESPIE J, OPRIESSNIG T, MENG X J, et al. Porcine Circovirus type 2 and porcine Circovirus-associated disease [J]. Journal of Veterinary Internal Medicine, 2009, 23(6): 1151−1163. DOI: 10.1111/j.1939-1676.2009.0389.x
[10] ZHAI S L, XI Y. Can porcine Circovirus type 3 cause persistent infection in pigs? [J]. The Veterinary Record, 2019, 184(20): 617−618. DOI: 10.1136/vr.l1940
[11] CHEN G H, TANG X Y, SUN Y, et al. Development of a SYBR green-based real-time quantitative PCR assay to detect PCV3 in pigs [J]. Journal of Virological Methods, 2018, 251: 129−132. DOI: 10.1016/j.jviromet.2017.10.012
[12] WANG J, ZHANG Y, ZHANG R, et al. Development of a SYBR green-based real-time quantitative PCR assay to detect PCV3 in pigs [J]. Molecular and Cellular Probes, 2017, 36: 58−61. DOI: 10.1016/j.mcp.2017.09.001
[13] ZHENG S, WU X, SHI J, et al. Rapid specific and visible detection of porcine Circovirus type 3 using loop-mediated isothermal amplification (LAMP) [J]. Transboundary and Emerging Diseases, 2018, 65(3): 597−601. DOI: 10.1111/tbed.12835
[14] WANG H N, LIU X N, ZENG F W, et al. Development of a real-time loop-mediated isothermal amplification assay for detection of porcine Circovirus 3 [J]. BMC Veterinary Research, 2019, 15(1): 1−5. DOI: 10.1186/s12917-018-1758-8
[15] DENG J H, LI X D, ZHENG D D, et al. Establishment and application of an indirect ELISA for porcine Circovirus 3 [J]. Archives of Virology, 2018, 163(2): 479−482. DOI: 10.1007/s00705-017-3607-7
[16] ZHANG Y N, ZHANG Z, WANG Z Y, et al. Development of a droplet digital PCR assay for sensitive detection of porcine Circovirus 3 [J]. Molecular and Cellular Probes, 2019, 43: 50−57. DOI: 10.1016/j.mcp.2018.11.005
[17] 代振江, 王伟丞, 曾智勇, 等. 猪圆环病毒基因组结构与功能研究进展 [J]. 畜牧与兽医, 2016, 48(3):151−154. DAI Z J, WANG W C, ZENG Z Y, et al. Research progress on genomic structure and function of porcine circovirus type 2 [J]. Animal Husbandry & Veterinary Medicine, 2016, 48(3): 151−154.(in Chinese)
[18] KLAUMANN F, CORREA-FIZ F, FRANZO G, et al. Current knowledge on Porcine Circovirus 3 (PCV-3): a novel virus with a yet unknown impact on the swine industry [J]. Frontiers in Veterinary Science, 2018, 5: 315. DOI: 10.3389/fvets.2018.00315
[19] 陈如敬, 周伦江, 吴学敏, 等. 猪伪狂犬病毒gB基因序列分析 [J]. 福建农业学报, 2016, 31(11):1139−1144. CHEN R J, ZHOU L J, WU X M, et al. Sequence analysis on gB gene from pseudorabies virus [J]. Fujian Journal of Agricultural Sciences, 2016, 31(11): 1139−1144.(in Chinese)
[20] 陈如敬, 陈铖, 吴学敏, 等. 猪巨细胞病毒福建株DPOL基因的克隆及序列分析 [J]. 中国畜牧兽医, 2017, 44(2):365−370. CHEN R J, CHEN C, WU X M, et al. Cloning and sequencing analysis of DPOL gene of porcine Cytomegalovirus [J]. China Animal Husbandry & Veterinary Medicine, 2017, 44(2): 365−370.(in Chinese)
[21] 陈如敬, 吴学敏, 车勇良, 等. 猪博卡病毒非结构蛋白NP1基因的克隆及基因分型研究 [J]. 中国畜牧兽医, 2015, 42(2):292−297. CHEN R J, WU X M, CHE Y L, et al. Cloning and genotyping classification of nonstructural protein NP1 gene of porcine bocavirus [J]. China Animal Husbandry & Veterinary Medicine, 2015, 42(2): 292−297.(in Chinese)
[22] 胡崇伟, 陈如敬, 陈秋勇, 等. 猪细环病毒1b型ORF3基因克隆及序列分析 [J]. 中国畜牧兽医, 2014, 41(8):76−80. HU C W, CHEN R J, CHEN Q Y, et al. Cloning and sequence analysis of ORF3 gene of porcine torque teno sus virus 1b [J]. China Animal Husbandry & Veterinary Medicine, 2014, 41(8): 76−80.(in Chinese)
[23] 陈如敬, 黄秋宇, 修金生, 等. 猪细环病毒k2型SYBR GreenⅠ实时荧光定量PCR方法的建立 [J]. 中国动物传染病学报, 2016, 24(5):10−15. DOI: 10.3969/j.issn.1674-6422.2016.05.003 CHEN R J, HUANG Q Y, XIU J S, et al. Development of a SYBR greenⅠquantitative real-time PCR method for porcine Torque teno sus virus k2 [J]. Chinese Journal of Veterinary Parasitology, 2016, 24(5): 10−15.(in Chinese) DOI: 10.3969/j.issn.1674-6422.2016.05.003
[24] ZHAO K, HAN F T, ZOU Y, et al. Rapid detection of porcine Circovirus type 2 using a TaqMan-based real-time PCR [J]. Virology Journal, 2010, 7(1): 1−5. DOI: 10.1186/1743-422X-7-1
[25] 张永宁, 梅琳, 张舟, 等. 猪圆环病毒3型研究进展 [J]. 东北农业大学学报, 2017, 48(9):89−96. DOI: 10.3969/j.issn.1005-9369.2017.09.011 ZHANG Y N, MEI L, ZHANG Z, et al. Research progress on porcine Circovirus 3 [J]. Journal of Northeast Agricultural University, 2017, 48(9): 89−96.(in Chinese) DOI: 10.3969/j.issn.1005-9369.2017.09.011
[26] 刘建奎, 魏春华, 陈小燕, 等. 福建省新发猪圆环病毒3型流行病学调查及遗传变异分析 [J]. 中国预防兽医学报, 2018, 40(2):160−163. LIU J K, WEI C H, CHEN X Y, et al. Epidemiological investigation and genetic diversity analysis of novel porcine Circovirus type 3 in Fujian Province [J]. Chinese Journal of Preventive Veterinary Medicine, 2018, 40(2): 160−163.(in Chinese)
[27] MALMSTEN A, MAGNUSSON U, RUIZ-FONS F, et al. A serologic survey of pathogens in wild boar (sus scrofa) in Sweden [J]. Journal of Wildlife Diseases, 2018, 54(2): 229−237. DOI: 10.7589/2017-05-120
[28] ZHENG S, WU X, ZHANG L, et al. The occurrence of porcine Circovirus 3 without clinical infection signs in Shandong Province [J]. Transboundary and Emerging Diseases, 2017, 64(5): 1337−1341. DOI: 10.1111/tbed.12667
[29] FRANZO G, GRASSI L, TUCCIARONE C M, et al. A wild circulation: High presence of Porcine Circovirus 3 in different mammalian wild hosts and ticks [J]. Transboundary and Emerging Diseases, 2019, 66(4): 1548−1557.
[30] FACCINI S, BARBIERI I, GILIOLI A, et al. Detection and genetic characterization of Porcine Circovirus type 3 in Italy [J]. Transboundary and Emerging Diseases, 2017, 64(6): 1661−1664. DOI: 10.1111/tbed.12714
[31] SUN J, WEI L, LU Z, et al. Retrospective study of porcine Circovirus 3 infection in China [J]. Transboundary and Emerging Diseases, 2018, 65(3): 607−613. DOI: 10.1111/tbed.12853
[32] YE X Y, BERG M, FOSSUM C, et al. Detection and genetic characterisation of porcine Circovirus 3 from pigs in Sweden [J]. Virus Genes, 2018, 54(3): 466−469. DOI: 10.1007/s11262-018-1553-4
[33] HAN H Y, ZHENG H H, ZHAO Y, et al. Development of a SYBR green I-based duplex real-time fluorescence quantitative PCR assay for the simultaneous detection of porcine epidemic diarrhea virus and porcine Circovirus 3 [J]. Molecular and Cellular Probes, 2019, 44: 44−50. DOI: 10.1016/j.mcp.2019.02.002
[34] KIM H R, PARK Y R, LIM D R, et al. Multiplex real-time polymerase chain reaction for the differential detection of porcine Circovirus 2 and 3 [J]. Journal of Virological Methods, 2017, 250: 11−16. DOI: 10.1016/j.jviromet.2017.09.021
-
期刊类型引用(3)
1. 李虎,孙鹏亮,邢潇月,张家浩,刘祥杰,李莲瑞. 新疆阿克苏地区猪圆环病毒3型TaqMan荧光定量PCR检测方法的建立. 塔里木大学学报. 2023(01): 45-50 .  百度学术
百度学术
2. 殷冬冬,王洁茹,尹磊,沈学怀,戴银,赵瑞宏,侯宏艳,胡晓苗,王桂军,潘孝成. 猪圆环病毒3型和4型TaqMan双重实时荧光定量PCR检测方法的建立. 安徽农业大学学报. 2023(06): 979-984 .  百度学术
百度学术
3. 尹佳彤,马湘海,袁晨阳,成豫. 猪圆环病毒3型检测方法研究进展. 畜禽业. 2021(04): 14+16 .  百度学术
百度学术
其他类型引用(6)





 下载:
下载: