Diversity and Network Structure Analysis of Culturable Bacteria in Sporophore Site Soil of Russula griseocarnosa
-
摘要:目的 通过对正红菇菇位土壤细菌的分离培养,探究其可培养细菌的多样性及其种间互作关系,为后续筛选促进正红菇菌丝体生长和子实体形成分化的菌根促生菌奠定基础。方法 利用LB和ISP2两种培养基通过传统分离培养的方法对正红菇菇位土壤中的细菌进行分离纯化,并利用16S rRNA基因序列分析初步确定分离菌株的分类地位,使用R语言中的Bipartite包分析可培养细菌属间的相互作用。结果 6份正红菇菇位土壤中共分离出128株细菌,隶属于3门16属34个OTUs,丰度最高的3个属分别是芽孢杆菌属、伯克霍尔德氏菌属和链霉菌属。属水平-采样品网络分析表明,可培养细菌分布具有随机的嵌套性。Bacillus属和Burkholderia属在细菌群落中存在较多的有效合作值(Effective partners)和亲密度(Closeness),被其他细菌所依赖程度(Species Strength)较高,是该群落中的重要组成类群。结论 正红菇菇位土壤中具有丰富的细菌资源,细菌的群落组成和网络结构相结合分析可以更清晰阐明细菌间的相互作用关系。Abstract:Objective The diversity of culturable bacteria and their inter-species interactions were explored by isolating and culturing sporophore site soil bacteria of R. griseocarnosa. It provides insights into the mycorrhizal helper bacteria which may promote the growth of the mycelium and the formation and differentiation of sporophore of R. griseocarnosa.Methods The bacteria in the sporophore site soil of R. griseocarnosa were isolated and purified by traditional methods of separation and culture using LB and ISP2 media, and the species of the isolated strains was preliminarily identified by 16S rRNA gene sequence analysis. Interactions between culturable bacterial genera were analyzed using the Bipartite package in R.Results A total of 128 strains of bacteria were isolated from 6 sporophore site soils of R. griseocarnosa, which belonged to 34 OTUs (16 genera of 3 phyla). The three genera with the highest abundance were Bacillus , Burkholderia and Streptomyces. Genus-level-Sample network analysis showed that the distribution of culturable bacteria was randomly nested. Bacillus and Burkholderia are important groups in the community, which have more Effective Partners, Closeness and high Species Strength in the bacterial community.Conclusion There are abundant bacterial resources in the sporophore site soil of R. griseocarnosa. Combining the analysis of bacterial abundance and network structure can predict the interaction between bacteria more clearly.
-
Keywords:
- Russula griseocarnosa /
- rhizosphere soil /
- culturable bacteria /
- network structure
-
0. 引言
【研究意义】正红菇(Russula griseocarnosa) 隶属于担子菌门(Basidiomycota),伞菌纲(Agaricomycetes),红菇目(Russulales),红菇科(Russulaceae),红菇属(Russula Pers.),又名灰肉红菇、大红菌、红锥菌、真红菇,是一种野生的可食用的珍贵外生菌根真菌,一年出菇2茬,一般发生在每年农历4月中旬至5月中旬和农历8月中旬至9月中旬,出菇地点相对固定,俗称“菇位”[1]。由于正红菇对其生长环境条件要求极为苛刻,目前尚无法进行人工培育[2]。土壤中的微生物可以调控外生菌根及其子实体的形成[3-6],能促进外生菌根形成的微生物被称为菌根促生菌 (Mycorrhization helper bacteria,MHB) [7],这些微生物对菌根的形成具有重要的影响,尤其是在促进菌根真菌菌丝生长和提高菌根侵染率等方面[8-10]。正红菇菌根根际是正红菇菌根形成以及正红菇子实体分化和生长的重要场所。研究正红菇菌根根际土壤细菌多样性对筛选正红菇菌丝体生长和子实体形成分化的菌根促生菌具有重要意义。【前人研究进展】近年来,学者对正红菇的研究主要集中在林分组成和土壤理化性质等对红菇生长的影响等方面[11-13],正红菇菌根根际土壤微生物的研究主要是利用变性梯度凝胶电泳分析[14, 15]和通过高通量测序[16]的方法来开展的。有研究表明,通过高通量测序显示出高丰度的细菌与可培养的方法获得的细菌存在显著的差异,并且可培养的方法可以获得一些高通量测序显示的较低丰度的细菌[17]。【本研究切入点】对正红菇菌根根际微生物分离培养是筛选正红菇菌根辅助细菌的关键一步,目前采用分离培养的方法对正红菇菌根根际土壤细菌的研究鲜有报道。【拟解决的关键问题】以正红菇菇位土壤为研究对象,通过纯培养与16S rRNA基因序列相结合的方法对正红菇菇位土壤内可培养细菌进行分离培养鉴定,分析正红菇菇位细菌群落组成与多样性,及该群落中物种间的网络相互作用,为筛选促进正红菇菌丝体生长和子实体形成分化的菌根促生菌奠定基础。
1. 材料与方法
1.1 土壤样品采集
于2020年9月在福建省三明市砂蕉村台江国有林场正红菇试验地采集土壤样品,在野外发现正红菇子实体后,采集其子实体并在原位插上标牌以定位,在定位的正红菇位点采集正红菇菇位土壤6份。先用灭菌的镊子拨开表面的腐殖质,用直径8 mm的打孔器收集正红菇子实体正下方0~5 cm土壤,每份样品采集5个样品混合作为一份样品,放进无菌的50 mL的离心管内,迅速放入保温箱内,低温运回实验室,并储存于4 ℃冰箱中。
1.2 土壤微生物的分离纯化
本试验采用LB培养基和ISP2培养基进行分离培养[18]。分离培养步骤:称取土壤样品10 g,置于盛有90 mL无菌水的三角瓶中,200 r·min−1振荡20 min。取土壤悬浮液1 mL置于9 mL PBS缓冲液中,逐级稀释至10−6倍,分别取10−3、10−4和10−5含量的土壤悬浮液均匀涂布于LB培养基和ISP2培养基,每个梯度重复3次,涂板均匀后将培养平板倒置于37 ℃恒温下培养,期间观察记录可培养细菌的菌落特征并计数,同时从平板上挑取不同培养性状的单菌落,继续在对应平板上划线纯化,直至获得纯培养细菌菌株。用灭菌的牙签轻轻挑取单菌落,接种在装有对应液体培养基的离心管中,置于摇床内30 ℃,200 r·min−1继续培养,最后用30%的甘油将细菌保存于−80 ℃冰箱中。
1.3 可培养微生物的分子鉴定
细菌的DNA提取采用菌落PCR的方法。具体方法如下:(1)用经灭菌的牙签挑取纯菌落于PCR管中,加入20 μL ddH2O,置于PCR仪中,98 ℃加热10 min。经过离心的上清液即为细菌DNA。(2)将所提取的DNA进行16S rDNA的PCR扩增。PCR扩增所用引物[19-20]:16S 27F:5′-AGAGTTTGATCCTGGCTCAG-3′和16S 1492R:5′-TACGGYTACCTTGTTACGACTT-3′。PCR扩增所用反应体系:DNA模板1 μL,上下游引物(10 μmol·L−1)各1 μL,PCR Mix 12 μL,用ddH2O补足至25 μL反应体系。PCR反应程序:95 ℃ 3 min;95 ℃ 15 s,55 ℃ 20 s,72 ℃ 1 min,35个循环;72 ℃ 10 min;保温:4 ℃。将经过琼脂糖凝胶电泳检测在1 500 bp附近有明显条带的PCR扩增产物送广州擎科生物技术有限公司进行序列测定。
1.4 数据分析
将测序结果与NCBI网站GenBank数据库中的序列进行Blast比对,选取相似度大于97%且相似度最高菌株的16S rRNA序列,将比对到同一序列的细菌菌株划分到一个操作分类单元(Operational taxonomic units,OTUs)。运用MEGA 5软件进行系统发育分析,根据序列的同源程度初步确定待鉴定的菌株在分类学上的地位。系统发育树构建方法采用Neighbor-joining,自展数(Bootstrap)为1 000,选定2条Aquifex pyrophilus序列[21]为外群。
根据鉴定结果,计算正红菇菇位土壤微生物的相对分离率[22](指分离到的某种细菌占分离到细菌总株数的百分率,用来衡量某种细菌的优势度)与正红菇菇位土壤细菌群落结构多样性[23]。多样性指数计算公式:
香农指数(Shannon-Weiner index):
H′= −∑PilnPi 优势度指数(Simpson index):
DJ=1−∑P2i 丰富度指数(Margalef index):
Ma=(S−1)/lnN 均匀度指数(Pielou index):
J′=−∑PilnPi/lnS 式中,S表示正红菇菇位土壤细菌的种类数,N表示正红菇菇位土壤细菌的总株数,Pi表示某种细菌的相对分离率。
细菌群落间的网络结构可视化与网络的嵌套结构(Nestedness metric based on overlap and decreasing fill,NODF)、物种依赖度(Species strength)、有效合作值(Effective partners)和亲密度(Closeness)等网络结构参数均使用R语言(4.1.2)的Bipartite包[24]进行分析和计算,并通过与零模型相比较以确定正红菇菇位细菌群落的组装是否为一个随机过程[25]。
2. 结果与分析
2.1 正红菇菇位土壤可培养细菌的组成
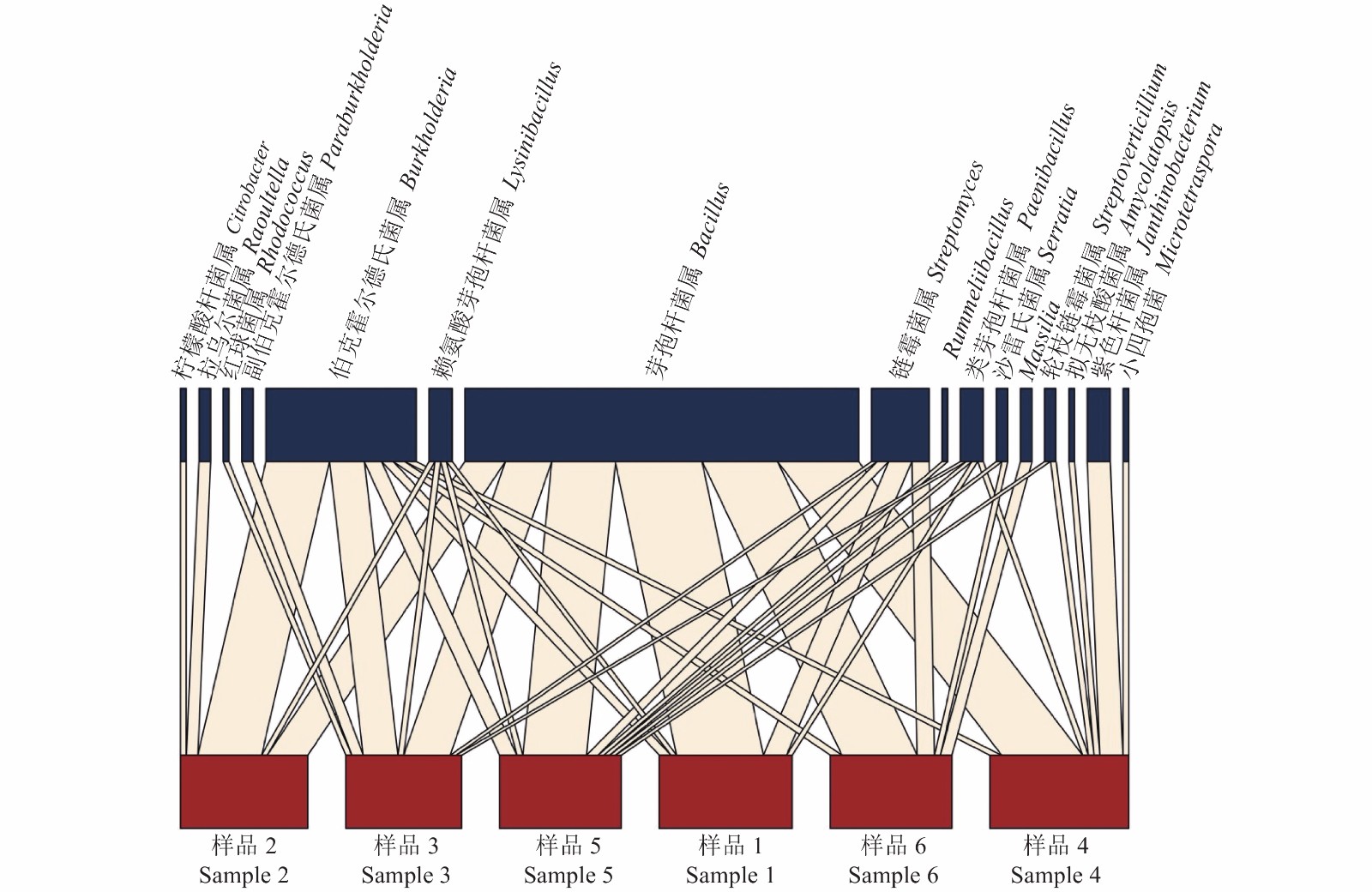
采用稀释平板分离法,利用LB和ISP2两种培养基从6份正红菇菇位土壤样品中一共分离到128株细菌。对分离到的细菌提取总DNA,使用引物27F和1492R进行PCR扩增后送公司测序,将测序结果在NCBI网站上进行BLAST比对,共鉴定到34个OTUs,隶属于3门16属,其具体信息如表1所示。以34个OUTs的16S rRNA序列及其参考序列构建的系统进化树如图1所示。正红菇菇位土壤可培养细菌主要分为3个大类:第一大类为厚壁菌门(Firmicutes),包括芽孢杆菌属(Bacillus)、赖氨酸芽孢杆菌属(Lysinibacillus)、类芽孢杆菌属(Paeinibacillus)等;第二大类为变形菌门(Proteobacteria),包括伯克霍尔德氏菌属(Burkholderia)、副伯克霍尔德氏菌属(Paraburkholderia)、沙雷氏菌属(Serratia)等;第三大类为放线菌门(Actinobacteria),主要是链霉菌属(Streptomyces.)的细菌。
表 1 正红菇菇位土壤中分离出的34个OUTs细菌的16S rRNA基因序列比对结果Table 1. 16S rRNA gene sequences of 34 OUTs microbes isolated from R. griseocarnosa rhizosphere soil代表菌株
Strain最大相似菌株及GenBank登录号
Similarity strain and GenBank accession No.相似度
Similarity/%菌株数
Number of strainsF422 卤代拟青霉 Amycolatopsis halotolerans(NR_043452) 99 1 L145 蜡样芽孢杆菌 Bacillus cereus(MN999986) 99 15 F124 堀越氏芽孢杆菌 Bacillus horikoshii(MT883498) 99 21 F202 巨大芽孢杆菌 Bacillus megaterium(MW391757) 99 1 L501 蕈状芽孢杆菌 Bacillus mycoides (KU877669) 99 4 L311 稻壳芽孢杆菌 Bacillus oryzaecorticis (MN330145) 99 7 L421 东京芽孢杆菌 Bacillus toyonensis(MN543844) 99 1 L215 热带芽孢杆菌 Bacillus tropicus (MW478751) 99 1 L405 苏云金芽孢杆菌 Bacillus thuringiensis (MN330087) 99 9 L127 越南芽孢杆菌 Bacillus wiedmannii (MH041257) 99 3 L617 芽孢杆菌属 Bacillus sp. (MG309547) 99 6 F301 洋葱伯克霍尔德氏菌 Burkholderia cepacia (LC462133) 99 19 F308 污染伯克霍尔德氏菌 Burkholderia contaminans (MN826151) 100 6 F410 伯克霍尔德氏菌属 Burkholderia sp. (JQ864385) 99 1 L213 柠檬酸杆菌属 Citrobacter sp. (AB673462) 99 1 F414 紫色杆菌属 Janthinobacterium sp. (EU098005) 99 4 L228 细长赖氨酸芽孢杆菌 Lysinibacillus macroides (MN263206) 99 4 F418 Massilia sp. (AB545620) 99 2 F421 马来小四孢菌 Microtetraspora malaysiensis (NR_024780) 99 1 F431 千叶类芽孢杆菌 Paenibacillus chibensis (MN826593) 99 1 F111 Paenibacillus chinjuensis (KP980606) 99 1 F508 松树土壤类芽孢杆菌 Paenibacillus pinihumi (NR_117367) 99 1 F326 类芽孢杆菌属 Paenibacillus sp. (MK215829) 98 1 F321 副伯克霍尔德氏菌属 Paraburkholderia sp. (OK445519) 99 2 L222 植生拉乌尔菌 Raoultella planticola (NR_119279) 99 2 F331 Rhodococcus soli (NR_134799) 99 1 F512 Rummeliibacillus sp. (MN589588) 99 1 F602 黏质沙雷氏菌 Serratia marcescens (GU220796) 99 2 F114 丙氨菌素链霉菌 Streptomyces alanosinicus (KJ571026) 99 2 F129 孔雀石褐链霉菌 Streptomyces malachitofuscus (MN428156) 99 4 F501 生米卡链霉菌 Streptomyces mycarofaciens (EF063483) 99 1 F604 结节链霉菌 Streptomyces nodosus (MN421090) 99 1 F119 链霉菌属 Streptomyces sp. (MF455322) 99 2 F613 Streptoverticillium reticulum (MF062672) 99 2 注:F和L开头指分别用ISP2培养基和LB培养基分离的菌株,字母后第一个数字为样品编号,后两位数字为菌株编号。
Note: F and L indicate the strains isolated from ISP2 media and LB media, respectively. The first number after the letter is the sample number, and the last two numbers are the strain number.2.2 正红菇菇位土壤可培养细菌多样性分析
6个正红菇菇位土壤样品中的可培养细菌丰度组成如图2所示,样品1~6中具有的优势类群不同,分别是厚壁菌门的芽孢杆菌属(Bacillus)、放线菌门的链霉菌属(Streptomyces.)和变形菌门的伯克霍尔德氏菌属(Burkholderia)的细菌(样品1);变形菌门的伯克霍尔德氏菌属(Burkholderia)、厚壁菌门的芽孢杆菌属(Bacillus)和放线菌门的链霉菌属(Streptomyces.) (样品2);变形菌门的伯克霍尔德氏菌属(Burkholderia)、厚壁菌门的芽孢杆菌属(Bacillus)和变形菌门的副伯克霍尔德氏菌属(Paraburkholderia)的细菌(样品3);厚壁菌门的芽孢杆菌属(Bacillus)、变形菌门的蓝黑紫色杆菌属(Janthinobacterium)和变形菌门的伯克霍尔德氏菌属(Burkholderia)的细菌(样品4);厚壁菌门的芽孢杆菌属(Bacillus)、变形菌门的伯克霍尔德氏菌属(Burkholderia)和放线菌门的链霉菌属(Streptomyces.)的细菌(样品5);厚壁菌门的芽孢杆菌属(Bacillus)、放线菌门的链霉菌属(Streptomyces.)和变形菌门的伯克霍尔德氏菌属(Burkholderia)的细菌(样品6)。通过对6份正红菇菇位土壤样品获得的128株细菌隶属的结果进行分析得知,丰度前3的属分别是厚壁菌门的芽孢杆菌属(Bacillus)(占53.13%)、变形菌门的伯克霍尔德氏菌属(Burkholderia)(占20.31%)、放线菌门的链霉菌属(Streptomyces.)(占9.38%)。对6个正红菇菇位土壤可培养细菌多样性结果如表2所示,各样品的香农指数在2.94~3.72,辛普森指数在0.81~0.91,丰富度指数在3.19~4.41,均匀度指数在1.22~1.38。
表 2 6个正红菇菇位土壤样品可培养细菌多样性指数Table 2. Diversity index of culturable microbes in 6 rhizosphere soil samples from sites of R. griseocarnosa样品
Samples香农指数
Shannon-Weiner index优势度指数
Simpson index丰富度指数
Margalef index均匀度指数
Pielou index样品1 Sample 1 2.94 0.81 3.19 1.22 样品2 Sample 2 2.97 0.82 3.24 1.24 样品3 Sample 3 3.55 0.91 4.01 1.38 样品4 Sample 4 3.72 0.91 4.41 1.37 样品5 Sample 5 3.23 0.87 3.61 1.30 样品6 Sample 6 3.40 0.89 3.61 1.37 2.3 正红菇菇位土壤可培养细菌群落的网络结构分析
正红菇菇位土壤可培养细菌属水平与采样品的网络结构分析结果表明(图3),分离细菌中丰度最大的是芽孢杆菌属(Bacillus)和伯克霍尔德氏菌属(Burkholderia),可在所有采样品分离到;部分细菌属仅在特定的采样品中分离到,如柠檬酸杆菌属(Citrobacter)和拉乌尔菌属(Raoultella)仅在样品2中分离到;红球菌属(Rhodococcus)和副伯克霍尔德氏菌属(Paraburkholderia)仅在样品3中分离到;拟无枝酸菌属(Amycolatopsis)、蓝黑紫色杆菌属(Janthinobacterium)和小四孢菌(Microtetraspora)属仅在样品4中分离到;Rummeliibacillus属仅在样品5中分离到;轮枝链霉菌属(Streptoverticillium)仅在样品6中分离到。
分析正红菇菇位土壤可培养细菌属间的相互作用结果表明(表3),芽孢杆菌属(Bacillus)的物种依赖程度最高为3.10,其次是伯克霍尔德氏菌属(Burkholderia)为1.21;芽孢杆菌属(Bacillus)、伯克霍尔德氏菌属(Burkholderia)、赖氨酸芽孢杆菌属(Lysinibacillus)和类芽孢杆菌属(Paenibacillus)的有效合作值较其他细菌属更高,分别为5.79、4.68、4.00和4.00;芽孢杆菌属(Bacillus)和伯克霍尔德氏菌属(Burkholderia)的亲密度较高,为0.08;对网络中各细菌的依赖度、有效合作值和亲密度分析表明,芽孢杆菌属(Bacillus)和伯克霍尔德氏菌属(Burkholderia)在细菌群落中被其他属的细菌所依赖程度较高,并可能拥有更多的相互作用。与零模型相比,正红菇菇位土壤可培养细菌(属水平)与采样品的网络嵌套性(NODF=44.66)差异不显著(P=0.29),即样品中分离菌株在属水平的分布具有随机嵌套性。
表 3 正红菇菇位土壤中可培养细菌属间的网络相互作用Table 3. Network interactions among culturable microbial genera in R. griseocarnosa rhizosphere soil属名
Genus物种依赖
程度
Species
strength有效
合作值
Effective
partners亲密度
Closeness拟无枝酸菌属 Amycolatopsis 0.04 1.00 0.06 芽孢杆菌属 Bacillus 3.10 5.79 0.08 伯克霍尔德菌氏属 Burkholderia 1.21 4.68 0.08 柠檬酸杆菌属 Citrobacter 0.05 1.00 0.05 紫色杆菌属 Janthinobacterium 0.17 1.00 0.06 赖氨酸芽孢杆菌属 Lysinibacillus 0.19 4.00 0.07 Massilia 0.09 2.00 0.07 小四孢菌属 Microtetraspora 0.04 1.00 0.06 类芽孢杆菌属 Paenibacillus 0.19 4.00 0.07 副伯克霍尔德氏菌属 Paraburkholderia 0.10 1.00 0.06 拉乌尔菌属 Raoultella 0.09 1.00 0.05 红球菌属 Rhodococcus 0.05 1.00 0.06 Rummeliibacillus 0.05 1.00 0.06 沙雷氏菌属 Serratia 0.10 2.00 0.06 链霉菌属 Streptomyces 0.46 3.60 0.07 轮枝链霉菌属 Streptoverticillium 0.10 1.00 0.05 3. 讨论
本研究通过传统分离培养的方法从正红菇菇位土壤中分离到可培养细菌128株,其分别隶属于3个门16个属,可以划分为34个OTUs。丰度排前三的属中,芽孢杆菌属(Bacillus)和伯克霍尔德氏菌属(Burkholderia)在6个样品中均可以分离到,链霉菌属(Streptomyces.)仅可以在样品1、3、5、6中分离到。近年来,大量研究通过变性梯度凝胶电泳和高通量测序分析了正红菇菌根际微生物多样性。肖冬来等[14]发现正红菇菌根根际土壤细菌主要分为7个类群:α-变形菌门(Alphaproteobacteria)、β-变形菌门(Betaproteobacteria)、γ-变形菌门(Gammaproteobacteria)、酸杆菌门(Acidobateria)、浮霉菌门(Planctomycetes)、疣微菌门(Verrucomicrobia)及分类地位未知的不可培养细菌;Yu等[16]利用16S扩增子测序对正红菇菌根根际细菌进行了研究,结果表明正红菇菌根根际细菌的多样性比对照组更低,伯克霍尔德菌属-副伯克霍尔德菌属(Burkholderia-Paraburkholderia)、分枝杆菌属(Mycobacterium)、堆囊菌属(Sorangium)、酸杆菌属(Acidobacterium)、Roseiarcus属和Singulisphaera属的细菌可能对正红菇的生长具有一定的促进作用。然而以上研究无法获得正红菇菌根根际土壤微生物的纯培养,难以进一步开展土壤微生物与正红菇关系的相关研究。我们通过传统分离培养的方法对正红菇菇位可培养细菌进行了研究,表明其优势菌属是芽孢杆菌属(Bacillus)和伯克霍尔德氏菌属(Burkholderia)。
菌根对其生长环境中的微生物存在选择和调控的作用,同时环境微生物也影响着菌根的形成和生长以及基因的表达[26-28]。姜华等在6个松口蘑菌塘土壤中分离到的最多也是芽孢杆菌属(Bacillus)和伯克霍尔德氏菌属(Burkholderia)的细菌[21];安忠琦等在金钗石斛根际可培养细菌多样性的研究中分离到的最多也是芽孢杆菌属(Bacillus)的细菌[29];而万山平等在攀枝花块菌-华山松菌根根际土壤可培养细菌的多样性研究中分离到的最多是伯克霍尔德氏菌属(Burkholderia)的细菌[30]。同时,本研究从正红菇菇位土壤中也分离到了大量芽孢杆菌属(Bacillus)和伯克霍尔德氏菌属(Burkholderia)的细菌。有研究表明,从Tuber borchii中分离到的芽孢杆菌属(Bacillus)的细菌可以降解几丁质[31],以此来促进Tuber borchii产生孢子和形成子实体;从Laccaria laccata菌根和根际中分离到的芽孢杆菌可以促进外生菌根真菌菌丝的生长以及外生菌根的形成[32, 33];类芽孢杆菌(Paenibacillus)可以利用可溶性代谢物促进松茸生长[34]。盛江梅等[35]分到一株蜡样芽孢杆菌(Bacillus cereus)可以促进美味牛肝菌(Boletus edulis)菌丝体的生长并且促进其与宿主黑松形成菌根从而促进宿主植物生长。然而,芽孢杆菌属(Bacillus)和伯克霍尔德氏菌属(Burkholderia)的细菌与正红菇菌丝的生长和菌根形成的互作关系及影响机理还未见报道,有待开展进一步的研究。
Rivett等[36]学者认为,细菌的相对丰度在一定程度上反映了其在群落中的重要程度,相对丰度较高的细菌在群落中的相互作用可能更多。本研究中的芽孢杆菌属(Bacillus)和伯克霍尔德氏菌属(Burkholderia)具有相对较高的丰度同时也具有较高的相互作用,是正红菇菇位土壤中细菌群落的关键类群。但是,Franck等[17]的研究中通过高通量测序方法显示出具有较高丰度假单胞菌属的细菌种,在其传统分离培养和高通量测序的网络互作分析中都显示出较低的相互关系。因此,判断细菌在群落中的重要程度不仅要看其相对丰度,其在网络互作中的相互关系一样需要受到关注。除此之外,通过高通量测序和传统分离培养2种方法检测到的细菌在群落组成和相对丰度上存在明显的差异,在正红菇菇位土壤可培养细菌丰度占前5的细菌属中仅伯克霍尔德氏菌属(Burkholderia)是高通量测序分析结果中相对丰度前8的主要类群之一,且正红菇菇位土壤可培养细菌丰度最高的芽孢杆菌属(Bacillus)在高通量测序研究中并没有显示出较高的丰度[37]。因此将传统纯培养方法和高通量测序技术相结合才能更好地研究正红菇菌根根际中的细菌多样性。
-
表 1 正红菇菇位土壤中分离出的34个OUTs细菌的16S rRNA基因序列比对结果
Table 1 16S rRNA gene sequences of 34 OUTs microbes isolated from R. griseocarnosa rhizosphere soil
代表菌株
Strain最大相似菌株及GenBank登录号
Similarity strain and GenBank accession No.相似度
Similarity/%菌株数
Number of strainsF422 卤代拟青霉 Amycolatopsis halotolerans(NR_043452) 99 1 L145 蜡样芽孢杆菌 Bacillus cereus(MN999986) 99 15 F124 堀越氏芽孢杆菌 Bacillus horikoshii(MT883498) 99 21 F202 巨大芽孢杆菌 Bacillus megaterium(MW391757) 99 1 L501 蕈状芽孢杆菌 Bacillus mycoides (KU877669) 99 4 L311 稻壳芽孢杆菌 Bacillus oryzaecorticis (MN330145) 99 7 L421 东京芽孢杆菌 Bacillus toyonensis(MN543844) 99 1 L215 热带芽孢杆菌 Bacillus tropicus (MW478751) 99 1 L405 苏云金芽孢杆菌 Bacillus thuringiensis (MN330087) 99 9 L127 越南芽孢杆菌 Bacillus wiedmannii (MH041257) 99 3 L617 芽孢杆菌属 Bacillus sp. (MG309547) 99 6 F301 洋葱伯克霍尔德氏菌 Burkholderia cepacia (LC462133) 99 19 F308 污染伯克霍尔德氏菌 Burkholderia contaminans (MN826151) 100 6 F410 伯克霍尔德氏菌属 Burkholderia sp. (JQ864385) 99 1 L213 柠檬酸杆菌属 Citrobacter sp. (AB673462) 99 1 F414 紫色杆菌属 Janthinobacterium sp. (EU098005) 99 4 L228 细长赖氨酸芽孢杆菌 Lysinibacillus macroides (MN263206) 99 4 F418 Massilia sp. (AB545620) 99 2 F421 马来小四孢菌 Microtetraspora malaysiensis (NR_024780) 99 1 F431 千叶类芽孢杆菌 Paenibacillus chibensis (MN826593) 99 1 F111 Paenibacillus chinjuensis (KP980606) 99 1 F508 松树土壤类芽孢杆菌 Paenibacillus pinihumi (NR_117367) 99 1 F326 类芽孢杆菌属 Paenibacillus sp. (MK215829) 98 1 F321 副伯克霍尔德氏菌属 Paraburkholderia sp. (OK445519) 99 2 L222 植生拉乌尔菌 Raoultella planticola (NR_119279) 99 2 F331 Rhodococcus soli (NR_134799) 99 1 F512 Rummeliibacillus sp. (MN589588) 99 1 F602 黏质沙雷氏菌 Serratia marcescens (GU220796) 99 2 F114 丙氨菌素链霉菌 Streptomyces alanosinicus (KJ571026) 99 2 F129 孔雀石褐链霉菌 Streptomyces malachitofuscus (MN428156) 99 4 F501 生米卡链霉菌 Streptomyces mycarofaciens (EF063483) 99 1 F604 结节链霉菌 Streptomyces nodosus (MN421090) 99 1 F119 链霉菌属 Streptomyces sp. (MF455322) 99 2 F613 Streptoverticillium reticulum (MF062672) 99 2 注:F和L开头指分别用ISP2培养基和LB培养基分离的菌株,字母后第一个数字为样品编号,后两位数字为菌株编号。
Note: F and L indicate the strains isolated from ISP2 media and LB media, respectively. The first number after the letter is the sample number, and the last two numbers are the strain number.表 2 6个正红菇菇位土壤样品可培养细菌多样性指数
Table 2 Diversity index of culturable microbes in 6 rhizosphere soil samples from sites of R. griseocarnosa
样品
Samples香农指数
Shannon-Weiner index优势度指数
Simpson index丰富度指数
Margalef index均匀度指数
Pielou index样品1 Sample 1 2.94 0.81 3.19 1.22 样品2 Sample 2 2.97 0.82 3.24 1.24 样品3 Sample 3 3.55 0.91 4.01 1.38 样品4 Sample 4 3.72 0.91 4.41 1.37 样品5 Sample 5 3.23 0.87 3.61 1.30 样品6 Sample 6 3.40 0.89 3.61 1.37 表 3 正红菇菇位土壤中可培养细菌属间的网络相互作用
Table 3 Network interactions among culturable microbial genera in R. griseocarnosa rhizosphere soil
属名
Genus物种依赖
程度
Species
strength有效
合作值
Effective
partners亲密度
Closeness拟无枝酸菌属 Amycolatopsis 0.04 1.00 0.06 芽孢杆菌属 Bacillus 3.10 5.79 0.08 伯克霍尔德菌氏属 Burkholderia 1.21 4.68 0.08 柠檬酸杆菌属 Citrobacter 0.05 1.00 0.05 紫色杆菌属 Janthinobacterium 0.17 1.00 0.06 赖氨酸芽孢杆菌属 Lysinibacillus 0.19 4.00 0.07 Massilia 0.09 2.00 0.07 小四孢菌属 Microtetraspora 0.04 1.00 0.06 类芽孢杆菌属 Paenibacillus 0.19 4.00 0.07 副伯克霍尔德氏菌属 Paraburkholderia 0.10 1.00 0.06 拉乌尔菌属 Raoultella 0.09 1.00 0.05 红球菌属 Rhodococcus 0.05 1.00 0.06 Rummeliibacillus 0.05 1.00 0.06 沙雷氏菌属 Serratia 0.10 2.00 0.06 链霉菌属 Streptomyces 0.46 3.60 0.07 轮枝链霉菌属 Streptoverticillium 0.10 1.00 0.05 -
[1] 钱建新, 陈仁毅, 张惠兰. 正红菇的生长环境研究 [J]. 福建林业科技, 2003, 30(4):52−54. DOI: 10.3969/j.issn.1002-7351.2003.04.015 QIAN J X, CHEN R Y, ZHANG H L. The study on growth environment of Russula vinosa [J]. Journal of Fujian Forestry Science and Technology, 2003, 30(4): 52−54.(in Chinese) DOI: 10.3969/j.issn.1002-7351.2003.04.015
[2] 涂育合, 陈永聪, 郑肇快. 正红菇依存森林的群落学特征 [J]. 植物资源与环境学报, 2001, 10(2):26−30. DOI: 10.3969/j.issn.1674-7895.2001.02.007 TU Y H, CHEN Y C, ZHENG Z K. Feature of forest community with Russula vinosa lindbl Fr. depending on for existance in Dingban Village of Datian County [J]. Journal of Plant Resources and Environment, 2001, 10(2): 26−30.(in Chinese) DOI: 10.3969/j.issn.1674-7895.2001.02.007
[3] MUNSCH P, ALATOSSAVA T, MARTTINEN N, et al. Pseudomonas costantinii sp. nov., another causal agent of brown blotch disease, isolated from cultivated mushroom sporophores in Finland [J]. International Journal of Systematic and Evolutionary Microbiology, 2002, 52: 1973−1983.
[4] RALPH N, ANDREJA D P, HOBBS P J, et al. Volatile C8 compounds and pseudomonads influence primordium formation of Agaricus bisporus [J]. Mycologia, 2009, 101(5): 583−591. DOI: 10.3852/07-194
[5] YUN Y B, PARK S W, CHA J S, et al. Biological characterization of various strains of Pseudomonas tolaasii that causes brown blotch disease [J]. Journal of the Korean Society for Applied Biological Chemistry, 2013, 56(1): 41−45. DOI: 10.1007/s13765-012-2242-y
[6] 盛江梅, 吴小芹. 菌根真菌与植物根际微生物互作关系研究 [J]. 西北林学院学报, 2007, 22(5):104−108,135. DOI: 10.3969/j.issn.1001-7461.2007.05.026 SHENG J M, WU X Q. Interaction between mycorrhizal fungi and rhizosphere microorganisms [J]. Journal of Northwest Forestry University, 2007, 22(5): 104−108,135.(in Chinese) DOI: 10.3969/j.issn.1001-7461.2007.05.026
[7] GARBAYE J. Helper Bacteria - a New Dimension to the Mycorrhizal Symbiosis [J]. New Phytologist, 1994, 128(2): 197−210. DOI: 10.1111/j.1469-8137.1994.tb04003.x
[8] ASPRAY T J, JONES E E, DAVIES M W, et al. Increased hyphal branching and growth of ectomycorrhizal fungus Lactarius rufus by the helper bacterium Paenibacillus sp [J]. Mycorrhiza, 2013, 23(5): 403−410. DOI: 10.1007/s00572-013-0483-1
[9] FOUNOUNE H, DUPONNOIS R, BÂ A M, et al. Mycorrhiza helper bacteria stimulate ectomycorrhizal symbiosis of Acacia holosericea with Pisolithus alba [J]. New Phytologist, 2002, 153(1): 81−89. DOI: 10.1046/j.0028-646X.2001.00284.x
[10] FOUNOUNE H, DUPONNOIS R, MEYER J M, et al. Interactions between ectomycorrhizal symbiosis and fluorescent pseudomonads on Acacia holosericea: Isolation of mycorrhiza helper bacteria (MHB) from a Soudano-Sahelian soil [J]. FEMS Microbiology Ecology, 2002, 41(1): 37−46. DOI: 10.1111/j.1574-6941.2002.tb00964.x
[11] 陈克华. 野生正红菇生态增产技术示范 [J]. 福建农业, 2007(12):15. CHEN K H. Demonstration of ecological yield increasing technology of wild Pleurotus ostreatus [J]. Fujian Agriculture, 2007(12): 15.(in Chinese)
[12] 郭永红, 罗孝坤, 弓明钦, 等. 大红菇出菇特性研究 [J]. 中国食用菌, 2011, 30(5):30−33. DOI: 10.3969/j.issn.1003-8310.2011.05.012 GUO Y H, LUO X K, GONG M Q, et al. Characteristics research of pruducing mushroom about Russula sanguinea [J]. Edible Fungi of China, 2011, 30(5): 30−33.(in Chinese) DOI: 10.3969/j.issn.1003-8310.2011.05.012
[13] 李忠, 钟莹莹, 陈逸湘. 广东梅州野生灰肉红菇资源调查 [J]. 食用菌, 2012, 34(4):8−9. LI Z, ZHONG Y Y, CHEN Y X. Investigation on wild red mushroom resources in Meizhou, Guangdong Province [J]. Edible Fungi, 2012, 34(4): 8−9.(in Chinese)
[14] 肖冬来, 陈丽华, 陈宇航, 等. 正红菇菌根际土壤细菌多样性 [J]. 福建食用菌, 2014, 2(4):66−72. XIAO D L, CHEN L H, CHEN Y H, et al. Bacterial Diversity of Russula griseocarnosa Mycorrhizosphere Soil [J]. Fujian Mushroom Journal, 2014, 2(4): 66−72.(in Chinese)
[15] 肖冬来, 陈丽华, 陈宇航, 等. 利用变性梯度凝胶电泳分析正红菇菌根围土壤真菌群落多样性 [J]. 热带作物学报, 2013, 34(12):2508−2512. XIAO D L, CHEN L H, CHEN Y H, et al. Analysis of fungal diversity of Russula griseocarnosa mycorrhizosphere soil with denaturing gradient gel electrophoresis [J]. Chinese Journal of Tropical Crops, 2013, 34(12): 2508−2512.(in Chinese)
[16] YU F, LIANG J F, SONG J, et al. Bacterial community selection of Russula griseocarnosa mycosphere soil [J]. Frontiers in Microbiology, 2020, 11: 347. DOI: 10.3389/fmicb.2020.00347
[17] STEFANI F, BELL T H, MARCHAND C, et al. Culture-Dependant and -Independent Methods Capture Different Microbial Community Fractions in Hydrocarbon-Contaminated Soils [J]. PLoS ONE, 2017, 10(6): e0128272.
[18] SHIRLING E B, GOTTLIEB D. Methods for characterization of Streptomyces species [J]. International Journal of Systematic Bacteriology, 1966, 16(3): 313−340. DOI: 10.1099/00207713-16-3-313
[19] WEISBURG W G, BARNS S M, PELLETIER D A, et al. 16S ribosomal DNA amplification for phylogenetic study [J]. Journal of Bacteriology, 1991, 173(2): 697−703. DOI: 10.1128/jb.173.2.697-703.1991
[20] EDWARDS U, ROGALL T, BLÖCKER H, et al. Isolation and direct complete nucleotide determination of entire genes. Characterization of a gene coding for 16S ribosomal RNA [J]. Nucleic Acids Research, 1989, 17(19): 7843−7853. DOI: 10.1093/nar/17.19.7843
[21] 姜华, 何承刚, 于富强, 等. 松口蘑(Tricholoma matsutake)菌塘土壤可培养细菌多样性 [J]. 生态学杂志, 2015, 34(1):150−156. JIANG H, HE C G, YU F Q, et al. Bacterial diversity cultured from shiros of Tricholoma matsutake [J]. Chinese Journal of Ecology, 2015, 34(1): 150−156.(in Chinese)
[22] 余仲东, 唐光辉, 曹支敏. 陕西小叶杨叶内生真菌群体多样性和结构特征 [J]. 林业科学, 2016, 52(6):86−92. YU Z D, TANG G H, CAO Z M. Diversity and community structure of endophytic fungi in the leaves of Populus simonii in Shaanxi Province [J]. Scientia Silvae Sinicae, 2016, 52(6): 86−92.(in Chinese)
[23] 郑梅霞, 朱育菁, 刘波, 等. 云南苍山芽胞杆菌多样性研究 [J]. 福建农业学报, 2019, 34(1):104−116. ZHENG M X, ZHU Y J, LIU B, et al. Microbial diversity of Bacillus community in soils at Cangshan, Yunnan [J]. Fujian Journal of Agricultural Sciences, 2019, 34(1): 104−116.(in Chinese)
[24] DORMANN C F, FRÜND J, BLÜTHGEN N, et al. Indices, Graphs and Null Models: Analyzing Bipartite Ecological Networks [J]. The Open Ecology Journal, 2009, 2(1): 7−24. DOI: 10.2174/1874213000902010007
[25] CORE R, RDCT R, TEAM R, et al. A Language and Environment for Statistical Computing [J]. Computing, 2015, 1: 12−21.
[26] BECKER D M, BAGLEY S T, PODILA G K. Effects of mycorrhizal-associated streptomycetes on growth of Laccaria bicolor, Cenococcum geophilum, and Armillaria species and on gene expression in Laccaria bicolor [J]. Mycologia, 1999, 91(1): 33−40. DOI: 10.2307/3761191
[27] FREY-KLETT P, CHAVATTE M, CLAUSSE M L, et al. Ectomycorrhizal symbiosis affects functional diversity of rhizosphere fluorescent pseudomonads [J]. The New Phytologist, 2005, 165(1): 317−328. DOI: 10.1111/j.1469-8137.2004.01212.x
[28] POOLE E J, BENDING G D, WHIPPS J M, et al. Bacteria associated with Pinus sylvestris-Lactarius rufus ectomycorrhizas and their effects on mycorrhiza formation in vitro [J]. New Phytologist, 2001, 151(3): 743−751. DOI: 10.1046/j.0028-646x.2001.00219.x
[29] 安忠琦, 詹伟, 吴庆珊, 等. 金钗石斛根际可培养细菌多样性及抑菌活性研究 [J]. 云南大学学报(自然科学版), 2018, 40(3):586−602. AN Z Q, ZHAN W, WU Q S, et al. Diversity and antimicrobial activities of the cultivable rhizosphric bacteria fom Dendrobium nobile [J]. Journal of Yunnan University (Natural Sciences Edition), 2018, 40(3): 586−602.(in Chinese)
[30] 万山平, 郑毅, 汤利, 等. 攀枝花块菌-华山松菌根根际土壤可培养细菌的多样性研究 [J]. 植物分类与资源学报, 2015, 37(6):861−870. WAN S P, ZHENG Y, TANG L, et al. Diversity of culturable bacteria associated with tuber panzhihuanense-Pinus armandii ectomycorrhizosphere soil [J]. Plant Diversity and Resources, 2015, 37(6): 861−870.(in Chinese)
[31] BARBIERI E, POTENZA L, ROSSI I, et al. Phylogenetic characterization and in situ detection of a Cytophaga-Flexibacter-Bacteroides phylogroup bacterium in Tuber borchii vittad. Ectomycorrhizal mycelium [J]. Applied and environmental microbiology, 2000, 66(11): 5035−5042. DOI: 10.1128/AEM.66.11.5035-5042.2000
[32] GARBAYE J, DUPONNOIS R, WAHL J L, et al. The bacteria associated with Laccaria laccata ectomycorrhizas or sporocarps: Effect on symbiosis establishment on Douglas fir [J]. Symbiosis, 1990, 9: 267−273.
[33] GARBAYE J, DUPONNOIS R. Specificity and function of mycorrhization helper bacteria (MHB) associated with the Pseudotsuga menziesii-Laccaria laccata symbiosis [J]. Symbiosis, 1992, 14(1-3): 335−344.
[34] OH S Y, LIM Y W. Root-associated bacteria influencing mycelial growth of Tricholoma matsutake (pine mushroom) [J]. Journal of Microbiology, 2018, 56(6): 399−407. DOI: 10.1007/s12275-018-7491-y
[35] 盛江梅, 吴小芹, 侯亮亮, 等. 一株黑松-美味牛肝菌菌根辅助细菌的筛选及鉴定 [J]. 应用与环境生物学报, 2010, 16(5):701−704. SHENG J M, WU X Q, HOU L L, et al. Isolation and identification of a MHB strain from the rhizosphere soil of Pinus thunbergi inoculated with Boletus edulis [J]. Chinese Journal of Applied & Environmental Biology, 2010, 16(5): 701−704.(in Chinese)
[36] RIVETT D W, BELL T. Abundance determines the functional role of bacterial phylotypes in complex communities [J]. Nature Microbiology, 2018, 3(7): 767−772. DOI: 10.1038/s41564-018-0180-0
[37] 禹飞. 灰肉红菇多组学特征分析及其与根际微生物的互作[D]. 北京: 中国林业科学研究院, 2020. YU F. Multi-omics analysis of Russula griseocarnosa and its interaction with mycosphere microorganisms[D]. Beijing: Chinese Academy of Forestry, 2020. (in Chinese)
-
期刊类型引用(8)
1. 曹瑾,苑壮,王红,刘忠宽,冯伟. 绿肥对河北坝上错季蔬菜地土壤的改良效果. 北方园艺. 2024(13): 65-73 .  百度学术
百度学术
2. 秦宇航,吴玉红,王吕,淡亚彬,陈浩,郝兴顺,王地,杨杰. 汉中地区4种冬绿肥还田腐解和养分释放特征. 草业科学. 2024(10): 2306-2315 .  百度学术
百度学术
3. 梁鑫宇,宋明丹,韩梅,李正鹏. 不同农作物秸秆腐解规律及模型预测评价. 核农学报. 2023(06): 1244-1252 .  百度学术
百度学术
4. 高帆,鞠玺凯,郭琪琳,李清华,刘庆. 豌豆茎秆在土壤中的腐解与养分释放特征. 青岛农业大学学报(自然科学版). 2023(03): 220-226 .  百度学术
百度学术
5. 张义宁,王俊. 绿肥腐解过程及其对旱作农田土壤碳氮和玉米产量的影响. 生态学杂志. 2023(11): 2613-2621 .  百度学术
百度学术
6. 谢帅,梁鑫宇,宋明丹,韩梅,李正鹏. 青海高原小麦秸秆–毛叶苕子混合腐解特征. 植物营养与肥料学报. 2022(04): 754-762 .  百度学术
百度学术
7. 王芳芳,梁魁景,刘海鹏,刘国荣,王玉纯,崔文宁. 北方不同树叶腐解的养分释放特征研究. 果树资源学报. 2022(06): 17-19 .  百度学术
百度学术
8. 余跑兰,孙永明,吴艳,肖小军,雷礼文,熊文. 不同还田方式下白三叶腐解特征及对茶叶产量和品质的影响. 华北农学报. 2022(S1): 186-192 .  百度学术
百度学术
其他类型引用(10)




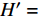
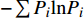
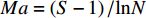
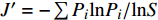
 下载:
下载: