Construction of Rhizobacteria with High-efficiency ACC Deaminase Using Promoter Replacement Technology
-
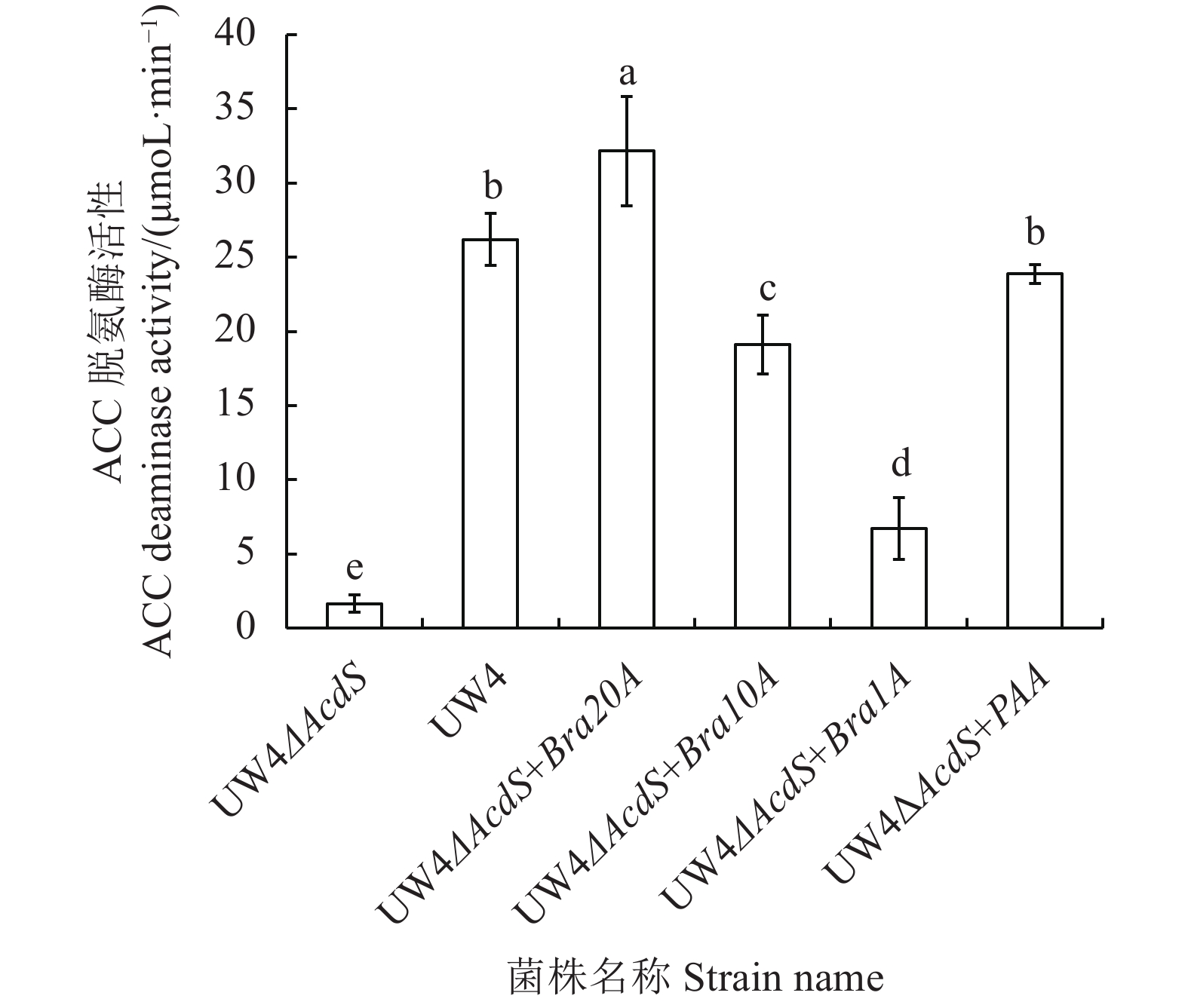
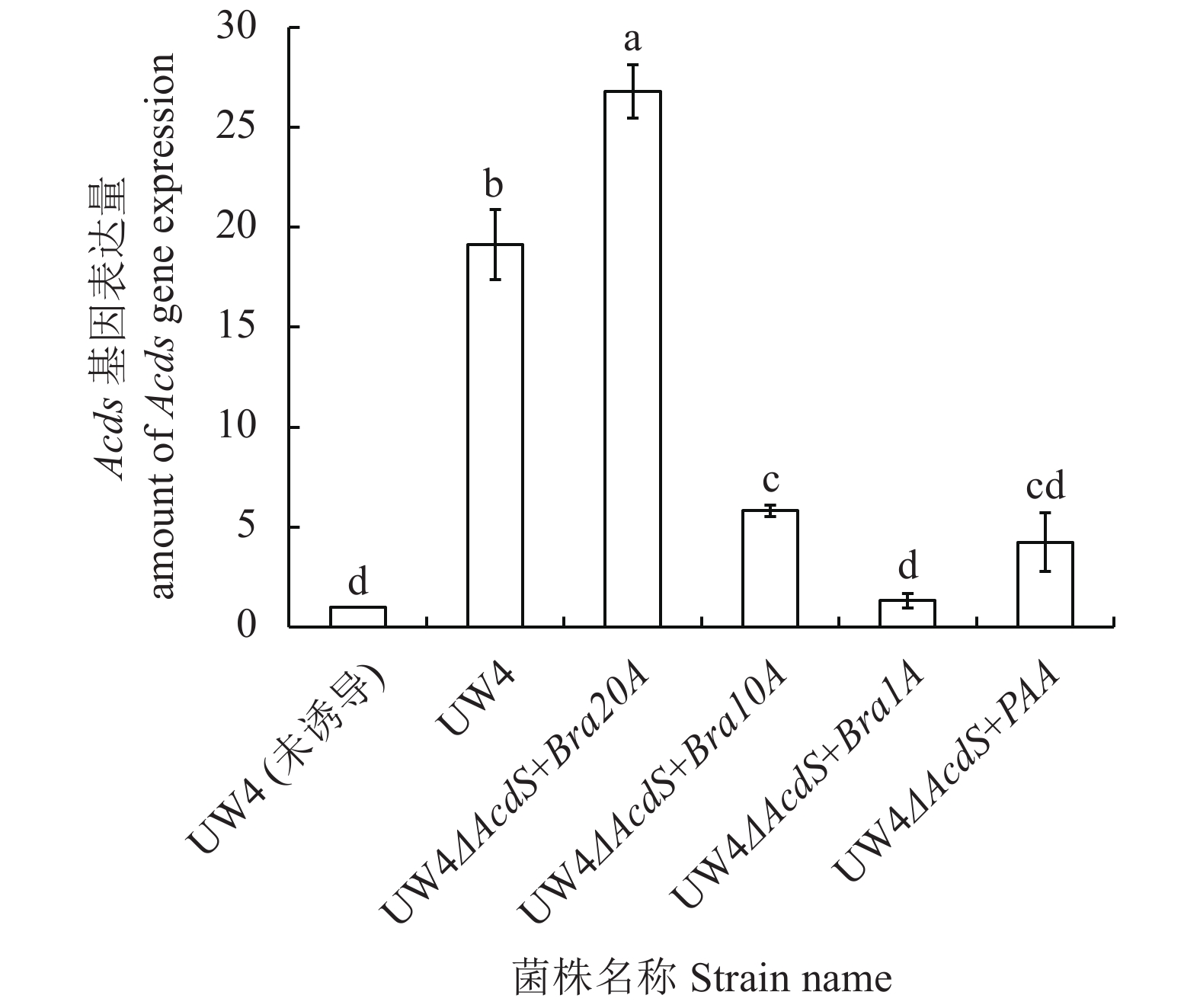
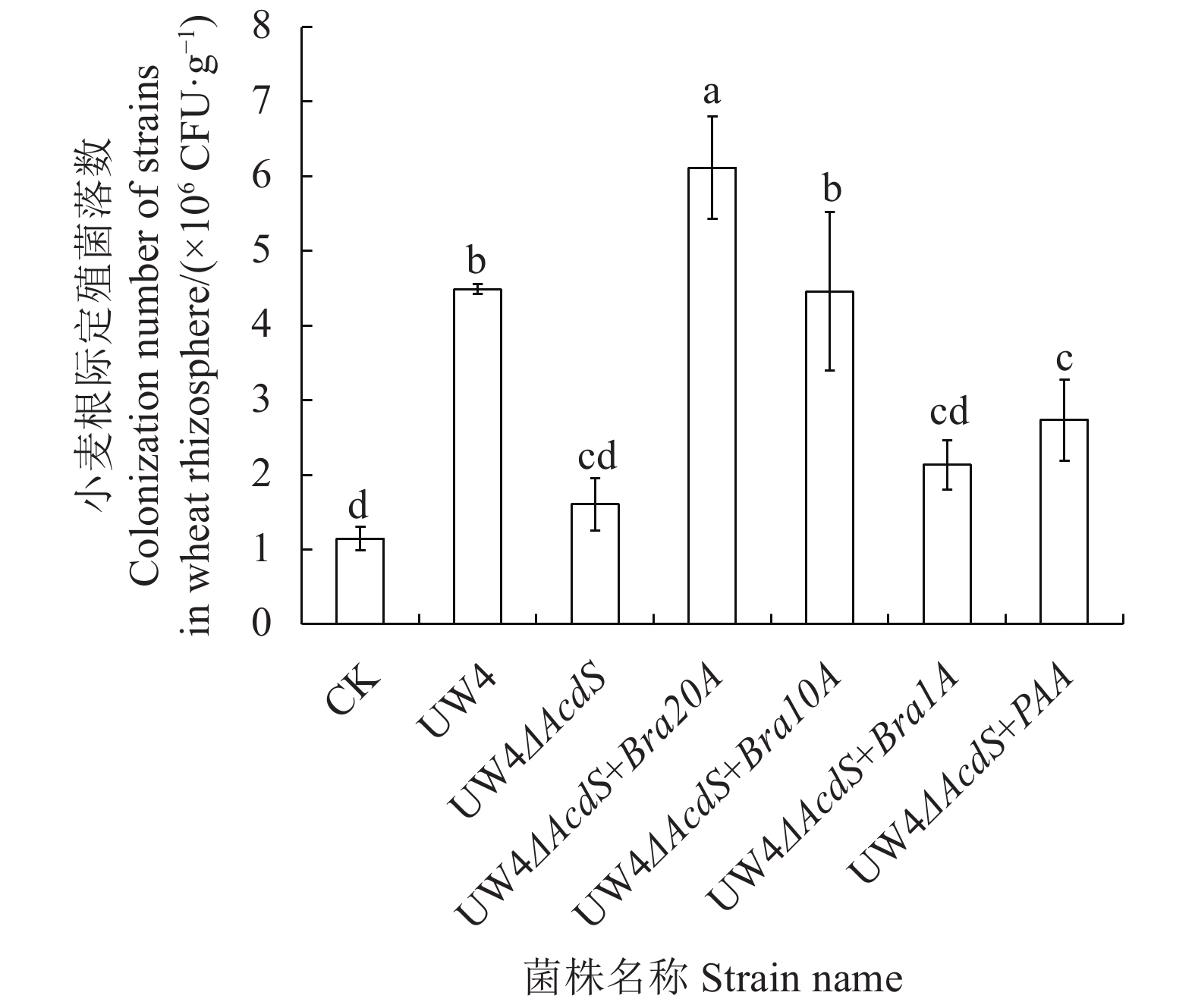
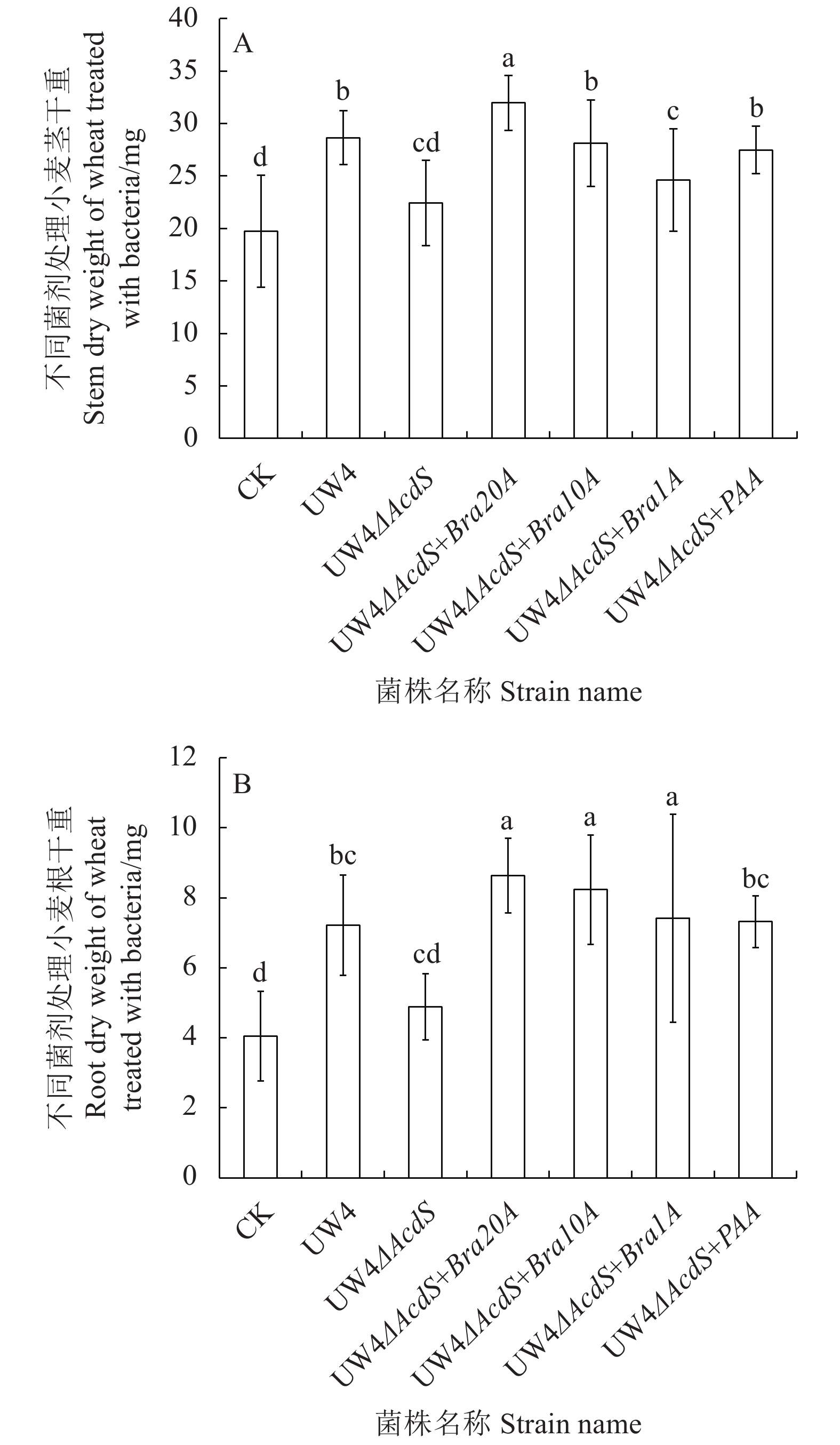
摘要:目的 探究细菌对代谢型趋化物的代谢速率对其趋化作用强弱的影响,同时为选育高效植物根际促生菌(Plant growth promoting rhizobacteria,PGPR)菌株开辟新路径。方法 采用基因克隆得到4种含不同强弱启动子序列的基因片段,将其连至表达载体pBBR1MCS-2,通过三菌杂交接合转移成功构建出生长速率基本一致的4种目标菌株。结果 ACC脱氨酶活性及AcdS基因表达量测定结果说明AcdS基因表达量、ACC脱氨酶活与启动子强弱之间呈现正相关关系;定性趋化结果表明菌株的ACC代谢速率越高,其对ACC的趋化能力也越强;各菌株在小麦根际定殖数量及对小麦生物量影响结果显示:UW4△AcdS+Bra20A菌株定殖数量最多,UW4△AcdS和UW4△AcdS +Bra1A菌株定殖数量较少;UW4△AcdS+Bra20A菌株处理后小麦茎干及根部重量均最重,UW4△AcdS+Bra1A和UW4△AcdS菌株处理后的小麦茎干较轻,UW4△AcdS菌株处理后的小麦根部重量也最轻。结论 ACC脱氨酶活性基本与启动子序列强弱呈正相关。菌株的ACC脱氨酶活性越高,其对ACC代谢速率越高,ACC代谢速率越高,其趋化作用越强,对植株的促生效果也越好。Abstract:Objective Strains of plant growth promoting rhizobacteria (PGPR) with varied ACC metabolic strengths were constructed by promoter replacement technology, and the resulting chemotaxis and effects on the growth of wheat seedlings analyzed.Methods Four gene fragments containing different promoter sequences were cloned and connected to the expression vector pBBR1MCS-2. Using the 3-parent hybridization method, strains of a similar growth rate was constructed.Results Four strains with basically a same growth rate were obtained by the hybridization and transformation method. The AcdS gene expression and ACC deaminase activity were found positively correlated to the strength of the promoter transferred to the strain. The higher the ACC metabolism rate, the greater the chemotactic ability of the strain. In the experiment on wheat seedling root growth and plant biomass with or without the PGPR strains, the colonization number in rhizosphere of UW4△AcdS+Bra20A was the highest, while those of UW4△AcdS and UW4△AcdS+Bra1A lower; the stem and root weight of wheat treated with UW4△AcdS+Bra20A the highest, the stem weight of wheat treated with UW4△AcdS+Bra1A and UW4△AcdS lower, and the root weight of wheat treated with UW4△AcdS lowest.Conclusion The promoter-replaced PGPRs with high enzyme activity displayed high ACC metabolic rate, which enhanced the chemotaxis and the plant-growth promotion ability.
-
Keywords:
- promoter /
- 3-parent hybridization /
- ACC deaminase /
- chemotaxis /
- metabolic rate /
- colonization
-
0. 引 言
【研究意义】荷叶离褶伞(Lyophyllum decastes,Lyd),又称鹿茸菇,属于离褶伞科(Lyophyllaceae),离褶伞属(Lyophyllum,Ly),是一种木腐型珍贵食药用真菌[1-2]。荷叶离褶伞子实体分叉成丛生的细枝,菌盖外观似鹿茸切片[3],广泛分布于北半球温带的山区和丛林之中[2, 4]。荷叶离褶伞口感丝滑爽脆,子实体脂肪含量低,粗纤维成分高,富含人体所需的氨基酸和微量元素[2]。荷叶离褶伞子实体含有粗蛋白22.16%,粗脂肪2.69%,多糖44.29%,粗纤维8.66%,属于高蛋白低脂肪的食用菌[5]。荷叶离褶伞富含的致密型膳食纤维可改善人体肠道的消化能力[6-7],是近年来新兴的工厂化栽培药食兼用菌品种[8-10],且在我国已实现工厂化栽培[2]。当前荷叶离褶伞工厂化栽培仍存在着发菌较慢、成活较迟、易发生有害菌污染等问题[4],获得荷叶离褶伞优良品种是栽培成功的关键[11-13]。筛选出菌种纯度高、菌丝遗传性状稳定、生长活力强和普通培养基易于栽培的品种是当下荷叶离褶伞菌株市场化规模化袋式栽培生产的关键。将本地区与其他地区引进的荷叶离褶伞菌株进行基因组序列突变比对和菌丝形态分析研究,对丰富本地区荷叶离褶伞种质资源有重要意义。【前人研究进展】在自然环境下荷叶离褶伞自身能产生基因组序列突变,且在漫长的进化过程中逐渐累积突变,形成子实体和菌丝形态差异明显的菌株[1]。不同地区的地理气候环境差异较大,对自然突变的荷叶离褶伞有着不同的筛选机制,形成地区之间基因组序列差异较大的菌株,突出表现为菌丝生长和形态的多样性和差异性。荷叶离褶伞子实体由组织化的菌丝体构成,菌丝稳定的生长速度决定子实体的大小[2]。菌丝的生长活力和遗传性状以及将优良性状稳定遗传到子代受其基因序列控制,而基因序列的突变是产生菌丝形态差异的根本原因[4,6]。通过基因序列差异性获得菌丝形态遗传稳定的荷叶离褶伞菌株,能克服由外界不利环境胁迫造成菌丝变异生长的假阳性现象,加快获得菌丝形态优良的菌株。【本研究切入点】 传统分类方法通过子实体生长和菌丝形态对不同地区来源的荷叶离褶伞菌株进行筛选和建立菌种资源库,不但进度慢且无法明确菌丝变异的原因。鉴定到基因组序列碱基突变且变异菌丝稳定遗传是有效增加荷叶离褶伞菌株资源库的关键,而采取真菌18S rRNA(V4)和ITS(ITS1~ITS4)序列结合菌丝形态对不同地区的荷叶离褶伞进行鉴定尚无相关报道。【拟解决的关键问题】通过比较从上海、云南和吉林等地引进的荷叶离褶伞与福建本地栽培的荷叶离褶伞之间基因序列和菌丝生长的差异性,获得基因序列变异大且菌丝形态稳定遗传的荷叶离褶伞,为选育菌丝生长较快且适合工厂袋式栽培的荷叶离褶伞菌株提供参考。
1. 材料与方法
1.1 试验材料
荷叶离褶伞菌株包括从云南、吉林和上海等地引进的Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15和Lyd-LR17,福建省本地工厂化栽培菌株Lyd-LRX和Lyd-LRY。
1.2 试验方法
1.2.1 荷叶离褶伞的纯化培养及电镜观察
荷叶离褶伞菌株均在实验室进行多次菌丝分离和继代培养。挑取荷叶离褶伞菌丝在PDA固体培养基上连续继代培养5次,获得纯化的菌丝体用于后续的菌丝生长试验。将相同量的菌丝块(直径<5 mm)接种至PDA固体培养基上暗培养10 d 和20 d(温度恒定在30 ℃,每个菌株5次重复),用化学方法制备扫描电镜观察样品,对荷叶离褶伞菌丝的生长情况和形态特征进行观察拍照。
1.2.2 荷叶离褶伞基因组DNA的提取
挑选荷叶离褶伞菌丝体(新鲜菌丝1 g左右)到研钵,加入液氮充分碾磨破碎,选取部分粉末转入2 mL离心管中,按照真菌基因组DNA提取试剂盒步骤提取荷叶离褶伞菌丝体DNA。提取的基因组DNA 经 1.20%琼脂糖凝胶电泳检测,确定荷叶离褶伞菌丝体DNA的完整性。
1.2.3 18S rRNA和ITS区段序列的PCR扩增和测序
配制Phanta Max Super-Fidelity DNA Polymerase(诺唯赞Vazyme Biotech, Co., Ltd#P505-d1, 中国)PCR反应试剂。PCR 反应体系(50 μL): 2×Phanta Max Buffer 25 μL, dNTPs 1 μL,引物(10 μmol·L−1)各 2 μL,DNA Polymerase 1 μL, DNA 模板1 μL (<50 ng), ddH2O 18 μL。PCR反应条件:95 °C变性 3 min;95 °C 变性15 s,55 °C 退火15 s,72 °C 延伸1 min,35 个循环;72 °C 总延伸5 min。对18S rRNA的V4变量区(623~853)和ITS1~ITS4区段分别进行PCR扩增。18S rRNA的引物序列为18S_3NDF(正向引物序列):5′ -GGCAAGTCTGGTGCCAG-3′;18S_V4_euk_R2(反向引物序列): 5′-ACGGTATCT(AG)ATC(AG)TCTTCG-3′。ITS区段引物序列为ITS1(正向引物序列): 5′-TCCGTAGGTGAACCTGCGG-3′;ITS4(反向引物序列): 5′-TCCTCCGCTTATTGATATGC-3′。引物由上海生工生物工程有限公司合成。采用PCR仪Eppendorf 5331(德国eppendorf公司)进行PCR扩增,PCR产物用1%琼脂糖凝胶电泳检测,确定PCR产物的大小和特异性。PCR产物使用通用型DNA纯化回收试剂盒进行回收(TIANGEN DP214,天根生化科技有限公司,中国)。纯化的PCR产物送北京擎科生物科技有限公司进行测序分析。
1.2.4 18S rRNA和ITS序列的同源性比对
测序获得荷叶离褶伞18S rRNA的V4区序列和ITS的ITS1~ITS4区序列,去除杂峰序列后,登录NCBI Blast(https://blast.ncbi.nlm.nih.gov/Blast.cgi)并选用Nucleotide BLAST进行核酸序列同源性比对,同时在NCBI GenBank获得同源性较高(>90%)的部分荷叶离褶伞菌株序列编号(Accession)及其序列信息,使用生物软件DNAMAN 9进行多重序列比对。
1.2.5 构建系统发育进化树
采用MEGA 7.0.26(MolecularEvolutionary Genetics Analysis,http://www.megasoftware.net/)软件分别对18S rRNA(V4)和ITS(ITS1~ITS4)序列同源性较高的荷叶离褶伞序列构建系统进化树。用 ClustalW 进行多序列匹配比对,重复数(Bootstrap)为1000,以Neighbor-Joining(NJ)法构建系统发育进化树。
2. 结果与分析
2.1 荷叶离褶伞菌株18S rRNA-ITS同源性分析和多重序列比对
18S rRNA(V4)序列的NCBI Blast比对结果显示,外地离褶伞菌株(Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15和Lyd-LR17)、福建本地离褶伞菌株(Lyd-LRX和Lyd-LRY)与NCBI登录的荷叶离褶伞菌株(Lyd-PBM3069和Lyd-JM87/16)序列同源性为97.55%~98.88%(表1);与NCBI登录的离褶伞属菌株Ly-shimeji(D84555.1)、Ly-sp. MSG166、Ly-sp. PBM 2688、Ly-praslinense 10295、Ly-leucophaeatum HAe251和Ly-shimeji(D84553.1)的序列同源性为94.21%~100%。序列同源性均大于94%,确定试验菌株Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15和Lyd-LR17菌株为荷叶离褶伞。
表 1 荷叶离褶伞菌株的18S rRNA(V4)序列同源性分析Table 1. Homology of 18S rRNA (V4) sequences in Lyd strains(单位:%) 菌株 Strain Lyd-LR1 Lyd-LR6 Lyd-LR10 Lyd-LR15 Lyd-LR17 Lyd-LRX Lyd-LRY Lyd-PBM3069 98.45 98.22 98.88 99.11 98.48 98.66 98.21 Lyd-JM87/16 98.23 97.55 98.66 98.44 97.83 97.99 97.54 Ly-shimeji(D84555.1) 97.96 97.90 98.59 98.83 97.95 98.13 97.90 Ly-sp. MSG166 96.69 96.44 97.10 97.32 96.96 96.88 96.64 Ly-sp. PBM 2688 96.47 96.21 96.88 97.10 96.75 96.65 96.42 Ly-praslinense 10295 94.92 94.65 95.31 95.54 95.23 95.09 94.85 Ly-leucophaeatum HAe251 94.48 94.21 94.87 95.09 94.79 94.64 94.41 Ly-shimeji(D84553.1) 100.00 93.44 98.36 98.36 100.00 95.08 93.22 根据18S rRNA多重序列比对结果(图1),与NCBI登录菌株Lyd-PBM3069和Lyd-JM87/16相比较,外地荷叶离褶伞菌株Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15和Lyd-LR17以及福建本地荷叶离褶伞菌株Lyd-LRX和Lyd-LRY均发生4处碱基替换以及1处碱基缺失。与本地荷叶离褶伞菌株Lyd-LRX和Lyd-LRY相比较,外地荷叶离褶伞菌株Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15和Lyd-LR17部分发生3处碱基缺失/插入突变。外地荷叶离褶伞菌株Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15和Lyd-LR17在真菌非常保守的18S rRNA(V4)序列发生碱基替换和碱基缺失的多处突变,需继续以碱基突变概率较高的ITS(ITS1-ITS4)序列进一步分析外地荷叶离褶伞菌株的基因序列差异性。
ITS(ITS1-ITS4)序列NCBI Blast比对结果显示,与Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15、Lyd-LR17、Lyd-LRX和Lyd-LRY菌株序列均高度同源(>80%)的荷叶离褶伞菌株达到45个,但序列同源性下降至80.09%~90.33%(表2),提示菌株的ITS(ITS1–ITS4)序列已发生较高比例的碱基突变。根据ITS(ITS1–ITS4)多重序列比对结果,与NCBI登录菌株Lyd-Ld418和Lyd-ywy1相比较,Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15、Lyd-LR17、Lyd-LRX和Lyd-LRY菌株均发生基本相同的7处碱基替换、1处碱基缺失以及1处碱基插入。与本地荷叶离褶伞菌株Lyd-LRX和Lyd-LRY相比较,外地荷叶离褶伞菌株Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15和Lyd-LR17菌株均发生相同的5处碱基替换以及1处碱基插入。与其他荷叶离褶伞菌株相比较, Lyd-LR1、Lyd-LR6菌株发生更高的碱基突变(图2)。与NCBI登录荷叶离褶伞菌株相比,外地荷叶离褶伞菌株和福建本地荷叶离褶伞菌株的ITS(ITS1–ITS4)序列均发生较高比例的碱基替换和碱基缺失,且外地荷叶离褶伞菌株Lyd-LR1和Lyd-LR6的碱基突变明显高于其他荷叶离褶伞菌株,而Lyd-LR6菌株ITS(ITS1–ITS4)序列发生碱基替换和碱基缺失的突变比例高于Lyd-LR1菌株。
表 2 荷叶离褶伞菌株的ITS(ITS1–ITS4)序列同源性分析Table 2. Homology of ITS (ITS1–ITS4) sequences in Lyd strains(单位:%) 菌株
StrainLyd-LR1 Lyd-LR6 Lyd-LR10 Lyd-LR15 Lyd-LR17 Lyd-LRX Lyd-LRY Lyd-dcy2255 89.72 83.52 90.33 84.64 89.38 86.52 82.92 Lyd-ywy1 89.42 82.76 90.05 84.33 89.06 86.06 82.49 Lyd-Ld418 89.27 82.92 89.94 84.45 89.24 85.67 82.58 Lyd-060926 89.30 82.82 90.08 84.07 88.79 85.80 82.24 Lyd-060801 89.15 82.66 89.94 83.94 88.61 85.91 82.06 Lyd-061011 88.98 82.35 89.77 83.33 88.02 85.76 81.65 Lyd-iNAT 88.34 82.37 88.99 83.26 87.95 85.23 81.26 Lyd-22/14DMRJU 88.69 81.74 88.98 83.14 88.69 85.60 80.09 2.2 荷叶离褶伞菌株的18S rRNA-ITS系统发育进化树分析
为明确外地荷叶离褶伞菌株和福建本地荷叶离褶伞菌株之间的进化关系,同时根据NCBI Blast提供的同源性分析结果,选取部分NCBI登录的荷叶离褶伞、离褶伞属菌株和其食用菌[18S rRNA(V4)同源性>80%]进行系统发育进化树分析。18S rRNA(V4区)序列的进化树分析结果显示,外地荷叶离褶伞菌株(Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15和Lyd-LR17)聚为一类,再与福建本地荷叶离褶伞菌株(Lyd-LRX和Lyd-LRY)聚为一类,最后与NCBI登录的荷叶离褶伞菌株(Lyophyllum decastes PBM3069和Lyophyllum decastes JM87/16)聚合(图3),但均不与NCBI登录的其他食用菌进行聚合,说明外地荷叶离褶伞菌株在遗传距离上有着近似的亲缘关系,但与福建本地荷叶离褶伞已存在遗传进化上的差别,而与NCBI登录的荷叶离褶伞和其他食用菌有较远的遗传距离和亲缘关系。
ITS(ITS1–ITS4)序列的系统发育进化树分析结果显示,Lyd-LR1和Lyd-LR6菌株聚合在一起,而Lyd-LR10、Lyd-LR15、Lyd-LR17、Lyd-LRX和Lyd-LRY菌株聚合在一起,再与NCBI登录的荷叶离褶伞菌株进行聚合(图4)。从遗传距离和进化关系进行分析,外地荷叶离褶伞菌株Lyd-LR10、Lyd-LR15和Lyd-LR17与福建本地离褶伞菌株Lyd-LRX和Lyd-LRY亲缘关系更接近。Lyd-LR1和Lyd-LR6菌株保持相近的亲缘关系,与其他荷叶离褶伞菌株亲缘关系较远。外地荷叶离褶伞菌株Lyd-LR1和Lyd-LR6的遗传进化距离大于其他荷叶离褶伞菌株,表现出较远的种属亲缘关系。
2.3 荷叶离褶伞的菌丝体生长和菌丝形态观察
将外地荷叶离褶伞菌株(Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15和Lyd-LR17)接菌至PDA培养基暗培养10 d 和20 d(30 ℃)。在菌丝体生长的第10 天,Lyd-LR6菌株在生长圈边缘形成厚实的菌丝体且菌丝体生长的速度明显快于Lyd-LR1、Lyd-LR10、Lyd-LR15和Lyd-LR17菌株;Lyd-LR10和Lyd-LR17菌株的菌丝体生长速度基本一致;Lyd-LR1和Lyd-LR15菌株的菌丝体厚度较低且菌丝体生长速度慢于Lyd-LR6、Lyd-LR10和Lyd-LR17菌株(图5)。在菌丝体生长的第20天,Lyd-LR1菌株的菌丝体不能铺满整个培养基,菌丝呈现松散放射状且菌丝厚度较薄,生长较慢;Lyd-LR6厚实的菌丝体呈平铺状且铺满整个培养基,菌丝厚度较高且均一,生长速度较快;Lyd-LR10、Lyd-LR15、Lyd-LR17、Lyd-LRX和Lyd-LRY菌丝体生长基本相同且菌丝体铺满整个培养基,菌丝体厚度较高且呈现突起状,生长较为迅速(图5)。荷叶离褶伞的菌丝体生长结果显示,Lyd-LR6菌丝体生长迅速,而Lyd-LR1菌丝体生长缓慢。Lyd-LR1和Lyd-LR6菌株之间的菌丝体生长速度差别明显且均不同于Lyd-LR10、Lyd-LR15和Lyd-LR17菌株的菌丝体生长。
挑取生长20 d的外地荷叶离褶伞菌株菌丝体进行扫描电镜观察,Lyd-LR10、Lyd-LR15、Lyd-LR17、Lyd-LRX和Lyd-LRY菌株的菌丝形态基本相似,均为圆柱形的菌丝,菌丝饱满且交织在一起;Lyd-LR1菌株的菌丝呈现梭子状,菌丝生长不规则且有聚合生长的倾向;Lyd-LR6菌株的菌丝生长更加不规则且呈现凹陷状,菌丝明显聚合生长(图6)。与Lyd-LR10、Lyd-LR15、Lyd-LR17、Lyd-LRX和Lyd-LRY菌株相比较,Lyd-LR1和Lyd-LR6菌株的菌丝形态发生了明显的变异,且Lyd-LR6菌株的菌丝形态变异幅度大于Lyd-LR1菌株。
3. 讨论与结论
快速鉴定外地引进的菌株是提高荷叶离褶伞品种选育效率的重要手段。目前对荷叶离褶伞菌株的研究主要集中在改良栽培技术、营养成分分析、甾类和多糖等活性物质的提取纯化[14-16],缺乏对于不同地区来源的荷叶离褶伞菌株的有效快速鉴定。随着分子生物学的发展,通过PCR扩增部分基因组序列对不同地区的食用菌菌株进行鉴定和确定其种属关系及基因组碱基序列变异情况的方法逐渐成熟,为优良菌株的筛选提供快捷方法[17]。采用传统的形态学鉴定食用菌有易于观察和比较优势,但菌丝形态多样性易受到外界因素的影响而出现假阳性。根据基因组序列同源性的特征(>95%)可确定食用菌菌株之间的序列差异性和种属亲缘关系[18]。基因组碱基序列的鉴定方法通过分析碱基序列的突变而明确菌丝形态变异和菌丝体性状能遗传稳定的本质原因。利用菌体形态学和基因组序列测序结合的方法,鉴定了蘑菇属的双孢蘑菇(Agaricus bisporus),香菇属的香菇(Lentinula edodes)和小包脚菇属的草菇(Volvariella volvacea)[19],以及快速将野生菌鉴定为Paxillus ammoniavirescens[18]。基因组部分序列测序和菌丝形态的结合是鉴定食用菌新菌株的有效方法。真菌食用菌具有物种多样性和序列保守性,其18S rRNA(V4)序列在遗传进化上较为保守,而ITS(ITS1–ITS4)序列常发生较高概率的碱基突变,常用于快速鉴定食用菌的基因序列同源性、种属关系和碱基序列突变程度,为进一步分析菌丝形态变异提供依据。
外地引进的荷叶离褶伞菌株(Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15和Lyd-LR17)与福建本地的荷叶离褶伞菌株(Lyd-LRX和Lyd-LRY)以及NCBI登录的荷叶离褶伞菌株在18S rRNA-ITS序列的系统发育进化树均聚合在一起,且菌株间的18S rRNA 序列同源性大于97.55%,与离褶伞菌株的同源性也大于94.21%,确定此次从外地引进的食用菌均为荷叶离褶伞菌株。18S rRNA-ITS序列的同源性分析、多重序列比对和遗传进化树分析均显示外地荷叶离褶伞菌株(Lyd-LR1、Lyd-LR6、Lyd-LR10、Lyd-LR15和Lyd-LR17)和福建本地荷叶离褶伞菌株(Lyd-LRX和Lyd-LRY)存在较高比例的碱基序列突变,ITS序列同源性下降至80.09%~89.72%,从而明确外地荷叶离褶伞菌株已明显不同于福建本地荷叶离褶伞菌株以及NCBI登录的荷叶离褶伞菌株(45个)。这些外地引进的荷叶离褶伞菌株具有丰富本地区荷叶离褶伞菌株资源库的研究价值。ITS(ITS1–ITS4)的多重序列比对和遗传进化树显示Lyd-LR1和Lyd-LR6菌株的基因序列差异性和碱基序列突变比例明显区别于其他荷叶离褶伞菌株,且仅在Lyd-LR6菌株发生的碱基替换和碱基缺失明显高于Lyd-LR1、Lyd-LR10、Lyd-LR15、Lyd-LR17、Lyd-LRX和Lyd-LRY。菌丝体生长速度和菌丝形态进一步表明,Lyd-LR1梭子状不规则菌丝和松散放射状菌丝体以及Lyd-LR6凹陷状聚合生长菌丝和边缘厚实平铺状菌丝体,均不同于Lyd-LR10、Lyd-LR15和Lyd-LR17圆柱形饱满的菌丝和突起状菌丝体,且Lyd-LR6菌丝体生长速度明显快于Lyd-LR1,Lyd-LR6菌丝形态的变异程度也高于Lyd-LR1。基因组碱基序列高比例的突变是造成Lyd-LR1和Lyd-LR6菌株菌丝体生长速度和菌丝形态明显不同于Lyd-LR10、Lyd-LR15、Lyd-LR17、Lyd-LRX和Lyd-LRY的根本原因。而与Lyd-LR1菌株相比,Lyd-LR6菌株的碱基序列突变比例更高且碱基突变位置更多变(碱基替换和碱基缺失),使得Lyd-LR6菌株的菌丝形态明显区别于Lyd-LR1菌株。
-
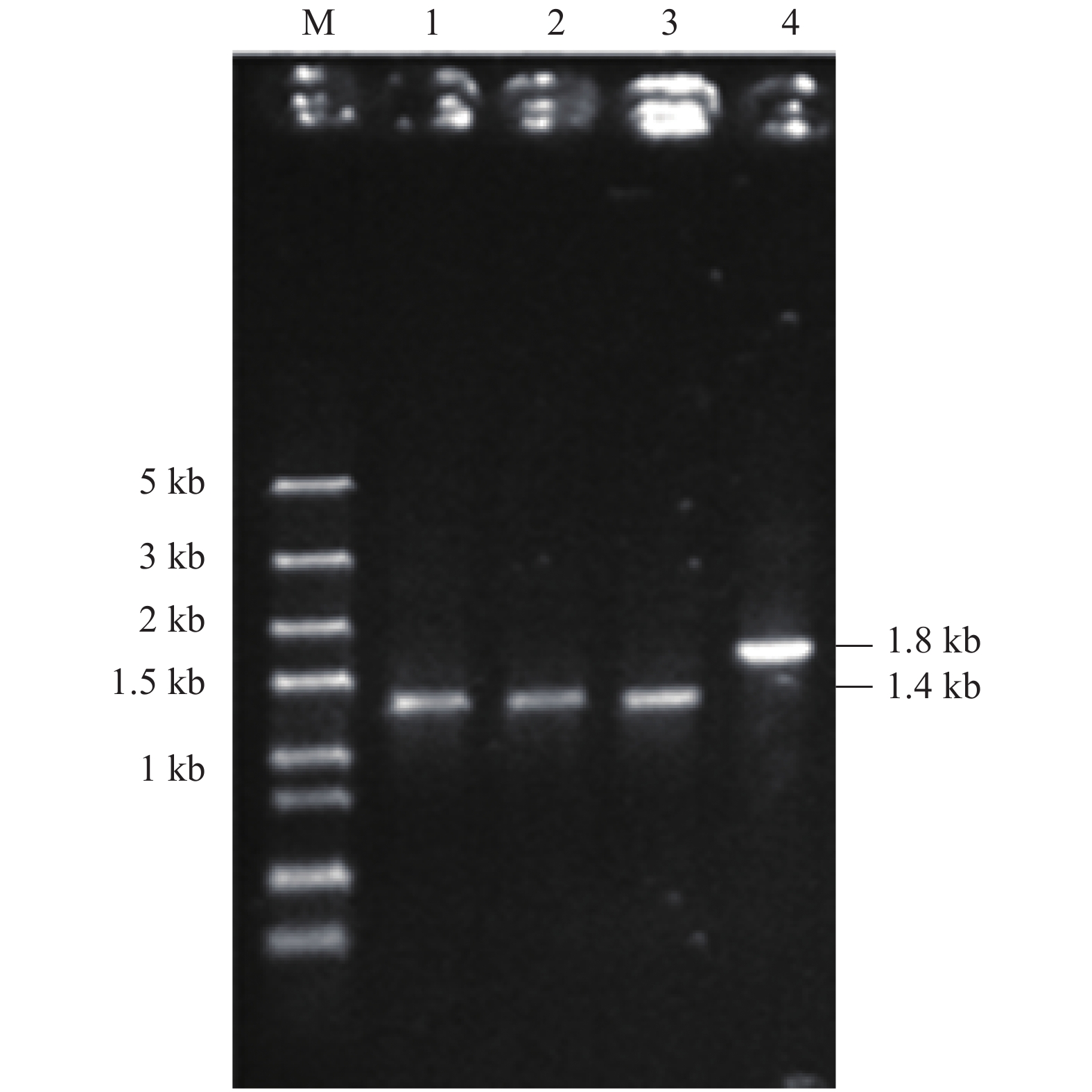
表 1 含不同强度启动子片段的引物设计
Table 1 Primers with promoters of different strength
引物名称
Name引物序列
Sequence扩增产物
PCR productBra20A-F CGGGATCCAATACTTGACATATCACTGTGATTA Bra20A CATATAATATGCGAAATCTGTAAGGCTAGCCAG GCTACACAGGGAATGAACCTGAATCGTTTTG Bra10A-F CGGGATCCACCTATTGACAATTAAAGGCTAAAA Bra10A TGCTATAATTCCACAAATCTGTAAGGCTAGCCA GGCTACACAGGGAATGAACCTGAATCGTTTTG Bra1A-F CGGGATCCTCCCTTTGATATTGCATCCCGCGTAT Bra1A ATAATATGTCAAATCTGTAAGGCTAGCCAGGCT ACACAGGGAATGAACCTGAATCGTTTTG A-R CCAAGCTTGTCAATCACGTATTTGGGTAAC 各片段下游引物 PAA-F CGGGATCCGGTTGAAACTCTGG PAA PAA-R CCAAGCTTTTTGACCCAGAC 注:上游酶切位点为Bam H Ⅰ,下游酶切位点为Hind III,分别以下划线标注。
Note: Upstream restriction sites Bam H I and downstream restriction sites Hind III are underlined. -
[1] PIZARRO-TOBÍAS P, UDAONDO Z, ROCA A, et al. Events in root colonization by Pseudomonas putida[M]. Netherlands: Springer, 2015.
[2] ZHANG F S, SHEN J B, ZHANG J L, et al. Rhizosphere processes and management for improving nutrient use efficiency and crop productivity[C]//International symposium of molecular environmental soil science at the interfaces in the earth's critical zone; ISMESS. College of Resource and Environmental Sciences, China Agricultural University. Amsterdam: Elsevier, 2009: 1–32.
[3] 沈德龙, 李俊, 姜昕. 我国微生物肥料产业现状及发展方向 [J]. 微生物学杂志, 2013, 33(3):1−4. SHEN D L, LI J, JIANG X. Status quo and development of microbial fertilizer industry in China [J]. Journal of Microbiology, 2013, 33(3): 1−4.(in Chinese)
[4] 胡小加, 余常兵, 李银水, 等. 枯草芽孢杆菌Tu-100对油菜根系分泌物所含氨基酸的趋化性研究 [J]. 土壤学报, 2010, 47(6):1243−1248. DOI: 10.11766/trxb200908070341 HU X J, YU C B, LI Y S, et al. Chemotaxis of Bacillus subtilis tu-100 toward amino acids in root exudates of oilseed rape [J]. Acta Pedologica Sinica, 2010, 47(6): 1243−1248.(in Chinese) DOI: 10.11766/trxb200908070341
[5] BULL C T. Relationship between root colonization and suppression of Gaeumannomyces graminis var. tritici by Pseudomonas fluorescens Strain 2-79 [J]. Phytopathology, 1991, 81(9): 954.
[6] PII Y, MIMMO T, TOMASI N, et al. Microbial interactions in the rhizosphere: Beneficial influences of plant growth-promoting rhizobacteria on nutrient acquisition process. A review [J]. Biology and Fertility of Soils, 2015, 51(4): 403−415.
[7] 王淼, 张莉, 刘瑛, 等. 趋化性参与内生细菌336x在小麦根系的内生定殖 [J]. 河南大学学报(自然科学版), 2012, 42(6):736−741. WANG M, ZHANG L, LIU Y, et al. Chemotaxis of endophytic bacteria 336x involved in endophytic colonization in wheat roots [J]. Journal of Henan University (Natural Science Edition), 2012, 42(6): 736−741.(in Chinese)
[8] LI T, ZHANG J, SHEN C H, et al. 1-aminocyclopropane-1-carboxylate: A novel and strong chemoattractant for the plant beneficial rhizobacterium Pseudomonas putida UW4 [J]. Molecular Plant-Microbe Interactions, 2019, 32(6): 750−759.
[9] SHAHAROONA B, JAMRO G M, ZAHIR Z A, et al. Effectiveness of various Pseudomonas spp. and Burkholderia caryophylli containing ACC-deaminase for improving growth and yield of wheat (Triticum aestivum L. ) [J]. Journal of Microbiology and Biotechnology, 2007, 17(8): 1300−1307.
[10] BRAATSCH S, HELMARK S, KRANZ H, et al. Escherichia coli strains with promoter libraries constructed by Red/ET recombination pave the way for transcriptional fine-tuning [J]. BioTechniques, 2008, 45(3): 335−337. DOI: 10.2144/000112907
[11] PENROSE D M, GLICK B R. Methods for isolating and characterizing ACC deaminase-containing plant growth-promoting rhizobacteria [J]. Physiologia Plantarum, 2003, 118(1): 10−15.
[12] TSO W W, ADLER J. Negative chemotaxis in Escherichia coli [J]. Journal of Bacteriology, 1974, 118(2): 560−576. DOI: 10.1128/jb.118.2.560-576.1974
[13] PORTER S L, WADHAMS G H, ARMITAGE J P. Signal processing in complex chemotaxis pathways [J]. Nature Reviews Microbiology, 2011, 9(3): 153−165.
[14] NI B, HUANG Z, FAN Z, et al. Comamonas testosteroni uses a chemoreceptor for tricarboxylic acid cycle intermediates to trigger chemotactic responses towards aromatic compounds [J]. Molecular Microbiology, 2013, 90(4): 813−823.
[15] LACAL J, ALFONSO C, LIU X X, et al. Identification of a chemoreceptor for tricarboxylic acid cycle intermediates: Differential chemotactic response towards receptor ligands [J]. The Journal of Biological Chemistry, 2010, 285(30): 23126−23136. DOI: 10.1074/jbc.M110.110403
[16] LUU R A, KOOTSTRA J D, NESTERYUK V, et al. Integration of chemotaxis, transport and catabolism in Pseudomonas putida and identification of the aromatic acid chemoreceptor PcaY [J]. Molecular Microbiology, 2015, 96(1): 134−147.
[17] JIANG X Z, LIU H J, NIU Y C, et al. Determination of temperature sensitive plasmid copy number in Escherichia coli by absolute and relative real time quantitation PCR [J]. Jundishapur Journal of Microbiology, 2017, 10(7): e14600.




 下载:
下载: