Microbial Community Structure and Diversity in Rhizosphere and Non-rhizosphere Soils at Fields of Three Varieties of Mangrove Plants
-
摘要:目的 研究秋茄、白骨壤、桐花树等3种红树植物根际与非根际土壤细菌群落结构及多样性特征,为阐明土壤微生物群落对红树植物的生长及其在红树林生态系统保护与修复中的重要作用提供科学支撑。方法 提取3种红树植物根际土壤及同一区域非根际土壤宏基因组DNA,利用Illumina NovaSeq测序技术,分析土壤细菌群落结构及多样性。结果 ①4组土壤样本共鉴定出细菌39门、55纲、126目、244科、511属,非根际土壤样本OTU数目大于3组红树植物土壤样本,同时其在观测物种数和多样性指数上也表现出了更高的丰富性与多样性;②4组土壤样本中变形菌门的相对丰度均大于60%,占据绝对主导地位。4组土壤样本之间相比较,秋茄根际土壤样本中厚壁菌门和梭杆菌门有更高的丰度,桐花树根际土壤样本里变形菌门的丰度有一定优势,白骨壤根际土壤样本里拟杆菌门的丰度高于其他3组。非根际土壤样本里的芽单胞菌门略微占有优势,但是其在拟杆菌门的丰度却低于3组根际土壤样本;③PCA分析发现桐花树与白骨壤根际土壤的群落组成较为接近,4组样本的根际细菌群落对应的 COG 类目大致相同。结论 红树植物非根际土壤的细菌群落有更高的多样性,不同红树植物的重要作用类群不同。Abstract:Objective Structure and diversity of the microbial communities in the rhizosphere and non-rhizosphere soils on lots grown with different mangrove plants were studied for improved protection and restoration of the ecosystem.Methods Metagenomic DNAs were extracted from the rhizosphere and field soils on Kandelia obovata, Avicennia marina, Aegiceras corniculatum lots and the field soil at an open lot for Illumina NovaSeq sequencing to determine the microbial community structure and diversity.Results ① A total of 39 phyla, 55 classes, 126 orders, 244 families, and 511 genera of microbes were identified in the soils. There were more OTUs in the non-rhizosphere field soils, as well as higher number of species and diversity index, than in the rhizosphere soils. ② In the 4 field soils, the greater than 60% relative abundances of Proteobacteria were significantly dominant in the communities. Firmicutes and Fusobacterium were more abundant on the lot at K. obovate field; Proteobacteria at A. corniculatum field; and Bacteroidetes at A. marina field. At phylum level, Bacillus was slightly more in the field soils, but Bacteroidetes less, than in their rhizosphere counterparts. ③ The PCA analysis indicated the microbial compositions of the rhizosphere soils at the A. corniculatum and A. marina fields to be similar, and the corresponding COG categories of communities in rhizosphere soils roughly the same.Conclusion The microbial communities in the non-rhizosphere field soils were more diverse than in the rhizosphere soils at the lots of various mangrove plants. The major functional microbes in the communities differed by the mangrove variety that grew on the soil.
-
Keywords:
- Mangrove plants /
- microbes /
- community structure /
- diversity
-
0. 引言
【研究意义】红树林多分布在热带与亚热带地区,其生存环境特殊,兼具陆地生态与海洋生态系统的特征,具有保护海岸、维护生物多样性等重要作用,被誉为“海岸卫士”[1]。但随着人为活动影响的加剧以及气候变化等因素,红树林面积正大量减少[2],如何对红树林湿地生态系统进行生态保护与修复,已成为湿地研究工作的重点内容。红树林蕴含的微生物资源丰富,而根际微生物已被证明在植物生长发育和防治病害过程中起到重要作用。同时土壤微生物在土壤养分循环、矿物质分解等方面起到重要作用,研究它们的群落结构对生态环境变化起到预警作用[3]。因此研究红树植物根际土壤微生物群落对于红树林湿地生态系统具有重要意义。【前人研究进展】红树林土壤微生物极具珍稀性[4]且多样性高于海岸非红树林区[5],已逐渐成为研究热点。殷萌清等对不同生境下红树林土壤微生物群落结构差异进行了研究[6];蓝亦琦等[7]将红树林引种到淡水区域研究其土壤微生物群落结构变化;研究表明红树林生态系统中细菌为微生物主要类群[8],而聚焦于不同红树植物根际土壤细菌群落结构差异的研究目前较少。【本研究切入点】秋茄(Kandelia candel)、白骨壤(Avicennia marina)、桐花树(Aegiceras corniculatum)作为福建红树植物的主要优势树种,其根际土壤细菌与非根际细菌群落的结构差异与多样性特征有待深入探讨。【拟解决的关键问题】选取同一生长区域内的3种红树植物的根际土壤以及它们的生长区内非根际土壤,通过Illumina高通量测序技术[9]研究红树植物根际土壤细菌与非根际细菌群落的结构差异与多样性特征。旨在了解红树植物根际与非根际土壤细菌群落结构差异,对进一步研究土壤细菌与红树植物生长发育及生态功能的关联性具有重要意义,同时为探究红树植物最适生长土壤微生态环境以及通过微生物调控红树林湿地生态系统的保护与修复奠定基础。
1. 材料与方法
1.1 材料
选取福建省泉州湾河口湿地自然保护区内分布在同一生长区域的秋茄、白骨壤、桐花树等红树植物,分别采集其根际土壤,以及它们的生长区内非根际土壤,4组样本按顺序编号为QQ.D、QB.D、QT.D、QCK.D,每组取3个重复,依次编号为01、02、03。
1.2 土壤样品采集
在选定的采样区内,采用多点混合采样法,采集每种红树植物20~40 cm的根际土壤样本,剔除植物残体、根系、小石块等杂质后,装入无菌密封袋,置于冰盒内,同时采集它们的生长区内非根区采集土壤样本,送回实验室,−20 ℃ 条件下冷冻保藏待用。
1.3 基因组DNA提取及细菌群落组成测定
采用天根土壤基因组DNA提取试剂盒提取土壤样本的总DNA,以提取土样的DNA为模板,采用细菌16S rDNA特异性引物799F (5′-AACMGGATTAGATA CCCKG-3′)和1193R (5′-ACGTCATCCCCACCTTC C-3′)扩增各样本 16SrRNA 基因的 V5~V7 可 变区[10]。PCR体系:5 μL 10×Buffer;5 μL dNTPs (2 mmol·L−1);1 μL DNA聚合酶;引物各1.5 μL;50 ngDNA模板;双蒸水补足50 μL。反应条件:94 ℃ 10 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 30 s,35 个循环;72 ℃ 10 min。随后送至北京诺禾致源科技股份有限公司进行Illumina NovaSeq高通量测序。
1.4 数据分析
测序得到的原始数据处理后得到有效数据,随后在97%相似水平下进行 OTUs聚类,利用RDP classifier进行OTU物种分类学注释,并在不同物种分类水平下统计每个样本的群落组成。使用R软件(Version 2.15.3)绘制稀释曲线和样本等级聚类曲线;采用mothur软件计算Alpha多样性指数Chao 1、Shannon指数等,并采用Wilxocon秩和检验进行Alpha多样性的组间差异分析;使用Qiime软件(Version 1.9.1)计算观测物种数(Observed-OTUs)、香农指数、Ace指数、文库覆盖率(Goods-coverage)以及谱系多样性指数;使用R软件(Version 2.15.3)绘制PCA图;基于Greengene数据库中OTU的tree和OTU上的基因信息,进行PICRUSt软件进行16S功能预测分析。
2. 结果与分析
2.1 细菌测序结果分析
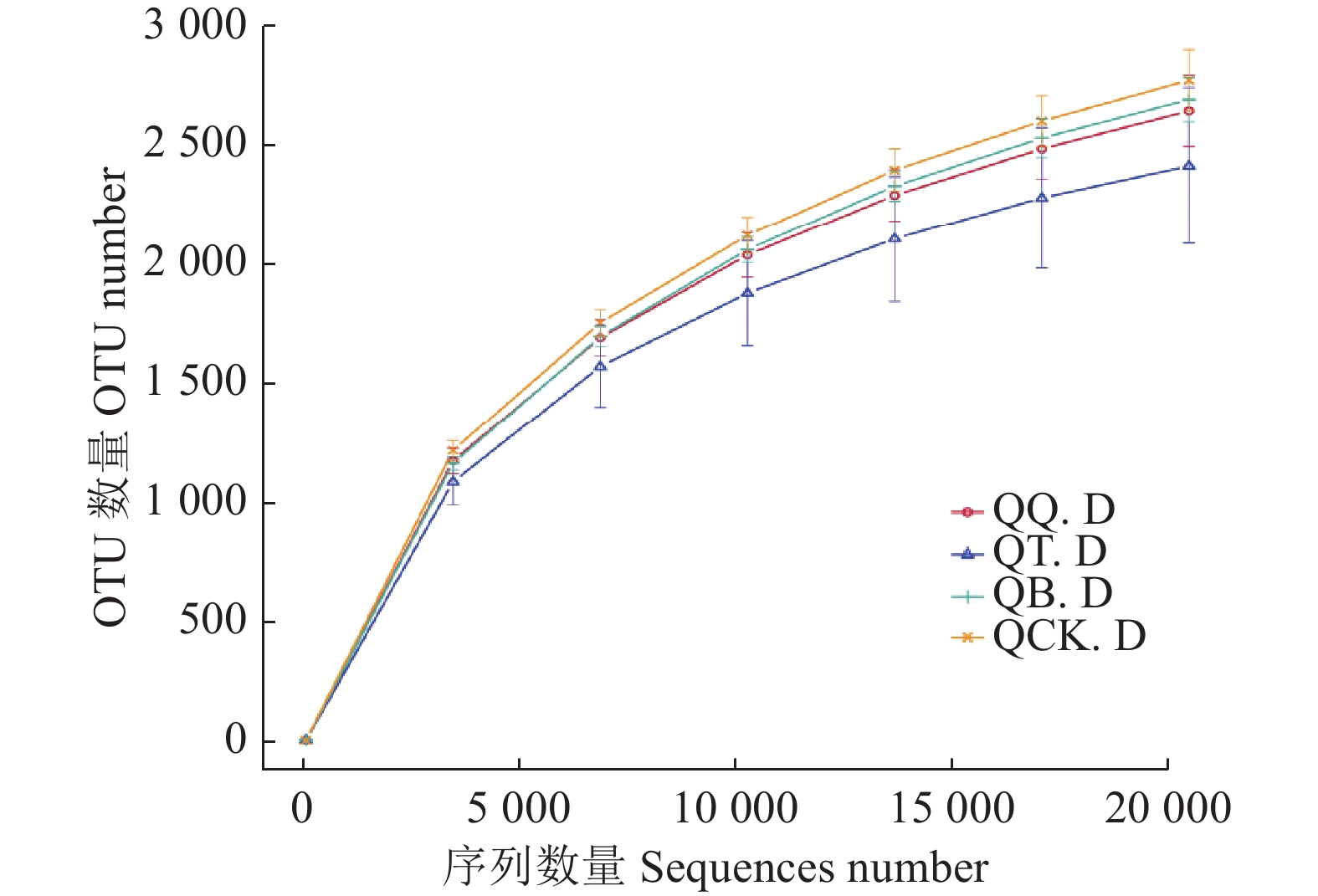
经高通量测序后,12个样本共得到759 513条高质量序列,有效序列531 818条,平均长度378 bp,样本测序深度在0.944~0.974,且每组处理中的3个样本之间测序结果较为一致,测序结果符合预期,较为理想。从图1可以看出,随着测序量的增大,各组稀释曲线趋于平缓,接近饱和,更多的测序量只能够产生少量新的OTUs[11],说明测序数据合理,能覆盖大部分主要细菌。
2.2 细菌群落组成分析
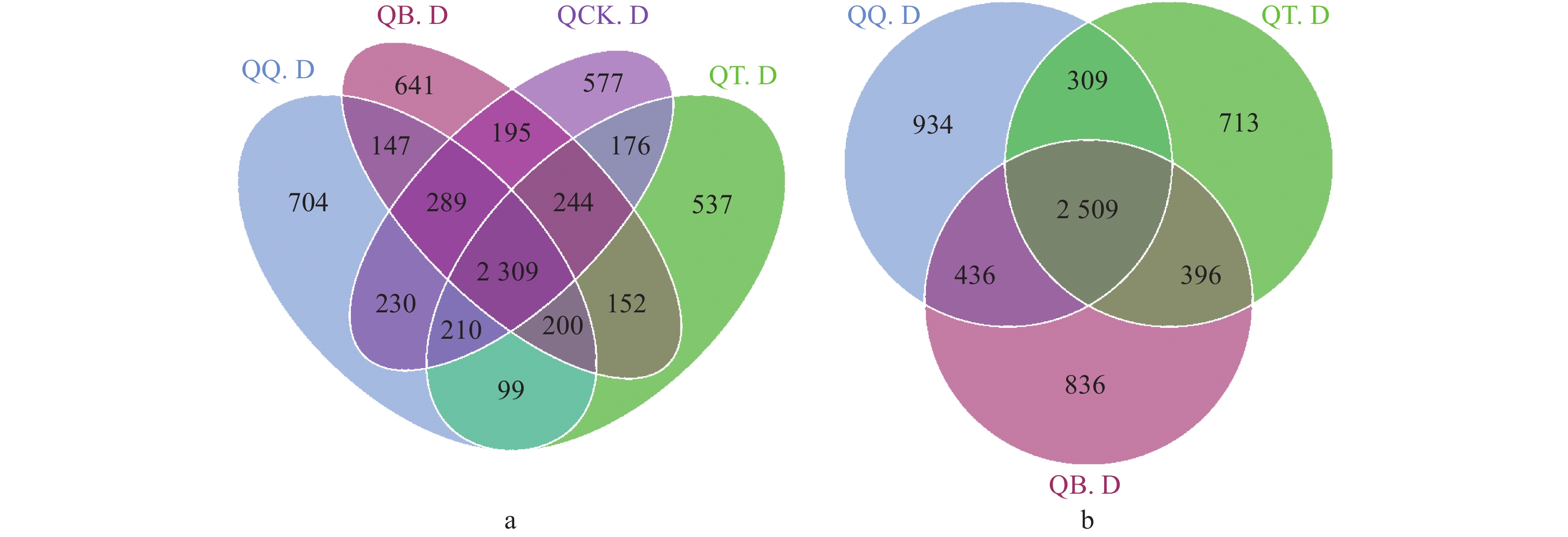
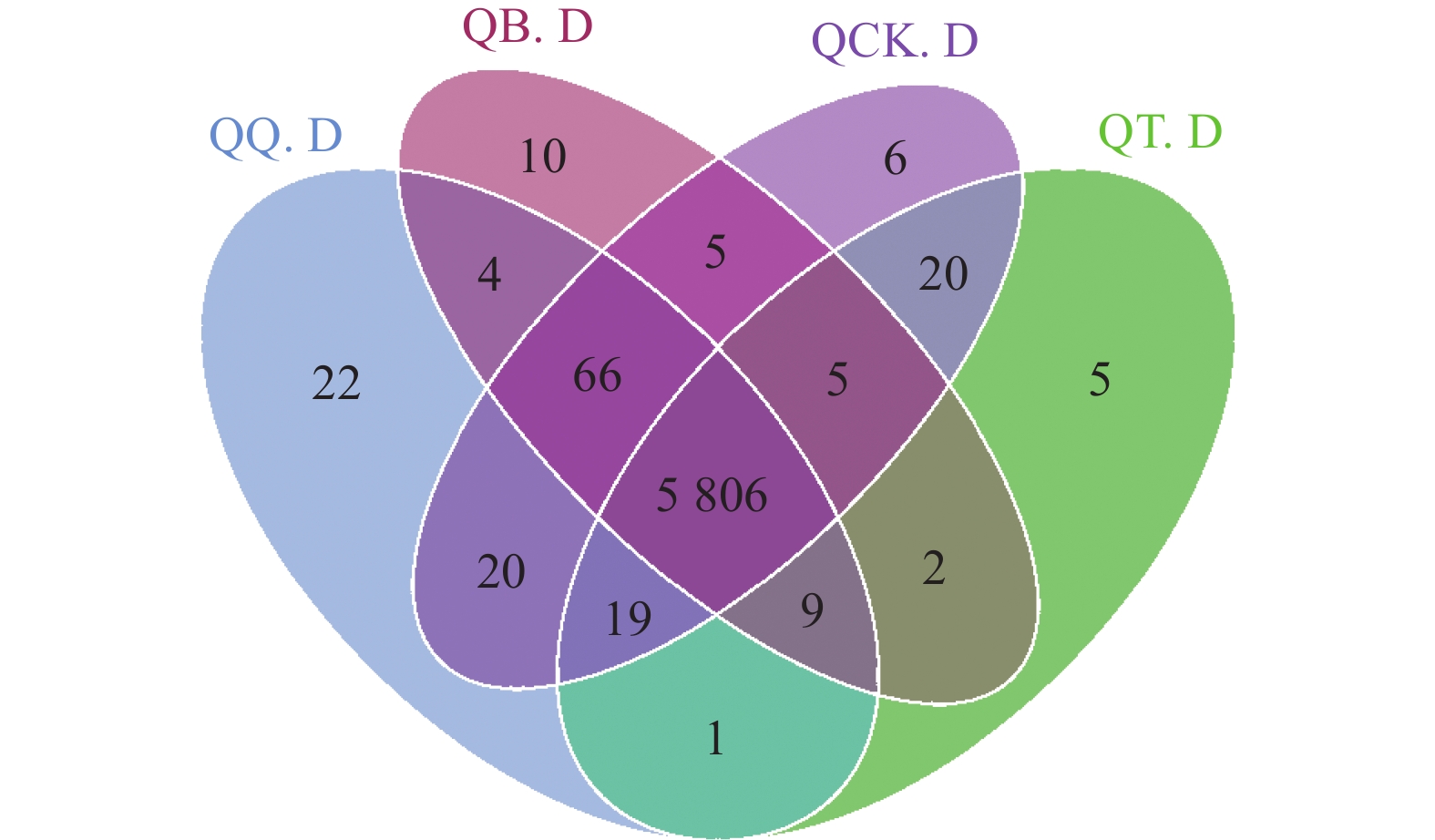
秋茄、白骨壤、桐花树根际土壤样本以及非根际土壤样本共鉴定出细菌39门、55纲、126目、244科、511属。由图2-a可以看出在秋茄根际土壤样本QQ.D、白骨壤根际土壤样本QB.D、桐花树根际土壤样本QT.D和非根际土壤样本QCK.D中有2 309种共有OTU,各有704、641、537和577种特有OTU。各组样本中QCK.D的OTU数目最多,特有OTU数低于QQ.D和QT.D。从图2-b可以看出,秋茄根际土壤样本QQ.D、白骨壤根际土壤样本QB.D、桐花树根际土壤样本QT.D中有2509种共有OTU,特有OTU数QQ.D(934)>QB.D(836)>QT.D(713)。
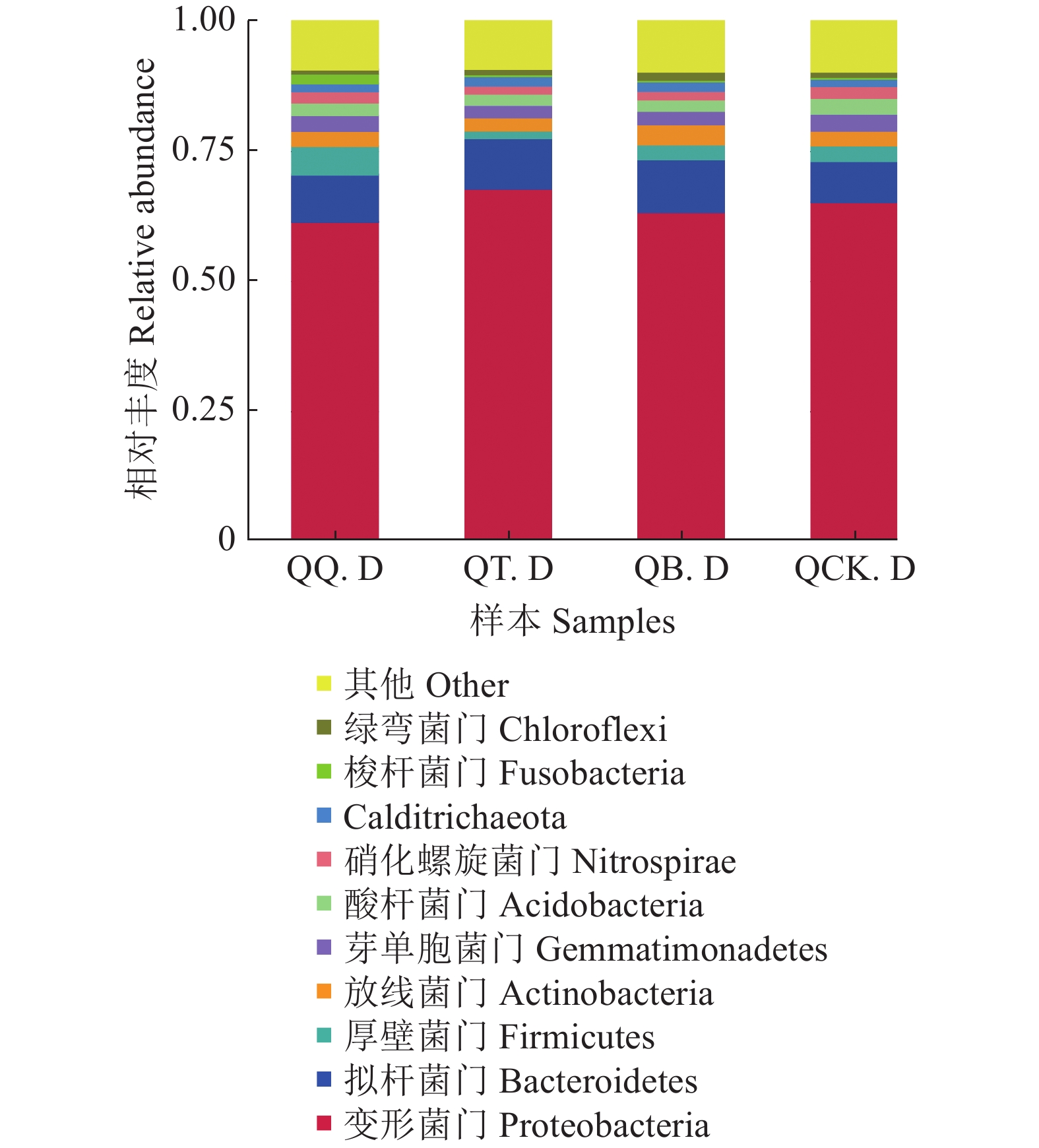
由图3可知,在门水平上,4组样本土壤细菌主要包括变形菌门Proteobacteria、拟杆菌门 Bacteroidetes、厚壁菌门Firmicutes、放线菌门Actinobacteria、芽单胞菌门 Gemmatimonadetes、酸杆菌门Acidobacteria、硝化螺旋菌门Nitrospirae、Calditrichaeota、梭杆菌门 Fusobacteria、绿弯菌门 Chloroflexi。其中变形菌门占比最大,其次是拟杆菌门。4组样本的变形菌门的相对丰度相差不大,但是非根际土样的拟杆菌门的相对丰度低于其他3组根际土样。秋茄根际土壤样本QQ.D中厚壁菌门的相对丰度(5.48%)高于白骨壤根际土壤样本QB.D(2.88%)和非根际土壤样本QCK.D(2.91%),而桐花树根际土壤样本QT.D(1.52%)则较低于其他3组。非根际土壤样本QCK.D的芽单胞菌门、放线菌门和硝化螺旋菌门的相对丰度高于其他3组样本,秋茄根际土壤样本QQ.D的梭杆菌门的相对丰度(1.80%)较其他3组更高。其他门的细菌丰度则相差不大。
由图4可知,在纲水平上,4组样本土壤细菌中占比最大的是变形菌纲,白骨壤根际土壤样本QB.D(39.01%)和桐花树根际土壤样本QT.D(38.69%)的相对丰度基本一致,大于非根际土壤样本QCK.D(32.00%),大于秋茄根际土壤样本QQ.D(26.30%)。其次是γ-变形菌纲,白骨壤根际土壤样本QB.D的相对丰度为16.88%,较其他3组样本低。
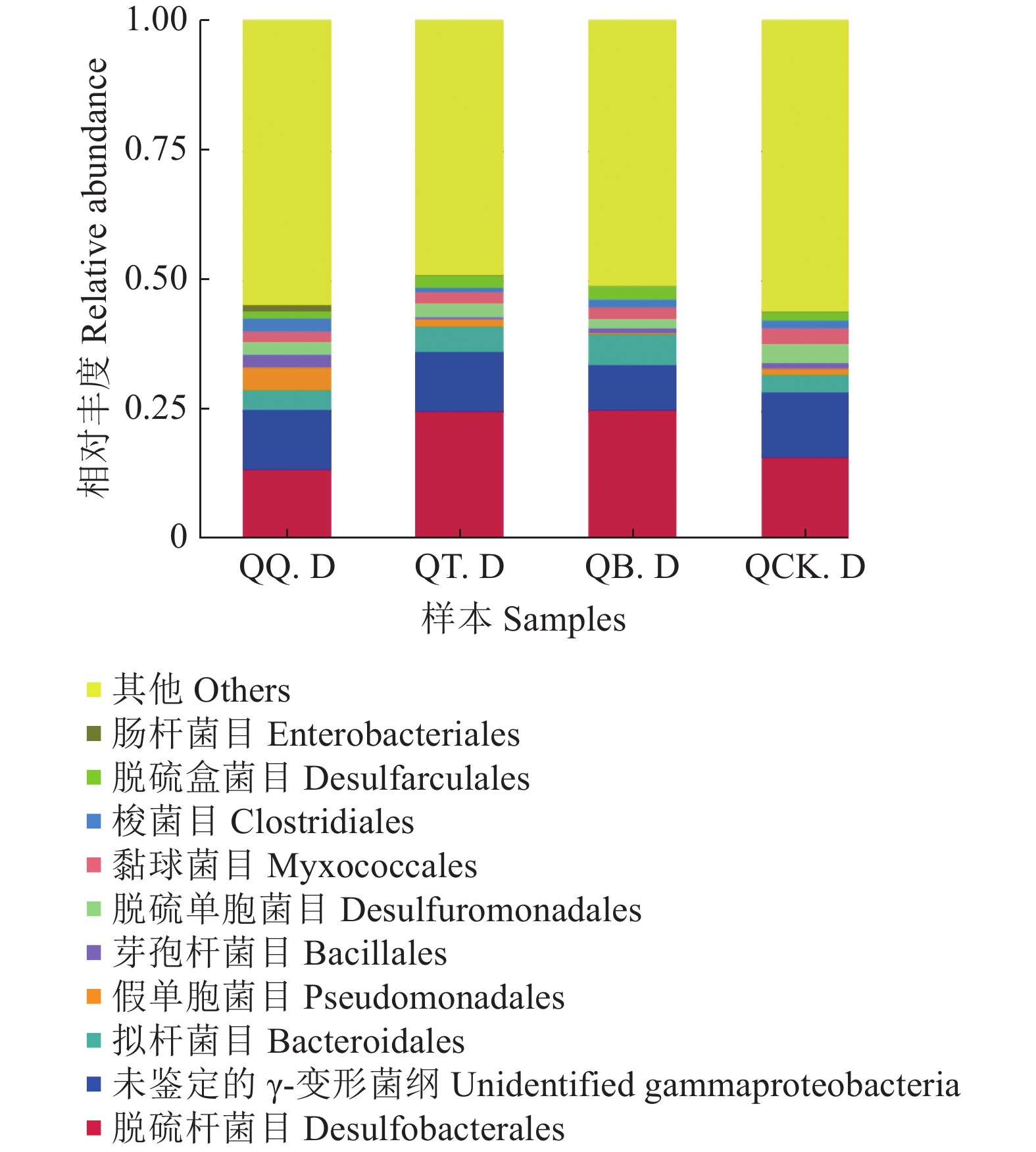
由图5可知,在目水平上,4组样本土壤细菌主要包括脱硫杆菌目Desulfobacterales、拟杆菌目Bacteroidales、假单胞菌目Pseudomonadales、芽孢杆菌目Bacillales、脱硫单胞菌目Desulfuromonadales、黏球菌目Myxococcales、梭菌目Clostridiales、脱硫盒菌目Desulfarculales、肠杆菌目Enterobacteriales。白骨壤根际土壤样本QB.D和桐花树根际土壤样本QT.D在脱硫杆菌目、拟杆菌目、脱硫盒菌目的相对丰度大于秋茄根际土壤样本QQ.D和非根际土壤样本QCK.D;QQ.D在假单胞菌目、芽孢杆菌目、梭菌目、肠杆菌目的相对丰度大于其他3组样本;QCK.D在脱硫单胞菌目、黏球菌目有更高丰富度。
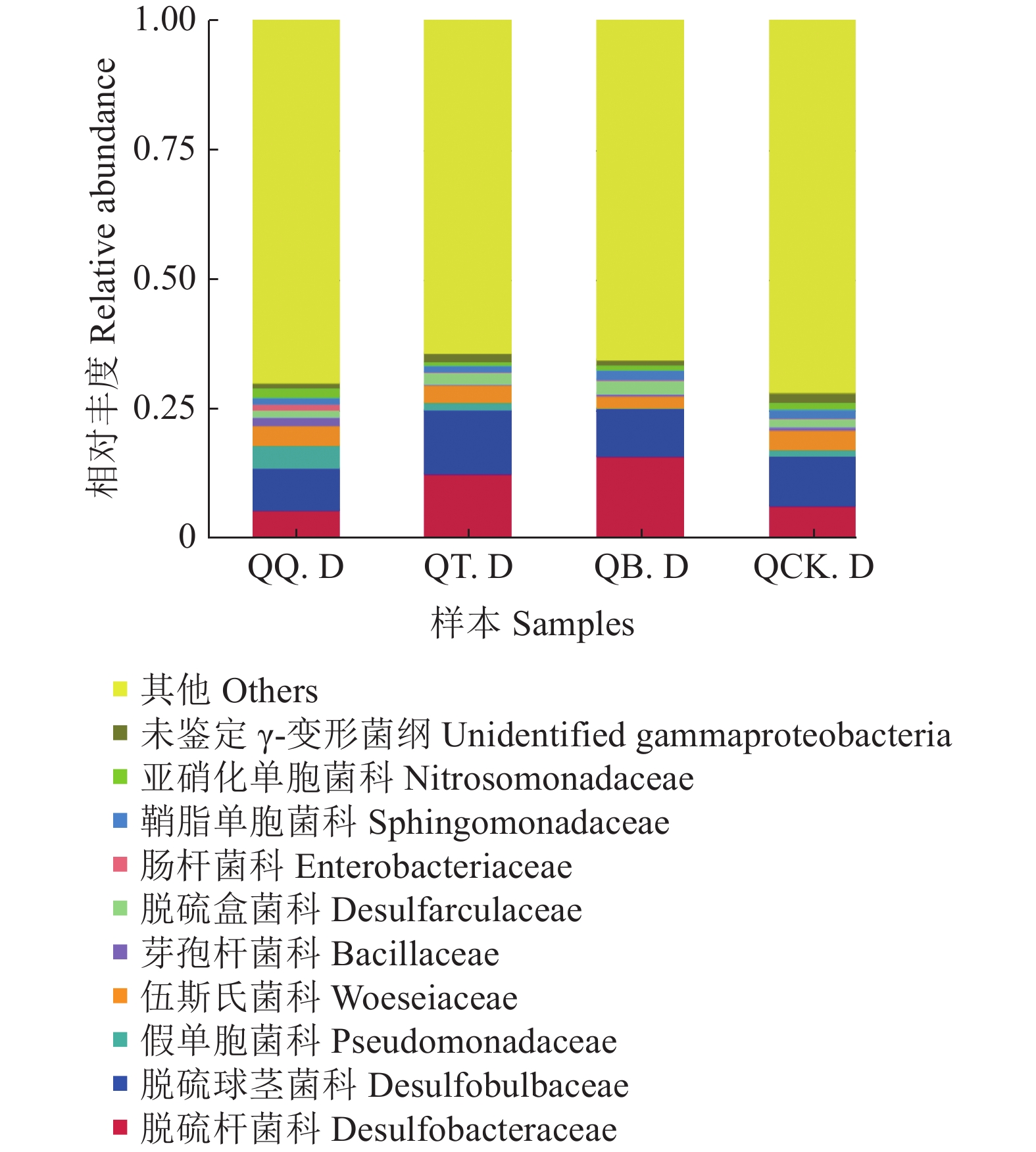
由图6可知,在科水平上,4组样本土壤细菌主要包括脱硫杆菌科Desulfobacteraceae、脱硫球茎菌科Desulfobulbaceae、假单胞菌科Pseudomonadaceae、伍斯氏菌科Woeseiaceae、芽孢杆菌科Bacillaceae、脱硫盒菌科Desulfarculaceae、肠杆菌科Enterobacteriaceae。白骨壤根际土壤样本QB.D和桐花树根际土壤样本QT.D在脱硫杆菌科、脱硫盒菌科的相对丰度大于秋茄根际土壤样本QQ.D和非根际土壤样本QCK.D,在亚硝化单胞菌科的相对丰度低于QQ.D和QCK.D。QQ.D在假单胞菌科、芽孢杆菌科、肠杆菌科上较其他3组更具丰富度,QT.D在脱硫球茎菌科上的丰富度高于其他3组样本。
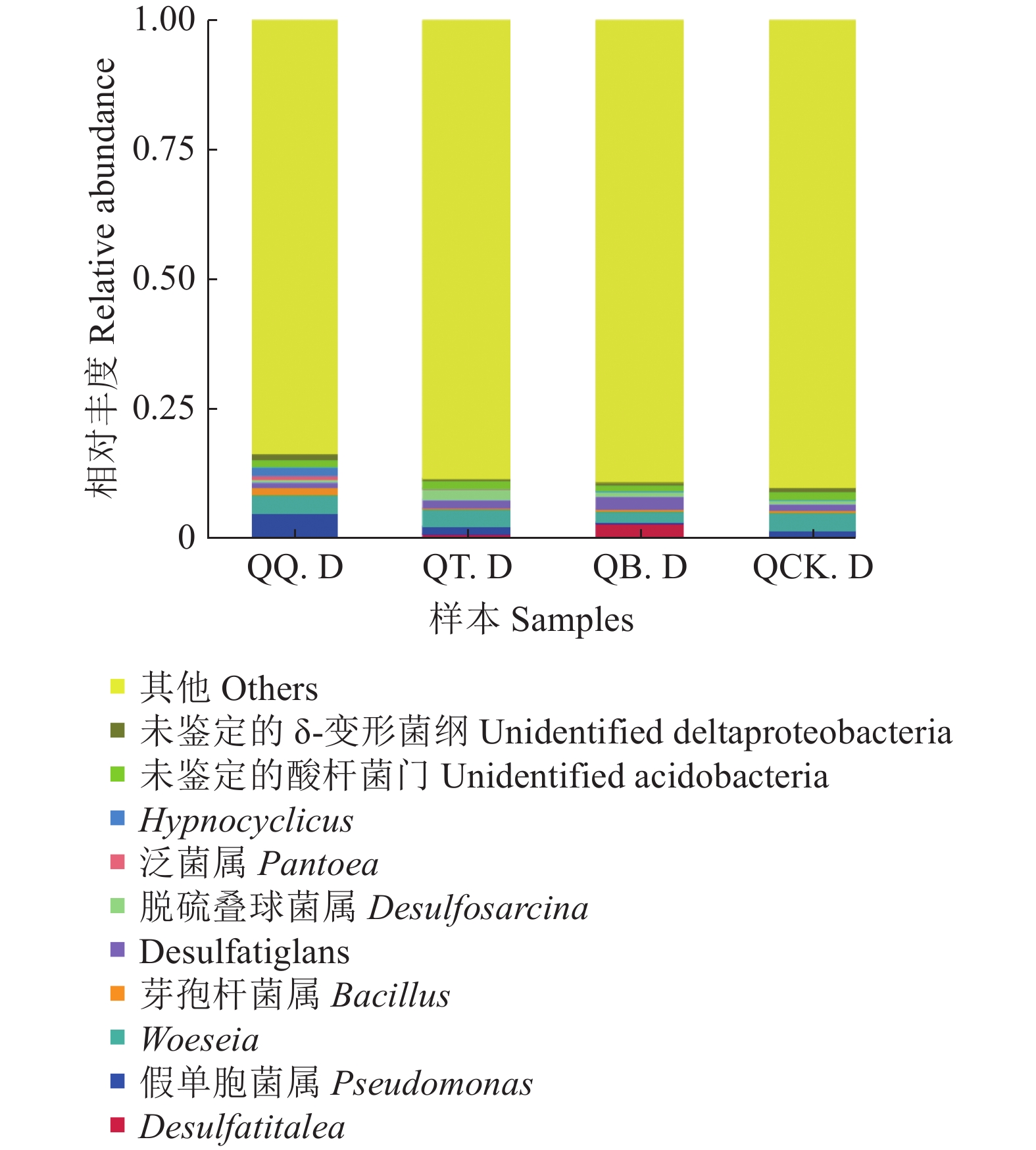
由图7可知,在属水平上,4组样本土壤细菌主要包括Desulfatitalea、假单胞菌属Pseudomonas、Woeseia、芽孢杆菌属Bacillus、Desulfatiglans、脱硫叠球菌属Desulfosarcina、泛菌属Pantoea、Hypnocyclicus。秋茄根际土壤样本QQ.D的细菌在假单胞菌(4.44%)、Hypnocyclicus(1.63%)、芽孢杆菌(1.52%)、泛菌属(0.75%)上更具丰富度,白骨壤根际土壤样本QB.D的细菌分布在Desulfatitalea(3.13%)较其他3组样本更多,桐花树根际土壤样本QT.D的细菌分布在脱硫叠球菌属(1.84%)较其他3组样本更多,非根际土壤样本QCK.D的细菌则在属水平上与其他3组样本比较无更高丰富度。
2.3 细菌群落Alpha多样性分析
各组样本基于OTU数的Alpha多样性指数如表1所示,观测物种数常用于近似表示实际观测到的样本中的OTU数;ACE指数则与观测物种数相关联,反映样本菌群的丰富度[12];谱系多样性指数则反映样本细菌群落的系统发育多样性[13];香农指数表示样本细菌群落的多样性[11];文库覆盖率coverage指数可以显示测序的深度以及覆盖率[14]。以上指数越大,则说明群落多样性越高。
表 1 4组土壤样本的Alpha 多样性指数Table 1. Alpha diversity indices of 4 sets of soils样本
Sample观测物种数
Observed
species丰度
指数
ACE谱系多样性指数
PD whole
tree香农
指数
Shannon文库覆盖率
Coverage/
%QQ.D 2642 3479.534 269.80 9.47 95.8 QB.D 2689 3478.222 268.81 9.46 95.9 QT.D 2412 3019.951 247.34 9.17 96.6 QCK.D 2770 3696.540 271.26 9.67 95.6 非根际土壤样本QCK.D观测到的物种数最多,其次是秋茄根际土壤样本QQ.D和白骨壤根际土壤样本QB.D基本一致,桐花树根际土壤样本QT.D观测到的物种数最少。QCK.D在各个多样性指数上均高于QQ.D、QB.D和QT.D,说明红树植物的非根际土壤细菌群落有着更高的丰富度与多样性,这与郑梅霞等[15]对于土壤细菌群落多样性的研究结果一致。
2.4 细菌群落Beta多样性分析
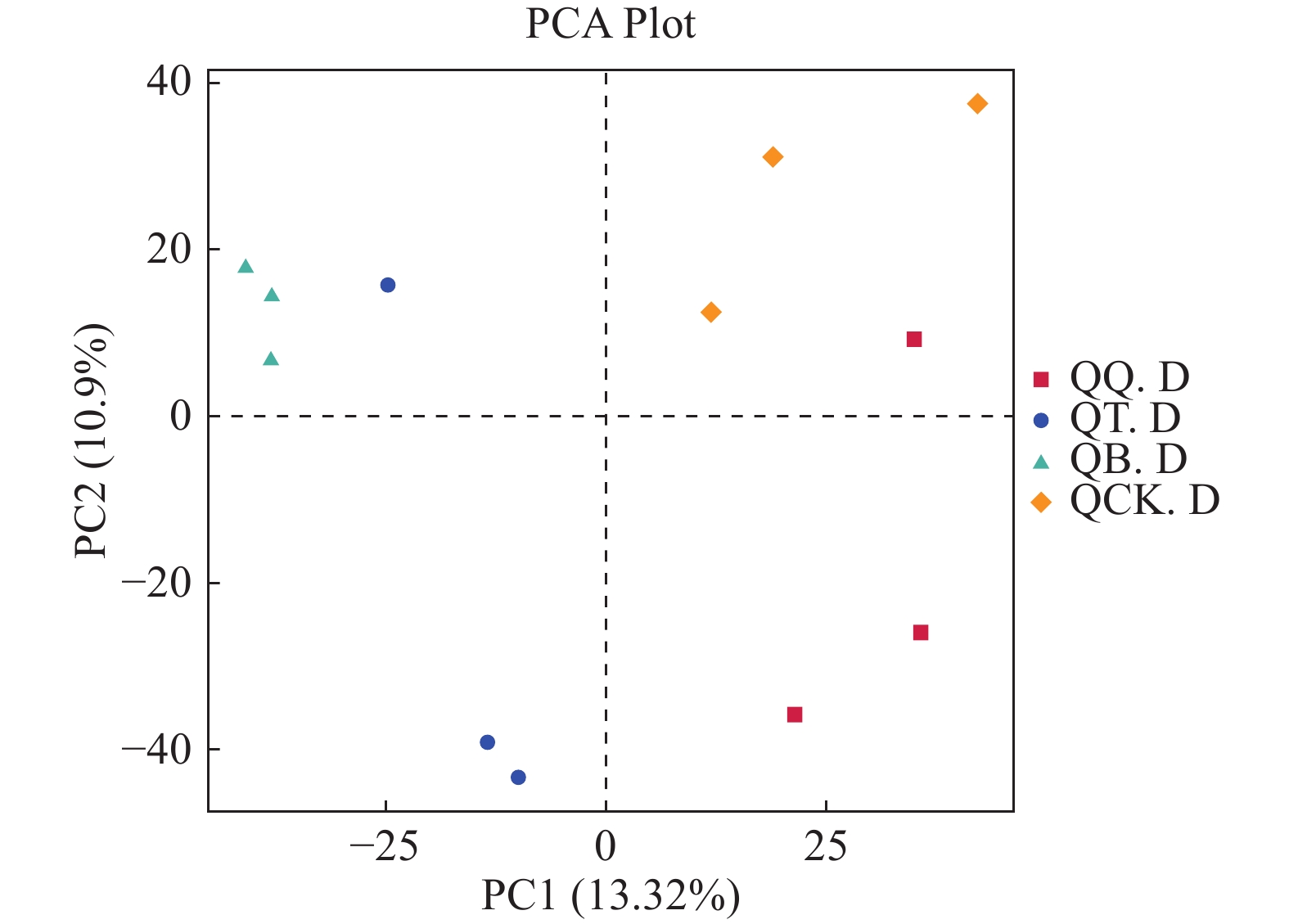
PCA是一种基于欧式距离的应用方差分解,对多维数据进行降维,从而提取出数据中最主要的元素和结构的方法[16]。从图8中可以看出,白骨壤根际土壤样本QB.D与桐花树根际土壤样本QT.D的其中一个样本以及非根际土壤样本QCK.D组与秋茄根际土壤样本QQ.D的其中一个样本在PCA图中的距离较为接近,说明群落组成较接近,从总体上看,4组样本群落组成各不相同,都可单独聚为一类,但是QB.D与QT.D的群落组成,QQ.D与QCK.D的群落组成更为接近。
2.5 细菌功能预测
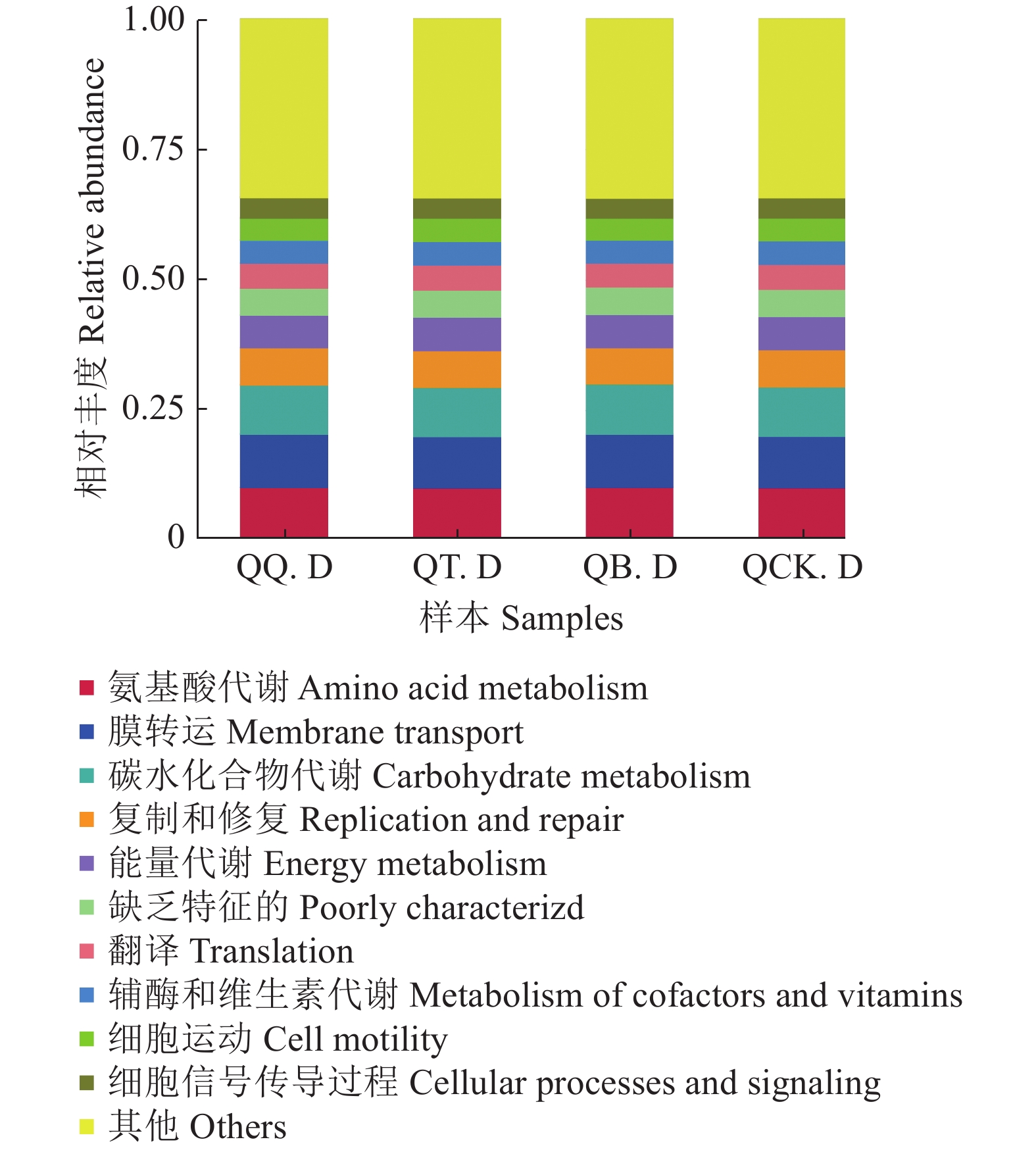
功能预测分析结果如图9,可以看出4组样本的根际细菌群落对应的 COG 类目大致相同,分别为氨基酸代谢、膜转运、碳水化合物代谢、复制和修复、能量代谢、翻译、辅酶和维生素代谢、细胞运动、细胞信号传导过程。差异COG类目如图10,秋茄根际土壤样本QQ.D、白骨壤根际土壤样本QB.D、桐花树根际土壤样本QT.D、非根际土壤样本QCK.D的特有COG类目分别为22、10、5、6,共有5 806 COG类目。
3. 讨论与结论
土壤是营养来源的基础,其环境对红树植物能否存活、繁衍至关关键。土壤微生物的群落结构对土壤质量有着很大的影响[17]。主要表现在土壤微生物与红树植物之间的相互作用,以及在物质的转化和能量流动上起到促进作用,不仅影响着红树植物的生长发育,同时还会为红树林湿地生态系统中的其他生物提供营养来源与生态位[18-19]。
基于土壤微生物群落结构的重要性,本研究对福建泉州湾河口湿地自然保护区内分布在同一生长区的秋茄、白骨壤、桐花树的根际土壤以及它们的非根际土壤进行Illumina高通量测序和分析,发现以下几点规律。
(1)红树植物根际土壤的细菌群落结构丰富度低非根际土壤QCK.D的OTU数目大于其他3组根际土壤样本,同时非根际土壤QCK.D的细菌群落结构在Alpha多样性指数上表现出较3组根际土壤更高的丰富度和多样性。造成这一特征的原因可能是植物根际通过分泌物创造不同的环境,而这种环境对于某些细菌有利,使得它们能够占据竞争优势,从而成为优势种群,同时降低了细菌群落的丰富度。在郑梅霞等[15]、殷萌清等[6]以及Srensen等[20]的研究中也发现了这类现象。
(2)不同红树植物对根际微生物具有选择性4组样本细菌群落的优势菌门分别为变形菌门、拟杆菌门、厚壁菌门、放线菌门和芽单胞菌门。本研究红树植物土壤细菌优势群落与Sadaf等[21]的研究结果类似。但4组土壤样本的第一优势门均为变形菌门,其次是拟杆菌门,但细菌群落分布各有不同。秋茄根际土壤QQ.D在厚壁菌门、梭杆菌门有更高的丰富度,桐花树根际土壤QT.D在变形菌门上的相对丰度高于其他3组,而白骨壤根际土壤QB.D则在拟杆菌门有一定优势。非根际土壤QCK.D在芽单胞菌门较3组根际土壤样本有一定的优势,但拟杆菌门低于3组根际土壤样本。可以看出不同红树植物对根际微生物的影响不同,具有一定选择性,导致细菌群落结构各异,这与一些研究[20,22-23]的结果相似。虽然不同红树植物土壤细菌群落结构各异,但本研究通过PCA分析发现桐花树与白骨壤根际土壤的群落组成更为接近,而秋茄根际土壤却与非根际土壤的细菌群落组成接近。推测白骨壤与桐花树的根系分泌物有一定相似性,起到相同的筛选作用,该方面有待进一步研究。
(3)在湿地生态系统中物质分解与能量来源的功能菌资源丰富在4组土壤样本中,变形菌门的相对丰度均大于60%,占据绝对主导地位。变形菌门多数菌属与氮循环相关,有研究报道称变形菌门为湿地环境第一优势门[24],与本研究结果相同。但是红树植物不同于其他植物,土壤细菌在硝化螺旋菌门、Calditrichaeota、梭杆菌门有一定丰富度。硝化螺旋菌门多为功能菌,能够催化亚硝酸盐的氧化过程[25];Calditrichaeota为新发现的细菌门,多发现于海洋细菌群落,目前发现的该门下细菌具有降解蛋白质基质的能力[26];在科水平上,秋茄根际土壤样本QQ.D在假单胞菌科、芽孢杆菌科、肠杆菌科较其他3组样本相对丰度更高,假单胞菌能分解葡萄糖,同时与硝酸盐还原有关;芽孢杆菌则能够防治病虫害、促进植物生长[27];肠杆菌科与代谢、发酵方面功能相关,并参与还原硝酸盐。白骨壤和桐花树的根际土壤QB.D、QT.D则在脱硫杆菌科、脱硫球茎菌科、脱硫盒菌科这些与硫循环有关的菌群上具更高的丰富度。猜测与红树植物的生长过程有关,红树植物会从土壤中吸收硫酸根离子,并转化为硫化物的形式累积在体内[23]。
本研究对秋茄、白骨壤、桐花树根际土壤以及它们的非根际土壤细菌群落组成进行了研究,比较了不同水平上的相对丰度,分析了优势菌群的相关作用,有助于研究土壤微生物群落结构对红树植物的生长促进作用,为红树林湿地生态系统的保护与修复奠定基础。有关红树植物与土壤菌群的相互作用及其调控机制,有待进一步研究。
-
表 1 4组土壤样本的Alpha 多样性指数
Table 1 Alpha diversity indices of 4 sets of soils
样本
Sample观测物种数
Observed
species丰度
指数
ACE谱系多样性指数
PD whole
tree香农
指数
Shannon文库覆盖率
Coverage/
%QQ.D 2642 3479.534 269.80 9.47 95.8 QB.D 2689 3478.222 268.81 9.46 95.9 QT.D 2412 3019.951 247.34 9.17 96.6 QCK.D 2770 3696.540 271.26 9.67 95.6 -
[1] 叶锦成, 陈毅青, 高琳, 等. 红树植物拟海桑及其亲本的根际细菌群落特征分析[J/OL]. 热带海洋学报: 1 − 15. (2021-10-18). [2022-01-24]. https://kns.cnki.net/kcms/detail/44.1500.p.20211014.1838.006.html. YE J C, CHEN Y Q, GAO L, et al. Analysis of rhizosphere bacterial community characteristics of mangrove plant Sonneratia × gulngai and its parents [J/OL]. Journal of Tropical Oceanography: 1 − 15. (2021-10-18). [2022-01-24]. https://kns.cnki.net/kcms/detail/44.1500.p.20211014.1838.006.html. (in Chinese)
[2] LUIS P, SAINT-GENIS G, VALLON L, et al. Contrasted ecological niches shape fungal and prokaryotic community structure in mangroves sediments [J]. Environmental Microbiology, 2019, 21(4): 1407−1424. DOI: 10.1111/1462-2920.14571
[3] 陈孟立, 曾全超, 黄懿梅, 等. 黄土丘陵区退耕还林还草对土壤细菌群落结构的影响 [J]. 环境科学, 2018, 39(4):1824−1832. CHEN M L, ZENG Q C, HUANG Y M, et al. Effects of the farmland-to-forest/grassland conversion program on the soil bacterial community in the loess hilly region [J]. Environmental Science, 2018, 39(4): 1824−1832.(in Chinese)
[4] 蒋云霞, 郑天凌, 田蕴. 红树林土壤微生物的研究: 过去、现在、未来 [J]. 微生物学报, 2006, 46(5):848−851. DOI: 10.3321/j.issn:0001-6209.2006.05.035 JIANG Y X, ZHENG T L, TIAN Y. Research on mangrove soil microorganisms: Past, present and future [J]. Acta Microbiologica Sinica, 2006, 46(5): 848−851.(in Chinese) DOI: 10.3321/j.issn:0001-6209.2006.05.035
[5] DIAS A C F, ANDREOTE F D, RIGONATO J, et al. The bacterial diversity in a Brazilian non-disturbed mangrove sediment [J]. Antonie Van Leeuwenhoek, 2010, 98(4): 541−551. DOI: 10.1007/s10482-010-9471-z
[6] 殷萌清, 冯建祥, 黄小芳, 等. 天然及人工红树林土壤微生物群落结构分析 [J]. 生态科学, 2017, 36(5):1−10. YIN M Q, FENG J X, HUANG X F, et al. Soil microbial community structure in natural and transplanted mangrove (Kandelia obovata) forests [J]. Ecological Science, 2017, 36(5): 1−10.(in Chinese)
[7] 蓝亦琦, 吴则焰, 陈军, 等. 淡水驯化条件下红树植物根际土壤微生物群落结构特征 [J]. 福建农业学报, 2019, 34(1):83−94. LAN Y Q, WU Z Y, CHEN J, et al. Characteristics of microbial community in rhizosphere soil of mangrove forest under freshwater stress [J]. Fujian Journal of Agricultural Sciences, 2019, 34(1): 83−94.(in Chinese)
[8] 曹启民, 郑康振, 陈耿, 等. 红树林生态系统微生物学研究进展 [J]. 生态环境, 2008, 17(2):839−845. CAO Q M, ZHENG K Z, CHEN G, et al. A review of studies on microbiology of mangrove ecosystems [J]. Ecology and Environment, 2008, 17(2): 839−845.(in Chinese)
[9] DEGNAN P H, OCHMAN H. Illumina-based analysis of microbial community diversity [J]. The ISME Journal, 2012, 6(1): 183−194. DOI: 10.1038/ismej.2011.74
[10] 黎烨, 雷少楠, 程志强, 等. 五种植被覆盖下红壤的细菌群落结构组成及其特征 [J]. 福建农林大学学报(自然科学版), 2020, 49(2):231−239. LI Y, LEI S N, CHENG Z Q, et al. Microbial community structure and characteristics of red soil microbiome under 5 types of vegetation coverages [J]. Journal of Fujian Agriculture and Forestry University (Natural Science Edition), 2020, 49(2): 231−239.(in Chinese)
[11] 秦宇蒙, 周笑犁, 管庆林, 等. 基于高通量测序分析番茄自然发酵过程中的真菌多样性 [J]. 福建农业学报, 2021, 36(9):1110−1118. QIN Y M, ZHOU X L, GUAN Q L, et al. Fungal diversity in natural tomato fermentation as shown by high-throughput sequencing [J]. Fujian Journal of Agricultural Sciences, 2021, 36(9): 1110−1118.(in Chinese)
[12] BAI L, CUI J Q, JIE W G, et al. Analysis of the community compositions of rhizosphere fungi in soybeans continuous cropping fields [J]. Microbiological Research, 2015, 180: 49−56. DOI: 10.1016/j.micres.2015.07.007
[13] 刘兰英, 黄薇, 吕新, 等. 长期施用沼液对槟榔芋根际土壤真菌群落多样性的影响 [J]. 福建农业学报, 2020, 35(9):1034−1042. LIU L Y, HUANG W, LYU X, et al. Effect of long-term biogas slurry application on fungal community in rhizosphere soil at Areca taro fields [J]. Fujian Journal of Agricultural Sciences, 2020, 35(9): 1034−1042.(in Chinese)
[14] 宋相宇, 李鸣, 王虎虎, 等. 高通量测序分析白切鸡菌群多样性 [J]. 食品科学, 2020, 41(17):246−252. DOI: 10.7506/spkx1002-6630-20190822-230 SONG X Y, LI M, WANG H H, et al. Analysis of bacterial community diversity of soft-boiled chicken by high-throughput sequencing [J]. Food Science, 2020, 41(17): 246−252.(in Chinese) DOI: 10.7506/spkx1002-6630-20190822-230
[15] 郑梅霞, 陈宏, 朱育菁, 等. 七叶一枝花根际与非根际土壤细菌群落多样性 [J]. 福建农业学报, 2020, 35(12):1357−1367. ZHENG M X, CHEN H, ZHU Y J, et al. Microbial diversity in rhizosphere and non-rhizosphere soils of Paris Polyphylla var. chinensis plants [J]. Fujian Journal of Agricultural Sciences, 2020, 35(12): 1357−1367.(in Chinese)
[16] AVERSHINA E, FRISLI T, RUDI K. De novo semi-alignment of 16S rRNA gene sequences for deep phylogenetic characterization of next generation sequencing data [J]. Microbes and Environments, 2013, 28(2): 211−216. DOI: 10.1264/jsme2.ME12157
[17] 焦晓丹, 吴凤芝. 土壤微生物多样性研究方法的进展 [J]. 土壤通报, 2004, 35(6):789−792. DOI: 10.3321/j.issn:0564-3945.2004.06.026 JIAO X D, WU F Z. Progress of the methods for studying soil microbial diversity [J]. Chinese Journal of Soil Science, 2004, 35(6): 789−792.(in Chinese) DOI: 10.3321/j.issn:0564-3945.2004.06.026
[18] BHARATHKUMAR S, PAUL D. Microbial diversity of culturable heterotrophs in the rhizosphere of salt marsh grass, porteresia coarctata (tateoka) in a mangrove ecosystem [J]. Journal of Basic Microbiology, 2008, 48(1): 10−15. DOI: 10.1002/jobm.200700282
[19] HIRSCH P R, MAUCHLINE T M, CLARK I M. Culture-independent molecular techniques for soil microbial ecology [J]. Soil Biology & Biochemistry, 2010, 42(6): 878−887.
[20] SöRENSEN J. The rhizosphere as a habitat for soil microorganisms[M]//van ELSAS J D, TREVORS J T, WELLINGTON E M H. Modern Soil Microbiology. New York, USA: Marcel Dekker, 1997: 21-45
[21] SADAF K, ANIRBAN B, APPA R P. Difficult-to-culture bacteria in the rhizosphere: The underexplored signature microbial groups [J]. Pedosphere, 2022, 32(1): 75−89. DOI: 10.1016/S1002-0160(21)60062-0
[22] 武钰坤, 刘永军, 司英明, 等. 人工湿地不同植物根际微生物群落多样性比较研究 [J]. 生态科学, 2012, 31(3):318−323. DOI: 10.3969/j.issn.1008-8873.2012.03.016 WU Y K, LIU Y J, SI Y M, et al. Study on rhizosphere microbial community diversity of different plants grownin the constructed wetland [J]. Ecological Science, 2012, 31(3): 318−323.(in Chinese) DOI: 10.3969/j.issn.1008-8873.2012.03.016
[23] 王岳坤, 洪葵. 红树林土壤细菌群落16SrDNAV3片段PCR产物的DGGE分析 [J]. 微生物学报, 2005, 45(2):198−201. WANG Y K, HONG K. Mangrove soil community analysis using DGGE of 16S rDNA V3 fragment polymerase chain reaction products [J]. Acta Microbiologica Sinica, 2005, 45(2): 198−201.(in Chinese)
[24] 李振灵, 丁彦礼, 白少元, 等. 潜流人工湿地基质结构与微生物群落特征的相关性 [J]. 环境科学, 2017, 38(9):3713−3720. LI Z L, DING Y L, BAI S Y, et al. Correlations between substrate structure and microbial community in subsurface flow constructed wetlands [J]. Environmental Science, 2017, 38(9): 3713−3720.(in Chinese)
[25] 洪义国, 焦黎静, 吴佳鹏, 等. 海洋亚硝酸盐氧化细菌的多样性分布及其生态功能研究进展 [J]. 热带海洋学报, 2021, 40(2):139−146. DOI: 10.11978/2020043 HONG Y G, JIAO L J, WU J P, et al. Progress on the community distribution and ecological functions of nitrite-oxidizing bacteria [J]. Journal of Tropical Oceanography, 2021, 40(2): 139−146.(in Chinese) DOI: 10.11978/2020043
[26] 张雪, 乔雪姣, 苏佳, 等. 垃圾渗滤液处理厂活性污泥微生物种群结构和功能分析 [J]. 北京大学学报(自然科学版), 2021, 57(5):927−937. ZHANG X, QIAO X J, SU J, et al. Microbial structure and function of activated sludge in landfill leachate treatment plant [J]. Acta Scientiarum Naturalium Universitatis Pekinensis, 2021, 57(5): 927−937.(in Chinese)
[27] 葛晓颖, 孙志刚, 李涛, 等. 设施番茄连作障碍与土壤芽孢杆菌和假单胞菌及微生物群落的关系分析 [J]. 农业环境科学学报, 2016, 35(3):514−523. DOI: 10.11654/jaes.2016.03.015 GE X Y, SUN Z G, LI T, et al. Soil Pseudomonas spp., Bacillus spp., and microbial communities under tomato continuous cropping in greenhouse production [J]. Journal of Agro-Environment Science, 2016, 35(3): 514−523.(in Chinese) DOI: 10.11654/jaes.2016.03.015
-
期刊类型引用(6)
1. 曾志浩,王思凡,林文波,郭萍萍,袁宗胜,刘芳. 油松林下种植牡丹土壤真菌群落结构及多样性. 福建林业科技. 2024(02): 1-7+18 .  百度学术
百度学术
2. 李骥,王芸,朱启聪,李国壕,张颖,蔺红苹,王锂韫,李敏,唐蜀昆. 濒危红树杂交种海南海桑的杂种劣势与其根际微生物的关联分析. 微生物学报. 2024(08): 2823-2843 .  百度学术
百度学术
3. 吴杨,卢秋云,姜学峰,李永红,彭俊文. 不同套作模式对魔芋根际土壤微生物群落的影响. 南方农业. 2024(11): 19-22 .  百度学术
百度学术
4. 刘敏,甘禧霖,黄小峰,王洋,吴昊,孙睿. 红树林沉积物中可培养细菌的分离及其系统进化分析. 海南热带海洋学院学报. 2023(02): 25-31+83 .  百度学术
百度学术
5. 郑智胜,黄卫红,陈小尘,黄兆斌,薛喜枚,陈锦江,张秋芳. 泉州湾桐花树和老鼠簕根际土壤细菌和真菌群落特征比较. 应用海洋学学报. 2023(04): 686-697 .  百度学术
百度学术
6. 阳长洪,卫婷,李慧君,黄枫城,吴炜龙,杨贵琼,甄珍,蔺中. 菲在红树林不同根际距离中的降解及微生物群落结构变化. 南方农业学报. 2023(12): 3538-3549 .  百度学术
百度学术
其他类型引用(1)




 下载:
下载: