Identification and Functions of ACY1 Family in Polygonatum cyrtonema
-
摘要:目的 筛选鉴定多花黄精中氨基酰化酶(aminoacylase 1,ACY1)基因,为多花黄精抗逆基因功能研究奠定基础。方法 基于多花黄精转录组数据,对多花黄精氨基酰化酶(PcACY1)基因家族进行鉴定,分析基本理化性质、基因结构、保守基序、进化关系等,并采用qRT-PCR验证ACY1家族成员在不同组织器官中和盐胁迫处理下的表达模式。结果 多花黄精ACY1家族有2个成员,均为亲水蛋白,具有信号肽,无跨膜结构,亚细胞定位预测结果显示2个成员均定位于细胞质中。与植物其他物种ACY1构建进化树发现,其进化特性可分为2类。植物ACY1启动子顺式作用元件预测结果显示,ACY1存在多种激素、胁迫和生长发育相关的响应元件。qRT-PCR分析结果显示,PcACY1成员在多花黄精不同组织器官中的表达水平差异较大且受盐胁迫诱导。结论 PcACY1在多花黄精生长发育和抵御逆境过程中可能发挥重要作用,这为进一步研究多花黄精ACY1的功能及遗传改良等奠定基础。Abstract:Objective Family of aminoacylase 1 genes (ACY1) in Polygonatum cyrtonema Hua was identified, and associated resistance functions analyzed.Methods Based on the transcriptome data, the family of P. cyrtonema Hua aminoacylase genes (PcACY1) was identified. Physicochemical properties, structure, conserved motifs, and evolutionary relationships of the members were analyzed. Expressions of PcACY1 in tissues/organs and under salt stress were detected by qRT-PCR.Results The PcACY1 family had two members located in the cytoplasm, both of which were hydrophilic proteins with signal peptides and no transmembrane structure. As shown in the phylogenetic tree, they exhibited two categories of evolutionary characteristics. The predicted cis-acting elements of the plant ACY1 promoter indicated the presence of multiple hormone-, stress-, and growth/development-related functions. PcACY1 displayed significantly differentiated expressions in different tissues and organs which could be induced by salt stress.Conclusion PcACY1 appeared to play an important role in the growth and development as well as stress resistance of P. cyrtonema .
-
Keywords:
- Polygonatum cyrtonema Hua /
- ACY1 /
- gene family identification /
- expression analysis
-
0. 引言
【研究意义】氨基酰化酶aminoacylase 1(ACY1,N-acyl-L-amino-acid amidohydrolase)是一种在胞内蛋白质降解时脱酰化从而影响蛋白功能和稳定性的金属酶[1],在植物的生长发育和逆境胁迫响应等方面也有着重要的作用。ACY1是一种能与锌指结构域相结合的同型二聚体胞质酶[2],具有Zn2+离子的锌结合结构域和促进锌结合结构域二聚的主域,其活性位点位于两个锌结合域之间[3, 4]。在真核细胞中,有超过50%的胞内蛋白在 N 端发生酰基化[5],这是一种影响蛋白功能和稳定性的翻译后修饰[6]。ACY1 作为一种金属酶,它能从氨基酸中去除与氨基相连的酰基[7],并可能在氧化应激反应中发挥着调节作用[8]。多花黄精(Polygonatum cyrtonema Hua)是百合科黄精属多年生草本植物。根状茎为主要的入药部位,《中国药典》规定作为黄精药用的有3种:黄精、多花黄精和滇黄精,是中药材黄精的原生药,且为药食同源的传统大宗药材,作药用时统称黄精,有补气养阴,健脾,润肺,益肾等功效[9, 10]。因此对多花黄精ACY1进行研究具有重要意义。【前人研究进展】氨基酰化酶最早是在动物的肝脏和肾脏中发现的[11],广泛存在于多种哺乳动物组织中,已被报道参与多种人类疾病的发生[12-15]。目前有关ACY1的研究主要集中在人类、动物[16, 17]和微生物[18, 19],在植物中的研究少有报道,目前仅有两篇研究显示植物转基因ACY1会影响烟草生长发育及抗逆性[20, 21]。【本研究切入点】关于多花黄精在分子生物学方面的研究相对较少,而ACY1基因在动物、微生物中广泛存在,且作用重要,关于多花黄精ACY1基因的研究有待深入进行。【拟解决的关键问题】基于转录组测序,对多花黄精ACY1基因家族进行鉴定,并对其表达模式进行分析,以期为多花黄精ACY1的功能研究及其在多花黄精生长和抗逆中的作用研究提供参考。
1. 材料与方法
1.1 试验材料
多花黄精转录组数据通过福建省邵武市大竹镇的野生多花黄精测序获得(另文发表)。经法国图卢兹综合科学研究所徐涵教授鉴定为百合科黄精属多花黄精Polygonatum cyrtonema Hua。qRT-PCR所用多花黄精组培苗由福建农林大学园艺植物生物工程研究所提供,收集组培苗5个组织部位(根、茎秆、根状茎、叶和芽),以及不同NaCl浓度处理24 h后的组培苗,对上述材料进行总RNA的提取并于–80 ℃保存。
1.2 试验方法
1.2.1 多花黄精ACY1基因家族成员鉴定及蛋白理化性质分析
使用多花黄精全长转录组中蛋白序列构建本地 blast 数据库,以拟南芥ACY1蛋白序列为种子序列进行 blast(1e-5)搜索;在 Pfam 数据库 (http://pfam.xfam.org/)下载M20_dimer (PF07687)和Peptidase_M20(PF01546)的隐马氏模型,利用 hmmer 3.0 软件(1e-5)进行搜索;去除两种方法的重复序列,使用Interpro(https://www.ebi.ac.uk/interpro/)进行结构域分析,删除不含N-acyl_aa_amidohydrolase结构域的序列,最终获得2个候选基因。运用在线网站对PcACY1进行蛋白质基本理化性质(ExPASy, https://web.expasy.org/protparam/)、信号肽(SignalP 4.1 Server, http://www.cbs.dtu.dk/services/SignalP-4.1/)、跨膜结构(TMHMM Server v.2.0, http://www.cbs.dtu.dk/services/TMHMM/)和亚细胞定位(WoLF PSORT, https://wolfpsort.hgc.jp/)预测分析。
1.2.2 多花黄精ACY1保守结构域分析、二级结构和三级结构预测
通过蛋白质分类家族网站InterPro(http://www.ebi.ac.uk/interpro/scan.html/)对PcACY1家族进行主要结构域分析,将PcACY1蛋白序列导入InterPro进行处理。利用在线软件SWISS-MODEL(https://swissmodel.expasy.org/)预测蛋白质的三维空间结构。利用在线软件NetPhos对PcACY1蛋白质进行磷酸化位点和糖基化位点预测。
1.2.3 植物ACY1家族的进化特性
根据NCBI(https://www.ncbi.nlm.nih.gov/)和植物基因组数据库Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)挑选32个植物物种的65条蛋白序列,并进行植物学分类,了解其在不同物种中的分布规律,并统计ACY1家族成员的物种分布情况。植物ACY1家族成员的进化树构建:利用MEGA7.0软 件,采用邻接法(Neighbor-joining,NJ)对ACY1家族包括多花黄精在内共67个序列进行发育进化树构建,自展法系数(Bootstrap)设置 1000次重复检验,最后采用 iTOL(https://itol.embl.de/)在线软件对进化树进行美化。
1.2.4 基因结构和保守基序分析
从植物基因组数据库Phytozome获得上述65条序列的CDS序列和gDNA序列,采用GSDS2.0在线网站(http://gsds.cbi.pku.edu.cn/)进行基因结构特征分析;使用网站MEME(http://meme-suite.org/tools/meme)和Web CD-Search Tool(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)查找分析其保守基序,使用TBtools[22]进行图像绘制。
1.2.5 植物ACY1家族启动子顺式作用元件分析
因多花黄精暂无基因组,为分析植物ACY1家族成员启动子区域包含的顺式作用元件,对65个ACY1基因起始密码子上游的2000 bp进行分析,序列来自NCBI数据库,这些序列通过PlantCARE (http://bioinformatics.psb.ugent.be/)数据库预测转录因子可识别并特异性结合的顺式作用元件,并进行详细分析与数目统计。
1.2.6 多花黄精ACY1盐胁迫及不同器官的转录组基因表达分析
利用多花组织部位和盐胁迫的转录组数据分析PcACY1的表达特征。采用对数log计算法对PcACY1家族基因的表达数据进行分析,并利用Tbtools软件绘制表达热图。
1.2.7 基因非生物胁迫表达数据挖掘
基因表达谱的数据从BAR Toronto eFP浏览器(http://bar.utoronto)中检索获得了以下物种的数据: Arabidopsis thaliana和Oryza sativa。这一选择基于包含多个ACY1同系物的物种的非生物胁迫数据可用性,并在BAR Toronto eFP浏览器中有表示。通过电子荧光象形图(eFP)获得的数据以FPKM归一化表示,在所有研究中,样本都进行了3次重复分析,而该数据库中只有平均值,因此使用该数据生成热图。
1.2.8 qRT-PCR分析
使用Hifair®1st Strand cDNA Synthesis kit试剂盒对提取的不同多花黄精材料总RNA进行cDNA合成。利用在线网站Primer3 Input(http://primer3.ut.ee/)和DNAMAN软件设计多花黄精ACY1家族基因的特异性引物(表1)并送至福州生工生物技术有限公司进行合成。以Actin为内参基因在Roche Light Cycler480仪器中进行qRT-PCR。以2−∆∆Ct法计算相对表达量。使用SPSS22进行单因素方差分析,使用Graph Pad Prism 8.0进行图像绘制。
表 1 qRT-PCR引物Table 1. Primers for qRT-PCR引物名称
Primer name引物序列(5′-3′)
Primer sequences( 5′-3′)PcACY1-1F CCTCCTCTCCCTCCTCGAAA PcACY1-1R GAGTTGAGGAGGAGGGAGGA PcACY1-2F CTCAACTCCCACACCGACTC PcACY1-2R GTACTGGATGGCGATGCACT ACTIN-F GGGCTCATAGACTGATCACAAG ACTIN-R TTGTGCTTACCGACCTAATGG 2. 结果与分析
2.1 多花黄精ACY1基因家族成员信息
基于多花黄精全长转录组数据库,鉴定获得2个PcACY1家族成员(表2),命名为PcACY1-1和PcACY1-2,对多花黄精ACY1蛋白质特性进行分析,PcACY1-1的CDS区序列长度为1371 bp,编码含456 aa,分子量为50.06 kD的稳定蛋白, PcACY1-2的CDS区序列长度为1317 bp,编码含438 aa,分子量为48.02 kD的不稳定蛋白,2个成员等电点(pI)为5.35和5.42,表明其主要在弱酸性细胞环境中起作用,均为亲水蛋白,具有信号肽,无跨膜结构,亚细胞定位预测结果显示,2个家族成员均定位在细胞质中。利用DNAMAN 7.0进行序列比对,结果如图1所示,PcACY1-1和PcACY1-2两基因的氨基酸序列比对相似性为53.39%。
表 2 多花黄精ACY1基因家族成员信息Table 2. Information on ACY1 family of P. cyrtonema基因名称
Gene name转录组基因ID
Accession No.编码区
CDS/bp长度
Length/aa分子量
MW/kD等电点
PI不稳定指数
Instability
index (II)亲水性
Gravity信号肽
Singal
peptide脂肪指数
Aliphatic
Index跨膜结构
Trans membrane
domain亚细胞定位
Subcellular
localizationPcACY1-1 A21014918a_transcript_7940 1371 456 50.06 5.35 32.99 −0.173 YES 87.52 0 细胞质 Cytoplasm PcACY1-2 A21014918a_transcript_9264 1317 438 48.02 5.42 50.75 −0.103 YES 86 0 细胞质 Cytoplasm 2.2 PcACY1家族成员保守结构域分析
对多花黄精ACY1的蛋白质结构域进行预测分析(表3),发现PcACY1-1含有肽酶M20、ArgE/DapE_CS和N-酰基-L-酰胺水解酶3个结构域,而PcACY1-2除含有前三个结构域外还含有肽酶M20-二聚体和Bact _外肽酶结构域。
表 3 PcACY1蛋白保守结构域Table 3. Conserved domain of PcACY1 protein基因名称
Gene name登录号
Accession名称
Name位置
MatchesPcACY1-1 IPR002933 Peptidase_M20 
IPR001261 ArgE/DapE_CS 
IPR010159 N-acyl_aa_amidohydrolase 
PcACY1-2 IPR001261 ArgE/DapE_CS 
IPR002933 Peptidase_M20 
IPR036264 Bact_exopeptidase_dim_dom 
IPR011650 Peptidase_M20_dimer 
IPR010159 N-acyl_aa_amidohydrolase 
2.3 多花黄精PcACY1蛋白质二级结构、三级结构预测
利用在线软件SWISS-MODEL中的同源建模预测多花黄精PcACY1蛋白质的三维空间结构,如图2所示,图中紫色部分为预测的结合位点。
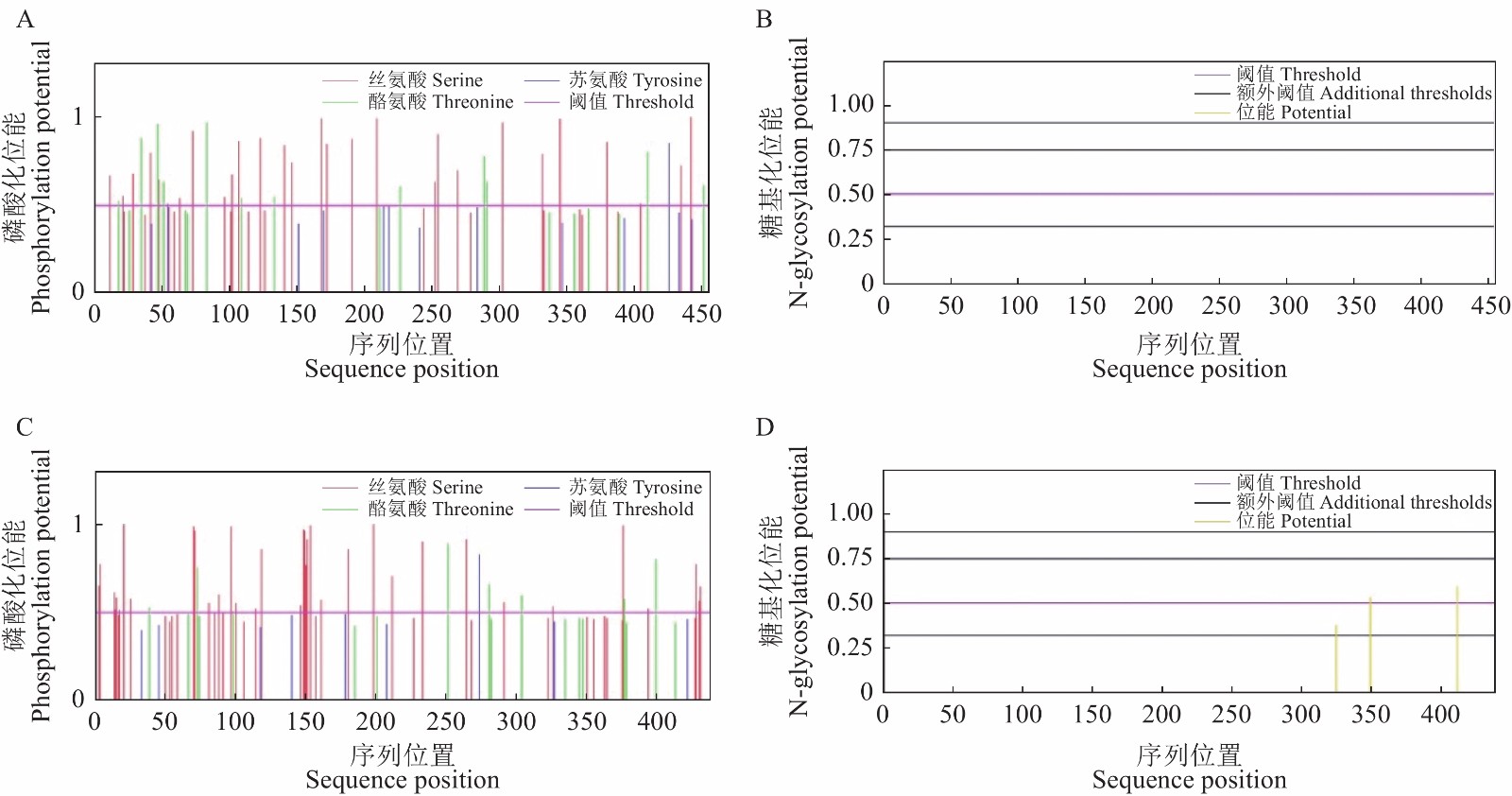
利用在线软件NetPhos对PcACY1蛋白质进行磷酸化位点和糖基化位点预测,结果如图3所示,PcACY1蛋白均存在着磷酸化位点,PcACY1-1共有73个,其中丝氨酸(Ser)40 个,苏氨酸(Thr)20个,酪氨酸(Tyr)13个,PcACY1-2共有磷酸化位点80个,其中丝氨酸(Ser)52 个,苏氨酸(Thr)18个,酪氨酸(Tyr)10个;当P 值>阈值,认为该位点存在糖基化,故PcACY1-1不具有糖基化位点,PcACY1-2具有2个糖基化位点,分别是位于348和411位的天冬酰胺(N)。
![]() 图 3 PcACY1蛋白磷酸化位点分析和糖基化位点分析A:PcACY1-1中预测磷酸位点位置,B:PcACY1-1中预测糖基化位点位置,C:PcACY1-2中预测磷酸位点位置,D:PcACY1-2中预测糖基化位点位置。Figure 3. Phosphorylation and N-glycosylation sites of PcACY1 proteinsA: Predicted phosphorylation sites in PcACY1-1; B: predicted N-glycosylation sites in PcACY1-1; C: predicted phosphorylation sites in PcACY1-2; D: predicted N-glycosylation sites in PcACY1-2.
图 3 PcACY1蛋白磷酸化位点分析和糖基化位点分析A:PcACY1-1中预测磷酸位点位置,B:PcACY1-1中预测糖基化位点位置,C:PcACY1-2中预测磷酸位点位置,D:PcACY1-2中预测糖基化位点位置。Figure 3. Phosphorylation and N-glycosylation sites of PcACY1 proteinsA: Predicted phosphorylation sites in PcACY1-1; B: predicted N-glycosylation sites in PcACY1-1; C: predicted phosphorylation sites in PcACY1-2; D: predicted N-glycosylation sites in PcACY1-2.2.4 植物ACY1家族的进化特性
为进一步了解植物 ACY1家族成员物种分布情况,从NCBI中获取32种植物的ACY1进行植物学分类,并对每个物种分布ACY1家族成员的数量情况进行统计分析,结果如表4所示。ACY1家族成员分布的物种较为广泛,不仅在被子植物有分布,而且在最古老的苔藓植物、蕨类植物植物中也有分布,32种植物的65个ACY1家族成员的物种分布在17目18科 中,其中双子叶植物占多数,大部分物种的 ACY1家族成员都是2个,只有蕨类植物江南卷柏是1个,少数具有3个成员。
表 4 植物ACY1家族成员分布和数量Table 4. Distribution and number of ACY1 family members in plants分类 Classification 物种
Species数量
Number门
Divisio纲
Classis目
Ordo科
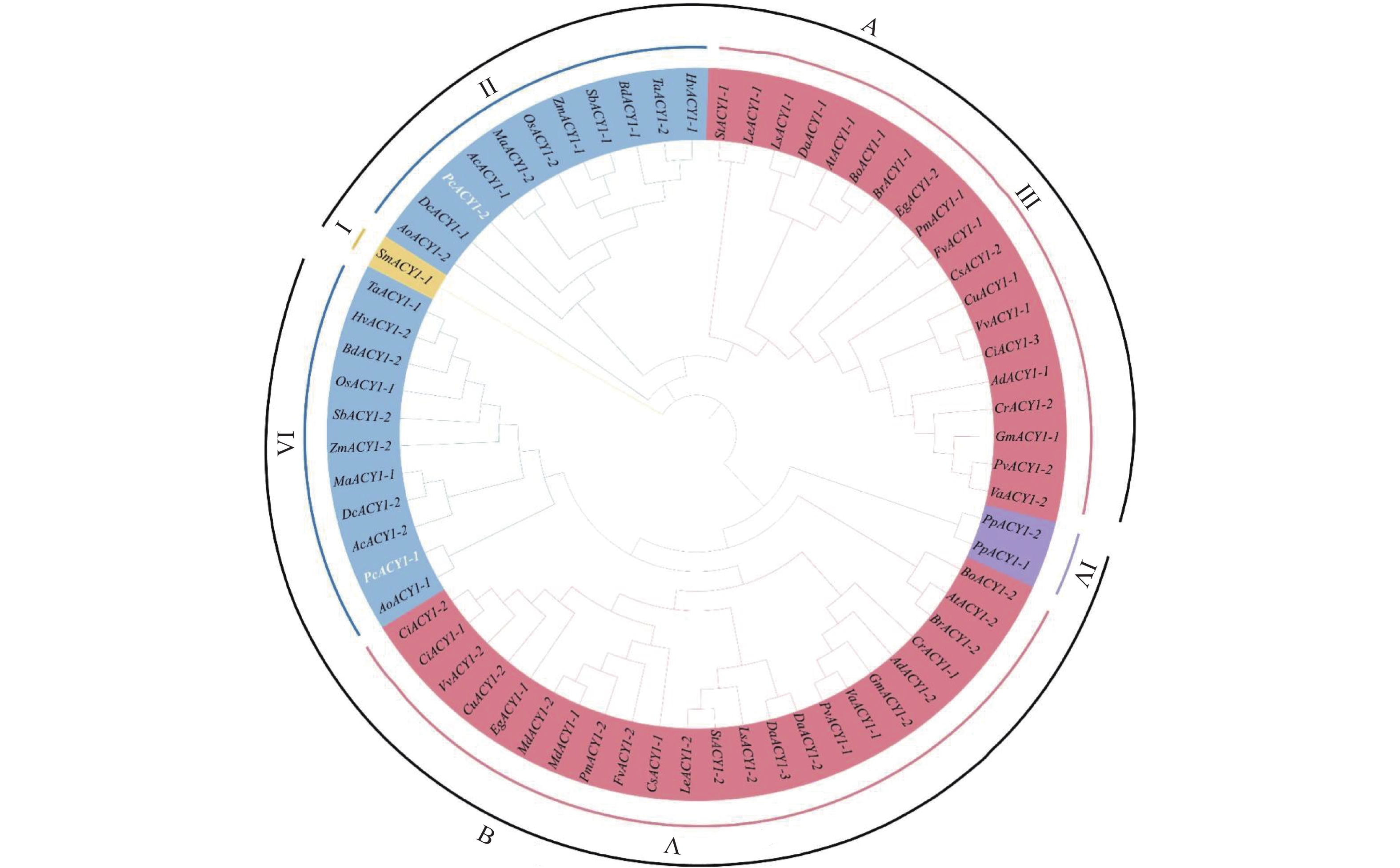
Quncs苔藓植物门 Bryophyta 藓纲 Musci 真藓目 Eubryales 葫芦藓科 Funariaceae 小立碗藓 Pp 2 蕨类植物门 Pteridophyta 石松纲 Lycopodiinae 卷柏目 Selaginellales 卷柏科 Selaginellaceae 江南卷柏 Sm 1 被子植物门 Angiosperms 单子叶植物纲 Monocotyledons 禾本目 Poales 禾本科 Poaceae 水稻 Os 2 小麦 Ta 2 玉米 Zm 2 二穗短柄草 Bd 2 大麦 Hv 2 双色高粱 Sb 2 百合目 Liliflorae 百合科 Liliaceae 石刁柏 Ao 2 粉状胚乳目 Farinosae 凤梨科 Bromeliaceae 菠萝 Ac 2 芭蕉目 Scitamineae 芭蕉科 Musaceae 香蕉 Ma 2 百合目 Liliflorae 薯蓣科 Dioscoreaceae 薯蓣 Dc 2 双子叶植物纲 Dicotyledons 蔷薇目 Rosales 豆科 Leguminosae 大豆 Gm 2 花生 Ad 2 豇豆 Va 2 鹰嘴豆 Cr 2 菜豆 Pv 2 蔷薇科 Rosaceae 野草莓 Fv 2 日本梅 Pm 2 苹果 Md 2 十字花目 Brassicales 十字花科 Brassicaceae 拟南芥 At 2 芜菁 Br 2 甘蓝 Bo 2 茄目 Solanales 茄科 Solanaceae 马铃薯 St 2 番茄 Le 2 葫芦目 Cucurbitales 葫芦科 Cucurbitaceae 黄瓜 Cu 2 无患子目 Sapindales 芸香科 Rutaceae 甜橙 Cs 2 葡萄目 Vitales 葡萄科 Vitaceae 葡萄 Vv 2 桃金娘目 Myrtales 桃金娘科 Myrtaceae 巨桉 Eg 2 伞形目 Apiales 伞形科 Apiaceae 胡萝卜 Da 3 壳斗目 Fagales 胡桃科 Juglandaceae 山核桃 Ci 3 菊目 Asterales 菊科 Asteraceae 莴苣 Ls 2 对32个物种以及多花黄精的ACY1家族进行系统发育进化树分析,结果如图4所示,不同物种的ACY1基因可分为2大类,即A和B,这两大类又可细分为6小类,命名为ClassⅠ-Ⅵ,由于基因的差异性分为两个大的分支A和B,两个分支中再因纲目单子叶和双子叶分为两部分,单子叶是ClassⅡ和Ⅵ,双子叶是ClassⅢ和Ⅴ,苔藓门和蕨类门分属ClassⅠ和ClassⅣ,除少数物种外,多数物种的ACY1家族成员都有着科内物种聚集更近的现象,如单子叶植物的禾本科、双子叶植物的豆科、蔷薇科和十字花科等,此外,古老的苔藓植物小立碗藓和蕨类植物江南卷柏,单独聚为一枝,并且由图2可知,多花黄精和石刁柏在亲缘关系上最近,这与它们同属于百合科植物一致,其中多花黄精的两个ACY1基因各分布在ClassⅡ和Ⅵ中,推测多花黄精两个ACY1基因在功能上具有其特异性。
![]() 图 4 植物ACY1家族成员进化树蓝色区域(A-ClassⅡ和B-ClassⅥ)为单子叶植物,红色区域(A-ClassⅢ和B-ClassⅤ)为双子叶植物,黄色区域(A-ClassⅠ)为苔藓植物,紫色区域(B-ClassⅣ)为蕨类植物。Figure 4. Phylogenetic tree of ACY1 family members in plantsBlue-colored areas (A-Class II and B-Class VI) are monocotyledons; red-colored areas (A-Class III and B-Class V), dicotyledons; yellow-colored areas (A-Class I), mosses; and purple-colored areas (B-Class IV), ferns.
图 4 植物ACY1家族成员进化树蓝色区域(A-ClassⅡ和B-ClassⅥ)为单子叶植物,红色区域(A-ClassⅢ和B-ClassⅤ)为双子叶植物,黄色区域(A-ClassⅠ)为苔藓植物,紫色区域(B-ClassⅣ)为蕨类植物。Figure 4. Phylogenetic tree of ACY1 family members in plantsBlue-colored areas (A-Class II and B-Class VI) are monocotyledons; red-colored areas (A-Class III and B-Class V), dicotyledons; yellow-colored areas (A-Class I), mosses; and purple-colored areas (B-Class IV), ferns.2.5 基因结构和保守基序分析
基因结构分析(图5-A)显示,ACY1基因除多花黄精、莴苣和黄瓜的一个成员以外都具有外显子和内含子,除此外ACY1基因家族在不同物种间绝大部分的外显子数和内含子数无较大差异,都具有外显子3~5个,内含子2~4个的典型结构,且亲缘关系相近的基因,其基因结构和长度也较为相似,但不同亚组物种的内含子长度有其差异性。
保守基序(motif)分析(图5-B)显示,上述33个物种中ACY1家族中存在17个可信度较高的motif,分别命名为 motif 1 ~ motif 17,每条基因的motif数在6~14,而motif2、motif3、motif7、motif8、motif10和motif11是所有基因的共有motif,是这家族最为保守的6个基序,推测是该基因功能的构成成分,涵盖了M20_AcylaseI_like、Ac-peptdase-euk和ArgE3个保守结构域;除了MaACY1、AoACY1-1、AoACY1-1、BoACY1-2、TaACY1-1和CrACY1-2这6个基因外,其余基因的motif数基本为13个,其中ClassⅡ中大部分具有置信度较高的motif17,而ClassⅢ和ClassⅤ大部分具有置信度较高的motif16,总体而言ACY1家族保守性较高。
2.6 ACY1基因家族启动子顺式作用元件分析
对不同物种ACY1启动子区进行顺式作用元件预测和分析结果表明,植物65个ACY1家族成员的顺式作用元件的数目分布除核心元件外可分为4类:光响应元件、激素响应元件、逆境胁迫和生长发育相关的响应元件(图6)。其中光响应元件包括87.7%的基因都具有的G-Box,66.2%的基因有的Box-4,以及AE-box、GATA-motif、GT1-motif、Sp1和TCT-motif;激素响应元件包括73.8%基因具有的脱落酸响应(ABRE)、70.7%基因具有的茉莉酸甲酯响应(CGTCA-motif、TGACG-motif )、水杨酸响应(TCA-element)、赤霉素响应(P-box、GARE-motif、TATC-box)和生长素响应(AuxRR-core);逆境胁迫响应元件包括75.4%基因具有的厌氧诱导(ARE)、56.9%基因具有的干旱诱导(MBS),以及低温响应(LTR)、损伤防御(WUN-motif)、防御和应答响应元件(TC-rich repeats)和缺氧诱导(GC-motif);而生长发育相关则包括分生组织表达(CAT-box)、种子特异性表达相关(RY-element)、胚乳相关(GCN4-motif)、栅栏叶肉细胞分化(HD-Zip 1)、玉米蛋白代谢(O2-site)和昼夜节律(circadian)。
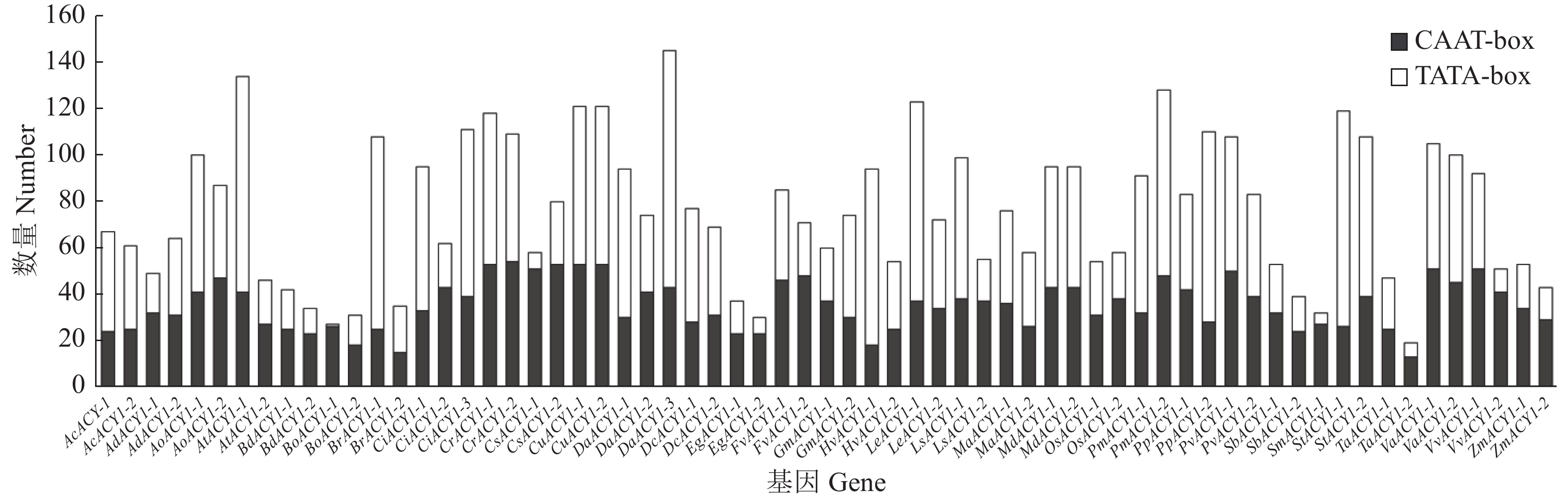
而不同物种间ACY1启动子区的核心元件即CAAT-box和TATA-box数目分布结果(图7)显示,每个基因都含有TATA-box与GAAT-box,其中CAAT-box数目大体差异不大,一般在20~40,而所有基因的TATA-box数目差异较大,数目从BoACY1-1的1个到DaACY1-1的102个间不等。
2.7 多花黄精ACY1盐胁迫及不同器官的转录组基因表达模式
基于NaCl处理下和不同组织器官的多花黄精转录组数据,搜集多花黄精ACY1家族表达的FPKM值,绘制热图并分析表达趋势(图8),在盐胁迫转录组中PcACY1的两个成员在受到48 h盐胁迫后有轻微上调表达,表明PcACY1-1和PcACY1-2可能响应盐胁迫,并被盐胁迫诱导上调表达。
如图6所示PcACY1基因的两个成员在各个组织部位均有表达,且 PcACY1-1在果实中表达量较高,在根中表达量次之,PcACY1-2在茎秆中表达量较高,根和根状茎中表达量相对较低,多花黄精ACY1家族成员在不同的组织器官表现出不同的表达模式,说明其成员可能存在功能的特异性分化。
2.8 植物ACY1基因在不同非生物胁迫下的表达模式分析
为研究不同物种ACY1基因在非生物胁迫下的表达模式,利用植物生物学生物分析资源BAR(http://bar.utoronto.ca/)中的转录组学数据集中挖掘模式植物拟南芥和水稻在非生物胁迫环境下的表达情况,并建立热图来比较不同同系物的表达,并根据与PcACY1基因的序列相似性确定基因命名。
在拟南芥中,由图9-A可知,AtACY1-1和AtACY1-2在苗和根部位的表达变化不一致,在低温条件时,AtACY1两成员在苗和根部位大体上均下调表达;在盐胁迫时,AtACY1两成员在苗中上调表达,在根中下调表达,且在6 h处理时明显下调,后续有略上升,但还是低于初始状态;模拟干旱条件下,AtACY1两成员在两部位均上调表达,且AtACY1-2表达变化更强烈;高温胁迫时,AtACY1两成员在苗和根部位表达量逐渐升高,AtACY1-1在根中表达量上升更强烈,AtACY1-2在苗中的表达量先下降后上升。而在水稻中,由图9-B可见,OsACY1-1在3种胁迫条件下均下调表达,而OsACY1-2均上调表达,但表达量OsACY1-1显著高于OsACY1-2。
2.9 多花黄精不同组织器官及ACY1盐胁迫的qRT-PCR验证分析
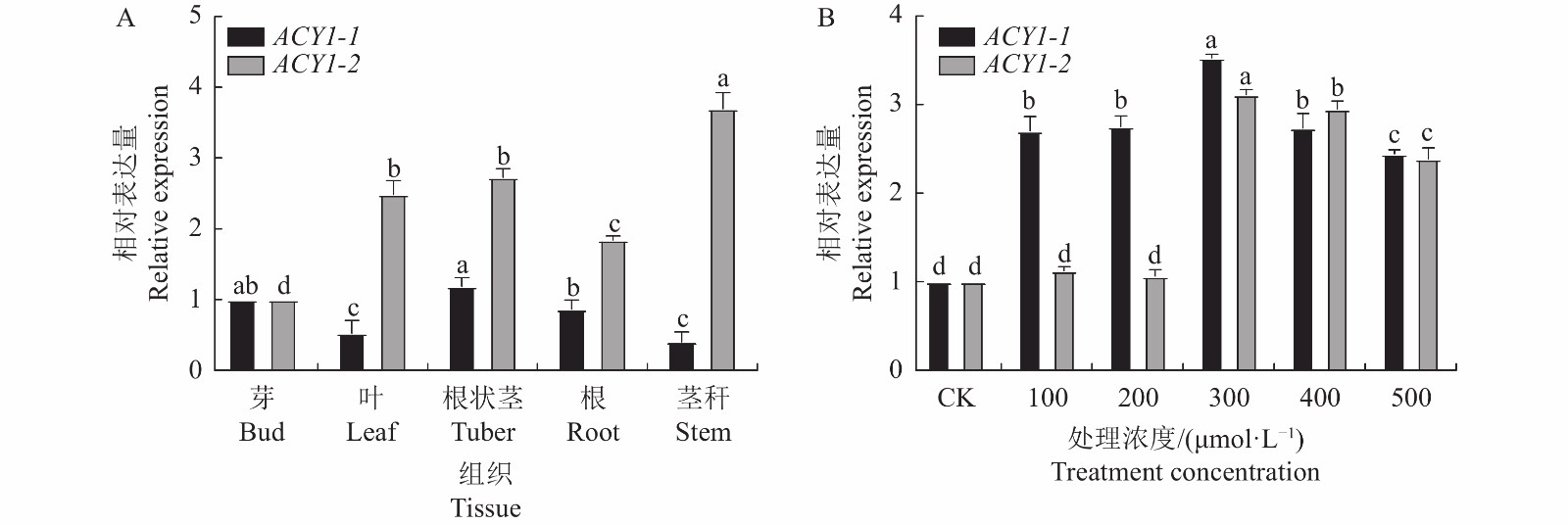
如图10-A所示,PcACY1-2在各组织器官中的相对表达量均高于PcACY1-1,PcACY1-1在根状茎中的表达量较高,PcACY1-2成员在茎秆中的表达量最高,是对照的5倍多,在根状茎中的表达量次之,与FPKM值的整体变化大趋相同。
![]() 图 10 PcACY1 家族成员在多花黄精不同的组织器官(A)和不同浓度NaCl处理下(B)的表达不同小写字母表示同一基因不同处理间差异显著(P<0. 05)。Figure 10. Expressions of PcACY1 family members in tissues and organs of P. cyrtonema (A) and under treatments of different concentration of NaCl (B)Datas with different lowercase letters indicate significant differences between different treatments of the same gene at P<0.05.
图 10 PcACY1 家族成员在多花黄精不同的组织器官(A)和不同浓度NaCl处理下(B)的表达不同小写字母表示同一基因不同处理间差异显著(P<0. 05)。Figure 10. Expressions of PcACY1 family members in tissues and organs of P. cyrtonema (A) and under treatments of different concentration of NaCl (B)Datas with different lowercase letters indicate significant differences between different treatments of the same gene at P<0.05.进一步通过qRT-PCR验证上述两个基因在盐胁迫下和不同组织器官中的表达模式,制图并分析。定量分析结果显示(图10-B),在NaCl处理下,相较于CK多花黄精ACY1家族成员在各浓度均呈现先上升表达模式,随着NaCl处理浓度的升高,多花黄精ACY1家族成员总体均呈现先上升后下降的表达模式,PcACY1-1在300 μmol·L-1时表达量达最高峰,后开始降低,PcACY1-2在400 μmol·L-1时到达最高点,后呈现降低趋势,值得注意的是PcACY1-1在100 μmol·L-1处理时就表现出显著上调表达,而PcACY1-2在300 μmol·L-1时才出现显著上调表达,说明PcACY1家族成员均受到盐胁迫的诱导,但两成员对盐胁迫强度的表达模式不一致。
3. 讨论与结论
3.1 PcACY1家族成员鉴定与进化特性
本研究通过生物信息学方法从多花黄精全长转录组中鉴定出2个PcACY1成员,亚细胞预测结果显示,PcACY1成员均定位于细胞质中,这与在动物中研究所表明在胞质中发挥作用[2]的结论一致,推测其可能通过影响多花黄精部分蛋白质的功能,从而调节其氧化应激反应。同时从植物基因组数据库Phytozome中筛选出32个物种的65个ACY1成员,包括古老的苔藓类植物、蕨类植物,还包括被子植物,推测苔藓类植物可能是植物ACY1家族的祖先;根据植物学分类和系统进化分析发现,大部分植物物种的ACY1家族成员都是两个,表明这些物种中ACY1基因的数量相对恒定,基因的特异性和物种的亲缘关系是影响植物ACY1家族成员进化的两个重要因素,且PcACY1-1和石刁柏ACY1-1聚在一起,由此可见,基因功能的特异性在影响植物ACY1成员聚类的程度是高于物种亲缘关系的。除个别物种如薯蓣、黄瓜和多花黄精外,其他大部分物种的ACY1成员均含有3或4个内含子,且保守基序结果的近似一致性也表明植物ACY1进化具有较高的保守性,这与以往研究发现编码ACY1的基因在鱼类、青蛙、小鼠和人类中进化上是保守的[15, 23, 24]较为一致。
3.2 PcACY1可能参与多花黄精的生长发育
在人类研究中, Chen等[12]发现ACY1通过ERK和TGF-1转化生长因子信号通路介导人神经母细胞瘤细胞的增殖和迁移。而有研究[20]检测了ACY1玉米不同时期和组织的表达情况,发现ZmACY1具有组织特异性和阶段特异性,同时发现过表达ZmACY1的转基因烟草株系的多数表型形状如根长、株高和茎粗等指标均超过野生型,这表明过量表达ZmACY1可促进本氏烟草的生长发育。本研究中植物ACY1启动子结合位点分析发现大部分植物都具有分生组织表达和昼夜节律等元件,同时转录组表达数据和qRT-PCR验证结果也表明ACY1可能参与多花黄精不同组织部位生长发育的调控,PcACY1成员表达具有组织特异性,且PcACY1-2成员在根状茎中的表达量较高,推测其可能在多花黄精根状茎膨大过程中起重要作用,因此,推测PcACY1是多花黄精生长发育过程不可或缺的调控因子。
3.3 PcACY1可能参与多花黄精的抗逆反应
在人类和动物研究中,氨基酰化酶1广泛参与尿素循环[7]、氨清除[21]和氧化应激反应[20, 25, 26]。氨基酰化酶1通过游离氨基酸脱乙酰化来完成这些蛋白质分解的最后一步,这些氨基酸可被回收并用于制造其他蛋白质[7]。如ACY1可催化N2-乙酰-L-鸟氨酸转化为L-鸟氨酸[27],L-鸟氨酸是一种非必需氨基酸[28],同时也是精氨酸酶降解的产物,主要用于尿素循环,以清除多余的NH4+。ACY1同时也能催化逆反应,如乙酰化氨基酸的合成[29]。在植物研究中也发现,氨基酰基酶-1 (DS2)已被发现在植物防御系统和水杨酸依赖通路中发挥作用,沉默DS2可增强烟草本氏菌对疫霉菌的抗性[21];ZmACY1可被盐、干旱、茉莉酸和水杨酸诱导变化[20],表明其在防御平衡方面起作用。本研究中,多花黄精ACY1基因家族两成员间在盐胁迫中的表达量和表达模式差异较大,PcACY1基因家族对盐胁迫的响应程度不同,但均有明显表达变化,表明其在盐胁迫方面有潜在作用,这也与前人研究结果较为一致。
-
图 3 PcACY1蛋白磷酸化位点分析和糖基化位点分析
A:PcACY1-1中预测磷酸位点位置,B:PcACY1-1中预测糖基化位点位置,C:PcACY1-2中预测磷酸位点位置,D:PcACY1-2中预测糖基化位点位置。
Figure 3. Phosphorylation and N-glycosylation sites of PcACY1 proteins
A: Predicted phosphorylation sites in PcACY1-1; B: predicted N-glycosylation sites in PcACY1-1; C: predicted phosphorylation sites in PcACY1-2; D: predicted N-glycosylation sites in PcACY1-2.
图 4 植物ACY1家族成员进化树
蓝色区域(A-ClassⅡ和B-ClassⅥ)为单子叶植物,红色区域(A-ClassⅢ和B-ClassⅤ)为双子叶植物,黄色区域(A-ClassⅠ)为苔藓植物,紫色区域(B-ClassⅣ)为蕨类植物。
Figure 4. Phylogenetic tree of ACY1 family members in plants
Blue-colored areas (A-Class II and B-Class VI) are monocotyledons; red-colored areas (A-Class III and B-Class V), dicotyledons; yellow-colored areas (A-Class I), mosses; and purple-colored areas (B-Class IV), ferns.
图 10 PcACY1 家族成员在多花黄精不同的组织器官(A)和不同浓度NaCl处理下(B)的表达
不同小写字母表示同一基因不同处理间差异显著(P<0. 05)。
Figure 10. Expressions of PcACY1 family members in tissues and organs of P. cyrtonema (A) and under treatments of different concentration of NaCl (B)
Datas with different lowercase letters indicate significant differences between different treatments of the same gene at P<0.05.
表 1 qRT-PCR引物
Table 1 Primers for qRT-PCR
引物名称
Primer name引物序列(5′-3′)
Primer sequences( 5′-3′)PcACY1-1F CCTCCTCTCCCTCCTCGAAA PcACY1-1R GAGTTGAGGAGGAGGGAGGA PcACY1-2F CTCAACTCCCACACCGACTC PcACY1-2R GTACTGGATGGCGATGCACT ACTIN-F GGGCTCATAGACTGATCACAAG ACTIN-R TTGTGCTTACCGACCTAATGG 表 2 多花黄精ACY1基因家族成员信息
Table 2 Information on ACY1 family of P. cyrtonema
基因名称
Gene name转录组基因ID
Accession No.编码区
CDS/bp长度
Length/aa分子量
MW/kD等电点
PI不稳定指数
Instability
index (II)亲水性
Gravity信号肽
Singal
peptide脂肪指数
Aliphatic
Index跨膜结构
Trans membrane
domain亚细胞定位
Subcellular
localizationPcACY1-1 A21014918a_transcript_7940 1371 456 50.06 5.35 32.99 −0.173 YES 87.52 0 细胞质 Cytoplasm PcACY1-2 A21014918a_transcript_9264 1317 438 48.02 5.42 50.75 −0.103 YES 86 0 细胞质 Cytoplasm 表 3 PcACY1蛋白保守结构域
Table 3 Conserved domain of PcACY1 protein
基因名称
Gene name登录号
Accession名称
Name位置
MatchesPcACY1-1 IPR002933 Peptidase_M20 
IPR001261 ArgE/DapE_CS 
IPR010159 N-acyl_aa_amidohydrolase 
PcACY1-2 IPR001261 ArgE/DapE_CS 
IPR002933 Peptidase_M20 
IPR036264 Bact_exopeptidase_dim_dom 
IPR011650 Peptidase_M20_dimer 
IPR010159 N-acyl_aa_amidohydrolase 
表 4 植物ACY1家族成员分布和数量
Table 4 Distribution and number of ACY1 family members in plants
分类 Classification 物种
Species数量
Number门
Divisio纲
Classis目
Ordo科
Quncs苔藓植物门 Bryophyta 藓纲 Musci 真藓目 Eubryales 葫芦藓科 Funariaceae 小立碗藓 Pp 2 蕨类植物门 Pteridophyta 石松纲 Lycopodiinae 卷柏目 Selaginellales 卷柏科 Selaginellaceae 江南卷柏 Sm 1 被子植物门 Angiosperms 单子叶植物纲 Monocotyledons 禾本目 Poales 禾本科 Poaceae 水稻 Os 2 小麦 Ta 2 玉米 Zm 2 二穗短柄草 Bd 2 大麦 Hv 2 双色高粱 Sb 2 百合目 Liliflorae 百合科 Liliaceae 石刁柏 Ao 2 粉状胚乳目 Farinosae 凤梨科 Bromeliaceae 菠萝 Ac 2 芭蕉目 Scitamineae 芭蕉科 Musaceae 香蕉 Ma 2 百合目 Liliflorae 薯蓣科 Dioscoreaceae 薯蓣 Dc 2 双子叶植物纲 Dicotyledons 蔷薇目 Rosales 豆科 Leguminosae 大豆 Gm 2 花生 Ad 2 豇豆 Va 2 鹰嘴豆 Cr 2 菜豆 Pv 2 蔷薇科 Rosaceae 野草莓 Fv 2 日本梅 Pm 2 苹果 Md 2 十字花目 Brassicales 十字花科 Brassicaceae 拟南芥 At 2 芜菁 Br 2 甘蓝 Bo 2 茄目 Solanales 茄科 Solanaceae 马铃薯 St 2 番茄 Le 2 葫芦目 Cucurbitales 葫芦科 Cucurbitaceae 黄瓜 Cu 2 无患子目 Sapindales 芸香科 Rutaceae 甜橙 Cs 2 葡萄目 Vitales 葡萄科 Vitaceae 葡萄 Vv 2 桃金娘目 Myrtales 桃金娘科 Myrtaceae 巨桉 Eg 2 伞形目 Apiales 伞形科 Apiaceae 胡萝卜 Da 3 壳斗目 Fagales 胡桃科 Juglandaceae 山核桃 Ci 3 菊目 Asterales 菊科 Asteraceae 莴苣 Ls 2 -
[1] 于冰. ACY-1在结直肠癌中的表达及作用机制[D]. 济南: 山东大学, 2017. YU B. Expression and mechanism of ACY-1 in colorectal cancer [D]. Jinan: Shandong University, 2017. (in Chinese)
[2] COOK R M, BURKE B J, BUCHHAGEN D L, et al. Human aminoacylase-1. Cloning, sequence, and expression analysis of a chromosome 3p21 gene inactivated in small cell lung cancer [J]. Journal of Biological Chemistry, 1993, 268(23): 17010−17017. DOI: 10.1016/S0021-9258(19)85294-8
[3] LINDNER H A, LUNIN V V, ALARY A, et al. Essential roles of zinc ligation and enzyme dimerization for catalysis in the aminoacylase-1/M20 family [J]. Journal of Biological Chemistry, 2003, 278(45): 44496−44504. DOI: 10.1074/jbc.M304233200
[4] FONES W S, LEE M. Hydrolysis of n-acyl derivatives of alanine and phenylalanine by acylase i and carboxypeptidase [J]. Journal of Biological Chemistry, 1953, 201(2): 847−856. DOI: 10.1016/S0021-9258(18)66242-8
[5] POLEVODA B, SHERMAN F. Nalpha-terminal acetylation of eukaryotic proteins [J]. The Journal of Biological Chemistry, 2000, 275(47): 36479−36482. DOI: 10.1074/jbc.R000023200
[6] SHI H J, HAYES M T, KIRANA C, et al. Overexpression of aminoacylase 1 is associated with colorectal cancer progression [J]. Human Pathology, 2013, 44(6): 1089−1097. DOI: 10.1016/j.humpath.2012.09.015
[7] PERRIER J, DURAND A, GIARDINA T, et al. Catabolism of intracellular N-terminal acetylated proteins: Involvement of acylpeptide hydrolase and acylase [J]. Biochimie, 2005, 87(8): 673−685. DOI: 10.1016/j.biochi.2005.04.002
[8] 张露莹. ACY1调控mTOR信号通路的机制[D]. 广州: 广州医科大学, 2020. ZHANG L Y. Mechanism of ACY1 regulation of mTOR signaling pathway [D]. Guangzhou: Guangzhou Medical University, 2020. (in Chinese)
[9] 国家药典委员会. 中华人民共和国药典: 一部[M]. 北京: 中国医药科技出版社, 2010. [10] 中国科学院中国植物志编辑委员会. 中国植物志: 第十五卷[M]. 北京: 科学出版社, 1978. [11] BIRNBAUM S M, LEVINTOW L, KINGSLEY R B, et al. Specificity of amino acid acylases [J]. Journal of Biological Chemistry, 1952, 194(1): 455−470. DOI: 10.1016/S0021-9258(18)55898-1
[12] CHEN Z M, GAO J H, SUN J, et al. Aminoacylase 1 (ACY-1) mediates the proliferation and migration of neuroblastoma cells in humans through the ERK/transforming growth factor β (TGF-β) signaling pathways [J]. Medical Science Monitor, 2020, 27: e928813.
[13] TYLKI-SZYMANSKA A, GRADOWSKA W, SOMMER A, et al. Aminoacylase 1 deficiency associated with autistic behavior [J]. Journal of Inherited Metabolic Disease, 2010, 33(3): 211−214.
[14] SASS J O, VAITHILINGAM J, GEMPERLE-BRITSCHGI C, et al. Expanding the phenotype in aminoacylase 1 (ACY1) deficiency: Characterization of the molecular defect in a 63-year-old woman with generalized dystonia [J]. Metabolic Brain Disease, 2016, 31(3): 587−592. DOI: 10.1007/s11011-015-9778-6
[15] ZHONG Y, ONUKI J, YAMASAKI T, et al. Genome-wide analysis identifies a tumor suppressor role for aminoacylase 1 in iron-induced rat renal cell carcinoma [J]. Carcinogenesis, 2009, 30(1): 158−164. DOI: 10.1093/carcin/bgn255
[16] CHENG Q A, GU S H, LIU Z W, et al. Expressional divergence of the fatty acid-amino acid conjugate-hydrolyzing aminoacylase 1 (L-ACY-1) in Helicoverpa armigera and Helicoverpa assulta [J]. Scientific Reports, 2017, 7: 8721. DOI: 10.1038/s41598-017-09185-2
[17] JIN G Z, DONG H, YU W L, et al. A novel panel of biomarkers in distinction of small well-differentiated HCC from dysplastic nodules and outcome values [J]. BMC Cancer, 2013, 13: 161. DOI: 10.1186/1471-2407-13-161
[18] STOCKER P, BRUNEL J M, DE REZENDE L, et al. Aminoacylase 1-catalysed deacetylation of bioactives epoxides mycotoxin-derived mercapturates; 3, 4-epoxyprecocenes as models of cytotoxic epoxides [J]. Biochimie, 2012, 94(8): 1668−1675. DOI: 10.1016/j.biochi.2012.01.006
[19] KOREISHI M, NAKATANI Y, OOI M, et al. Purification, characterization, molecular cloning, and expression of a new aminoacylase from Streptomyces mobaraensis that can hydrolyze N-(middle/long)-chain-fatty-acyl-L-amino acids as well as N-short-chain-acyl-L-amino acids [J]. Bioscience, Biotechnology, and Biochemistry, 2009, 73(9): 1940−1947. DOI: 10.1271/bbb.90081
[20] CHEN D B, LI J H, JIAO F C, et al. ZmACY-1 antagonistically regulates growth and stress responses in Nicotiana benthamiana [J]. Frontiers in Plant Science, 2021, 12: 593001. DOI: 10.3389/fpls.2021.593001
[21] NAKANO M, NISHIHARA M, YOSHIOKA H, et al. Silencing of DS2 aminoacylase-like genes confirms basal resistance to Phytophthora infestans in Nicotiana benthamiana [J]. Plant Signaling & Behavior, 2014, 9(2): e28004.
[22] CHEN C J, CHEN H, ZHANG Y, et al. TBtools: An integrative toolkit developed for interactive analyses of big biological data [J]. Molecular Plant, 2020, 13(8): 1194−1202. DOI: 10.1016/j.molp.2020.06.009
[23] BAUMEIER C, KAISER D, HEEREN J, et al. Caloric restriction and intermittent fasting alter hepatic lipid droplet proteome and diacylglycerol species and prevent diabetes in NZO mice [J]. Biochimica et Biophysica Acta (BBA) - Molecular and Cell Biology of Lipids, 2015, 1851(5): 566−576. DOI: 10.1016/j.bbalip.2015.01.013
[24] LINDNER H A, TÄFLER-NAUMANN M, RÖHM K H. N-acetylamino acid utilization by kidney aminoacylase-1 [J]. Biochimie, 2008, 90(5): 773−780. DOI: 10.1016/j.biochi.2007.12.006
[25] CAIRA S, IANNELLI A, SCIARRILLO R, et al. Differential representation of liver proteins in obese human subjects suggests novel biomarkers and promising targets for drug development in obesity [J]. Journal of Enzyme Inhibition and Medicinal Chemistry, 2017, 32(1): 672−682. DOI: 10.1080/14756366.2017.1292262
[26] KUHNS E H, SEIDL-ADAMS I, TUMLINSON J H. A lepidopteran aminoacylase (L-ACY-1) in Heliothis Virescens (Lepidoptera: Noctuidae) gut lumen hydrolyzes fatty acid-amino acid conjugates, elicitors of plant defense [J]. Insect Biochemistry and Molecular Biology, 2012, 42(1): 32−40. DOI: 10.1016/j.ibmb.2011.10.004
[27] KUTMON M, RIUTTA A, NUNES N, et al. WikiPathways: Capturing the full diversity of pathway knowledge [J]. Nucleic Acids Research, 2016, 44(D1): D488−D494. DOI: 10.1093/nar/gkv1024
[28] WU G, MORRIS S. Arginine metabolism: nitric oxide and beyond [J]. Biochemical Journal, 1998: 331-336.
[29] LINDNER H, HÖPFNER S, TÄFLER-NAUMANN M, et al. The distribution of aminoacylase I among mammalian species and localization of the enzyme in porcine kidney [J]. Biochimie, 2000, 82(2): 129−137. DOI: 10.1016/S0300-9084(00)00191-7




 下载:
下载: