SYBR Green I RT-PCR Assay for Quantitative Detection of Salmonella in Irrigation Water
-
摘要:目的 建立基于SYBR Green I嵌合染料法的灌溉水源中沙门氏菌实时荧光定量PCR(Quantitative Real-time PCR, qPCR)检测方法。方法 以沙门氏菌侵袭蛋白A (invA)基因为靶基因,设计1对特异性qPCR检测引物,在对qPCR反应体系和反应条件进行优化后,通过特异性实验和灵敏度实验对qPCR方法进行验证,并制作灌溉水源中沙门氏菌qPCR定量标准曲线,最后与国标方法对比检测灌溉水源中沙门氏菌污染来验证该方法的准确性。结果 经过优化的qPCR检测方法灵敏度检测结果显示最低检测限量为1×10−1 pg·μL−1,特异性检测结果显示具有良好的特异性,干扰菌株对本qPCR检测方法无影响,所制作的qPCR扩增标准曲线在2×100~2×105 cfu·mL−1有较好的线性关系,相关系数为0.999 6,能对沙门氏菌进行准确的定量分析。该方法在与国标方法对比检测灌溉水源中沙门氏菌污染时具有同等准确性。结论 本研究建立的基于SYBR Green I嵌合荧光法的实时荧光定量PCR检测方法,可以满足灌溉水源中沙门氏菌的定量检测要求。Abstract:Objective A SYBR Green I-based quantitative RT-PCR (qPCR) method was developed for detecting salmonella in irrigation water.Method For the methodology development a pair of primers were designed based on the sequences of the invasion protein A gene (invA) of Salmonella. qPCR reaction conditions were optimized, the assay tested for specificity and sensitivity, and a standard curve of amplification constructed. Test results on a specimen of contaminated irrigation water using the qPCR assay were compared for detection accuracy with those obtained from the national standard method.Result The newly developed qPCR assay showed a minimum detection limit of 1×10−1pg·μL−1 and free of interference from genomic nucleic acids of non-target microbes. The constructed linear standard curve between 2×100cfu·mL−1 and 2×105 cfu·mL−1 had a high correlation coefficient of 0.999 6. The assay demonstrated same accuracy as did the national standard method in detecting salmonella in irrigation water.Conclusion The newly established qPCR assay could be adequately applied for salmonella detection in agriculture irrigation water.
-
Keywords:
- Quantitative RT-PCR /
- Irrigated water /
- Salmonella
-
0. 引言
【研究意义】沙门氏菌是与人类健康相关的最重要的食源性致病菌之一,因其导致的食物中毒病例占细菌性食物中毒病例的前2位[1]。据世界卫生组织不完全统计,每年至少有1600万沙门氏菌感染病例,导致约60万人死亡。仅2005 — 2011年国外发生的19起蔬菜因食源性致病菌污染而引起的疾病案例中,有11起的致病元凶是沙门氏菌[2]。在我国沙门氏菌也是食源性疾病的主要病原体,其中80%左右的细菌性食源中毒事件为沙门氏菌污染所致[3]。目前国内已发现多种蔬菜中存在沙门氏菌污染情况[4-6]。蔬菜产地环境的质量安全是蔬菜质量安全的前提,其中灌溉水源又是产地环境中最重要因素之一,但蔬菜从农田到餐桌的每个环节均可能发生沙门氏菌污染[7-8],近年来,随着对蔬菜供应链环节中沙门氏菌污染风险和污染来源的深入研究,发现蔬菜沙门氏菌污染主要还是发生在栽培环节,特别是灌溉水源中沙门氏菌而导致的蔬菜沙门氏菌污染的风险最大,也是防控蔬菜中沙门氏菌污染重要节点[8-12]。【前人研究进展】沙门氏菌侵袭蛋白A (invA)基因是最常用的沙门氏菌分子检测靶标,根据invA基因序列已成功开发出了多个探针法的沙门氏菌实时荧光定量PCR(Quantitative Real-time PCR,qPCR)检测方法。González-Escalona等[13]根据invA序列设计沙门氏菌qPCR检测引物,在鲜食农产品中沙门氏菌最低检出限为0.08 cfu·g−1,鸡蛋中沙门氏菌最低检出限为0.05 cfu·g−1;Siala等[14]以invA序列为靶点设计了特异性qPCR检测引物对500种食物中沙门氏菌污染进行检测,发现禽肉、牛奶、红肉、鱼、蛋糕、乳制品、烤肉等食物中存在不同程度沙门氏菌污染;Bai等[15]采用以invA基因为靶基因的qPCR方法和富集培养方法对牛淋巴结中沙门氏菌污染进行检测,发现qPCR方法比富集培养方法具有更高的灵敏度。针对沙门氏菌的检测主要采用中华人民共和国国家标准《食品安全国家标准 食品微生物学检验 沙门氏菌检验》(GB 4789.4—2016)的方法[16],不但步骤繁琐且耗时费力,而最终检测结果也仅为定性分析。而qPCR方法则可以进行定量分析,目前已成功应用于沙门氏菌、副溶血弧菌、阴沟肠杆菌、蜡样芽孢杆菌、单核细胞增生李斯特氏菌等多种病原菌的定量分析[17-23]。【本研究切入点】目前有关沙门氏菌qPCR检测方法研究多基于探针法,而基于嵌合荧光法的沙门氏菌定量检测方法却鲜有研究报道。【拟解决的关键问题】以沙门氏菌特有侵袭蛋白A(invasion protein A)基因为靶基因,建立基于SYBR Green I嵌合荧光法的沙门氏菌实时荧光定量PCR检测方法,以期满足灌溉水源中沙门氏菌污染的定量检测需求,为蔬菜产地环境中沙门氏菌污染的防控和蔬菜质量安全的提升提供技术支撑。
1. 材料与方法
1.1 试验材料
1.1.1 菌株来源
供试菌株包括7个微生物菌株,其中1个为鼠伤寒沙门氏菌作为阳性对照菌株,其余6个菌株为干扰菌株。7个菌株来自中国工业微生物菌种保藏管理中心(CICC)、中国普通微生物菌种保藏管理中心(CGMCC)和美国典型培养物保存中心(ATCC)。具体菌株编号及来源等信息见表1。
表 1 供试菌株Table 1. Salmonella strains applied for experimentation序号
No.菌株名称
Strains name拉丁学名
Scientific name of strains菌株编号
Strains number菌株来源
Source of strains1 鼠伤寒沙门氏菌 Salmonella typhimurium 1.1174 CGMCC 2 金黄色葡萄球菌 Staphylococcus aureus 25923 ATCC 3 大肠埃希氏菌 Escherichia coli 25922 ATCC 4 阿氏肠杆菌 Enterobacter asburiae 1.876 CGMCC 5 大肠埃希氏菌O157:H7 Escherichia coli O157:H7 10907 CICC 6 单核细胞增生李斯特氏菌 Listeria monocytogenes 1.9136 CGMCC 7 英诺克李斯特氏菌 Listeria innocua 1.2990 CGMCC 1.1.2 材料与试剂
胰蛋白胨大豆肉汤(TSB)、胰蛋白胨大豆琼脂(TSA)、蛋白胨缓冲水(BPW)和无菌均质袋(北京陆桥技术有限责任公司);TB Green® Premix Ex Taq™ (Tli RNaseH Plus,日本Takara公司);细菌基因组DNA提取试剂盒(北京天根公司);NaCl(分析纯,阿拉丁公司);荧光定量PCR 八联管(无锡Nest公司)。
1.1.3 仪器与设备
实时荧光定量PCR仪(BIO-RAD™ CFX96 Touch,美国伯乐公司);拍击式均质器(easyMixTM,法国AES Chemunex公司);台式离心机(Sigma 1-14,德国希格玛公司);超净工作台(C2HJH-C1112B,上海智城公司);核酸蛋白定量仪(Qubit 2,美国Thermo Scientific公司);恒温培养箱(DHP-9082型,上海一恒)。
1.1.4 灌溉水源样品
供试灌溉水源样品采集自闽清县东桥镇绿辉蔬菜种植农场,属于当地地面径流,主要为山泉和小溪汇聚而成,使用干净的矿泉水瓶采集该处灌溉水源样品600 mL,样品3次重复,放入带有冰块的保温箱中运回实验室。具体灌溉水源理化指标:pH 7.32,总氮(TN)0.85 mg·L−1,总磷(TP)0.037 mg·L−1,高锰酸钾指数(CODMn)10.51 mg·L−1,氨氮(NH4-N)0.35 mg·L−1。
1.2 试验方法
1.2.1 引物设计
以沙门氏菌侵袭蛋白A (invA)基因为靶基因(Genbank Accession: M90846.1),采用Primer Premier 5.0软件设计了1对qPCR引物用于沙门氏菌实时荧光定量检测,引物序列invA-F: 5'-TATCGTGACCCGCGTAAATG-3,'invA-R: 5'- GGGAATCCCGGCAGAGTT-3'。引物交由上海生工生物工程公司合成。
1.2.2 沙门氏菌计数及基因组DNA提取
将−70 ℃保存的沙门氏菌菌株在TSA平板内划线后,置于37 ℃培养箱倒置培养16~24 h,在5 mL TSB液体培养基中使用无菌牙签挑取TSA平板内的单个沙门氏菌菌落进行接种,置于37 ℃、160 r·min−1振荡培养16~24 h后,取1 mL震荡培养的TSB液体培养基转接于新的100 mL TSB液体培养基中,37 ℃、160 r·min−1振荡培养至OD600=0.55的对数生长期。使用无菌生理盐水以10倍浓度梯度将对数生长期菌液进行稀释后,吸取100 μL稀释液在TSA平板上进行涂布,置37 ℃培养18~24 h后计数。
吸取1 mL沙门氏菌培养菌液在台式离心机上12 000 r·min−1离心4 min,弃上清,菌体沉淀使用细菌基因组DNA提取试剂盒按步骤提取。提取的沙门氏菌基因组DNA使用Qubit 2测定DNA浓度后,置于−20 ℃保存备用。
1.2.3 qPCR反应体系和反应条件优化
qPCR反应体系:12.5 μL 2×TB Green Premix Ex Taq (Tli RNaseH Plus),上下游引物(10 μmol·L −1),DNA模板10 ng,灭菌超纯水补足25 μL体积;反应条件:95 ℃,30 s;95 ℃,5 s,退火温度,30 s,72 ℃,30 s,40个循环。分别对qPCR反应体系和反应条件中的引物浓度、退火温度进行优化,其中引物终浓度分别为0.1、0.2、0.4、0.6、0.8、1.0 μmol·L −1;退火温度分别设置55、56、57、58、59、60、61 ℃。每个因子设置3个重复,同时设置无菌水为模板的阴性对照,利用Bio-Rad CFX Manager软件进行数据分析,以扩增曲线获得最小的Ct值和熔解曲线不产生非特异性扩增峰为指标,确定最佳引物浓度和退火温度。
1.2.4 qPCR反应特异性检测
以鼠伤寒沙门氏菌为阳性对照菌株,其余大肠埃希氏菌、产气肠杆菌、大肠埃希氏菌O157:H7等6个菌株为干扰菌株,采取已优化的反应体系和条件对沙门氏菌qPCR反应进行特异性分析。反应结束后,以PCR管内荧光信号强弱变化来判断阴阳性,阳性具有明显的荧光扩增曲线,阴性则没有明显的荧光扩增曲线。每个菌株DNA模板均设置3个重复,同时以无菌水为模板设置阴性对照。
1.2.5 qPCR反应灵敏度检测
将沙门氏菌DNA以10倍递减稀释为1×104~1×10−2 pg·μL−1 7个质量浓度,以上述浓度的沙门氏菌DNA为模板,对沙门氏菌qPCR反应灵敏度进行检测。反应结束后,阳性应具有明显的荧光扩增曲线,阴性则没有明显的荧光扩增曲线。每个浓度的DNA模板均设置3个重复,同时以无菌水为模板设置阴性对照。
1.2.6 标准曲线的建立
取6份24 mL沙门氏菌阴性的灌溉水源样品,加入1 mL稀释至适宜质量浓度的沙门氏菌培养液,制备沙门氏菌质量浓度为0、2×100、2×101、2×102、2×103、2×104、2×105 cfu·mL−1的灌溉水源样品,然后全部转移至含225 mL蛋白胨缓冲水的无菌均质袋中,置拍击式均质器中拍打1~2 min混匀。将含不同质量浓度沙门氏菌的灌溉水源样品置37 ℃预增菌4 h后,混匀后取1 mL预增菌液10 000 r·min−1离心5 min收集菌体,使用细菌基因组DNA提取试剂盒提取沙门氏菌DNA,用于灌溉水源中沙门氏菌的标准曲线建立。
以不同浓度沙门氏菌提取的DNA作为定量标准品,利用优化的qPCR反应体系和条件检测各浓度标准品的Ct值,以qPCR扩增Ct值为纵坐标,不同浓度沙门氏菌对数值为横坐标,建立Ct值与不同浓度沙门氏菌对数值线性函数关系的定量标准曲线。
1.2.7 qPCR方法与国标方法对比验证
采用qPCR方法和国标方法《食品安全国家标准 食品微生物学检验 沙门氏菌检验》(GB 4789.4—2016),分别对采自全省的39份灌溉水源样品中沙门氏菌污染情况进行检测,来验证所建立的qPCR方法在灌溉水源沙门氏菌污染检测方面的准确性。
2. 结果与分析
2.1 qPCR方法的建立
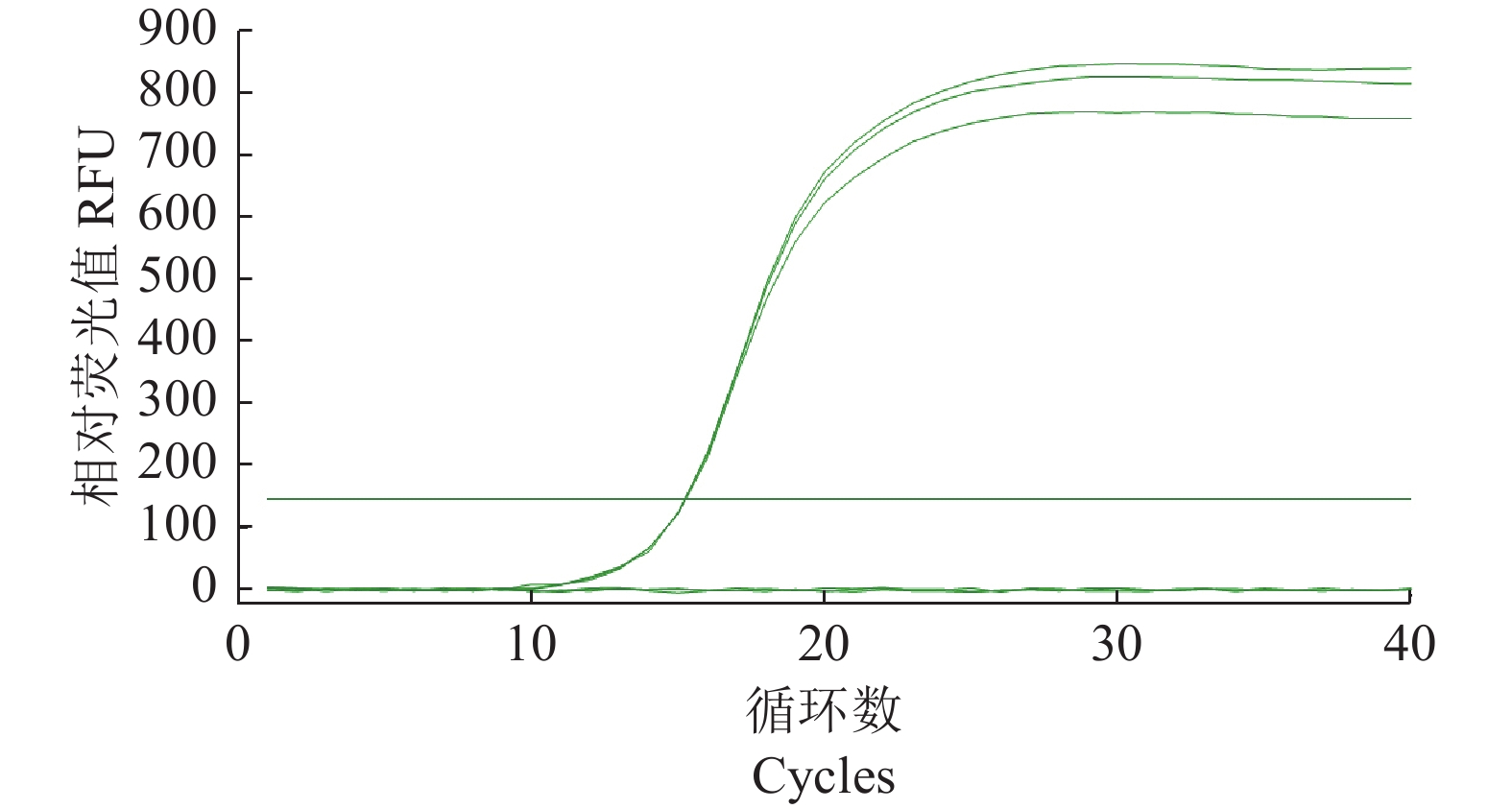
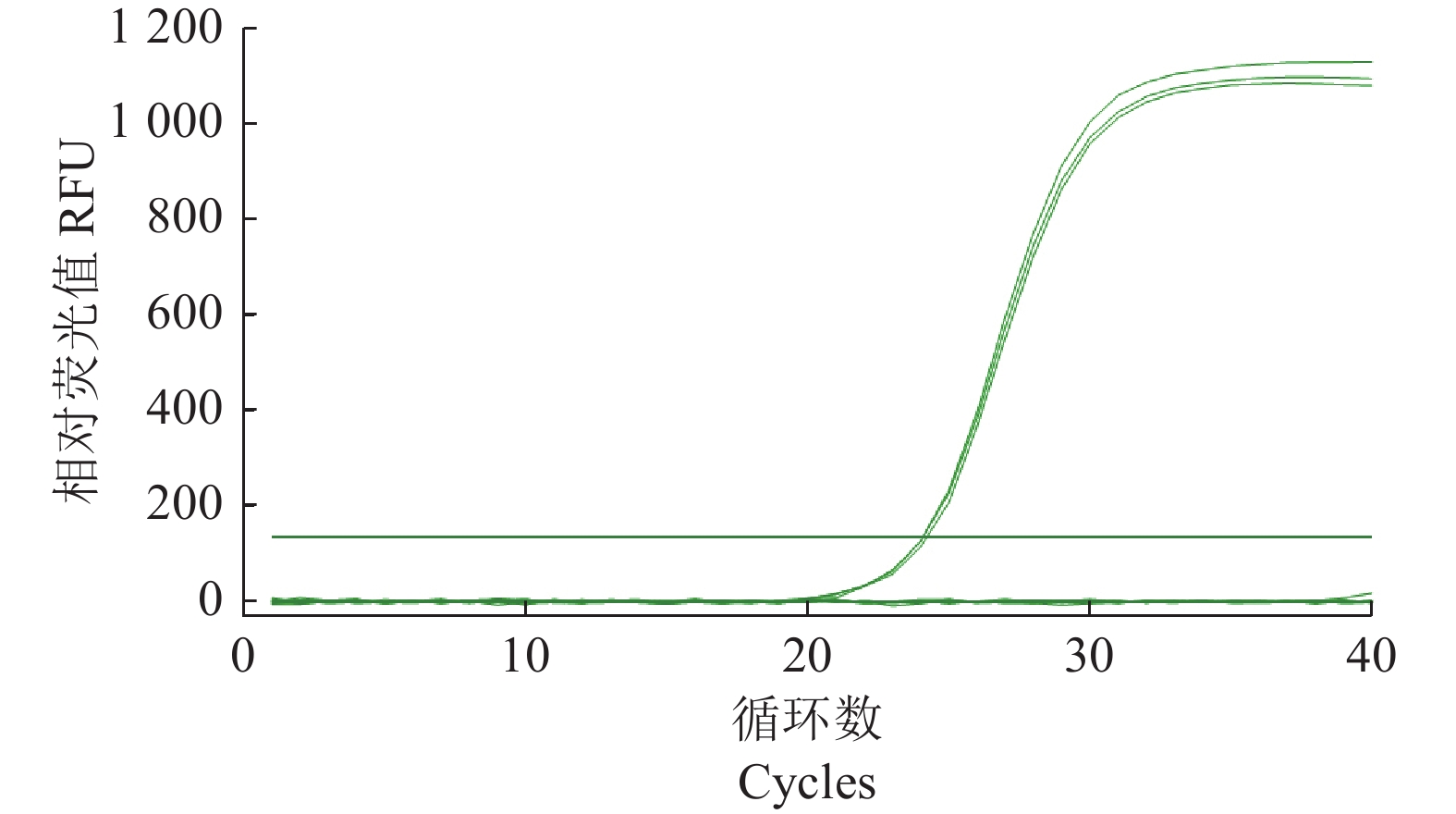
分别对qPCR反应体系和反应条件中引物浓度、退火温度进行优化,确定灌溉水源中沙门氏菌qPCR检测最佳反应体系(25 μL):12.5 μL 2×TB Green Premix Ex Taq (Tli RNaseH Plus),0.2 μmol·L−1 invA-F,0.2 μmol·L−1 invA-R,DNA模板10 ng,灭菌超纯水补足25 μL体积。优化退火温度后的反应条件如下:95 ℃,30 s;95 ℃,5 s,58 ℃,30 s,72 ℃,30 s,40个循环。优化的qPCR反应结果如图1所示,其扩增曲线良好反映了PCR的指数增长阶段和平台阶段,熔解曲线则只有单一扩增峰,未见非特异性扩增峰。
2.2 特异性检测结果
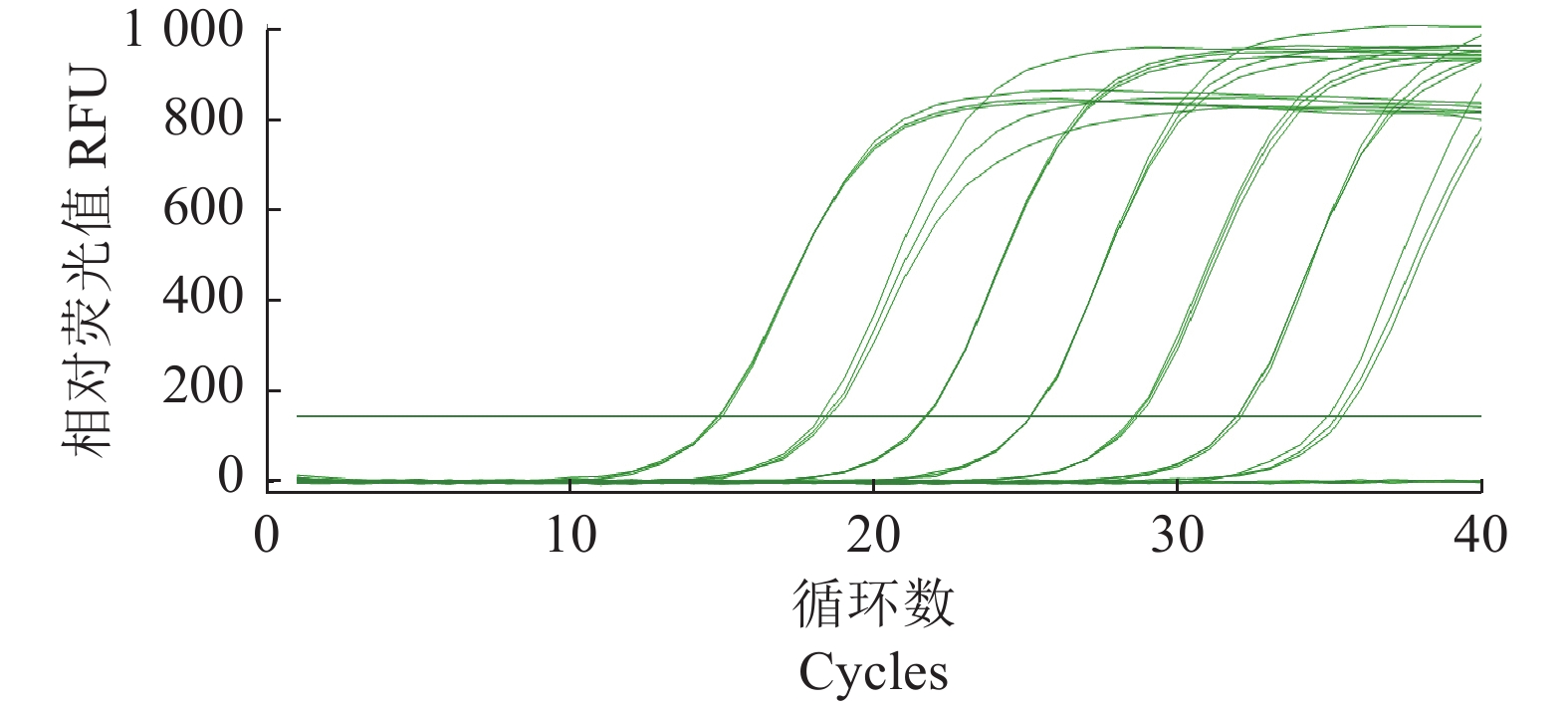
采用优化的qPCR方法,以鼠伤寒沙门氏菌菌株和干扰菌株DNA为模板进行qPCR反应特异性分析,结果如图2所示,除鼠伤寒沙门氏菌能产生明显的扩增曲线呈现阳性结果外,其余金黄色葡萄球菌、大肠埃希氏菌等干扰菌株DNA及无菌水阴性对照均无明显扩增曲线产生,呈现阴性结果。表明所建立的qPCR方法对沙门氏菌具有良好的特异性。
2.3 灵敏度检测结果
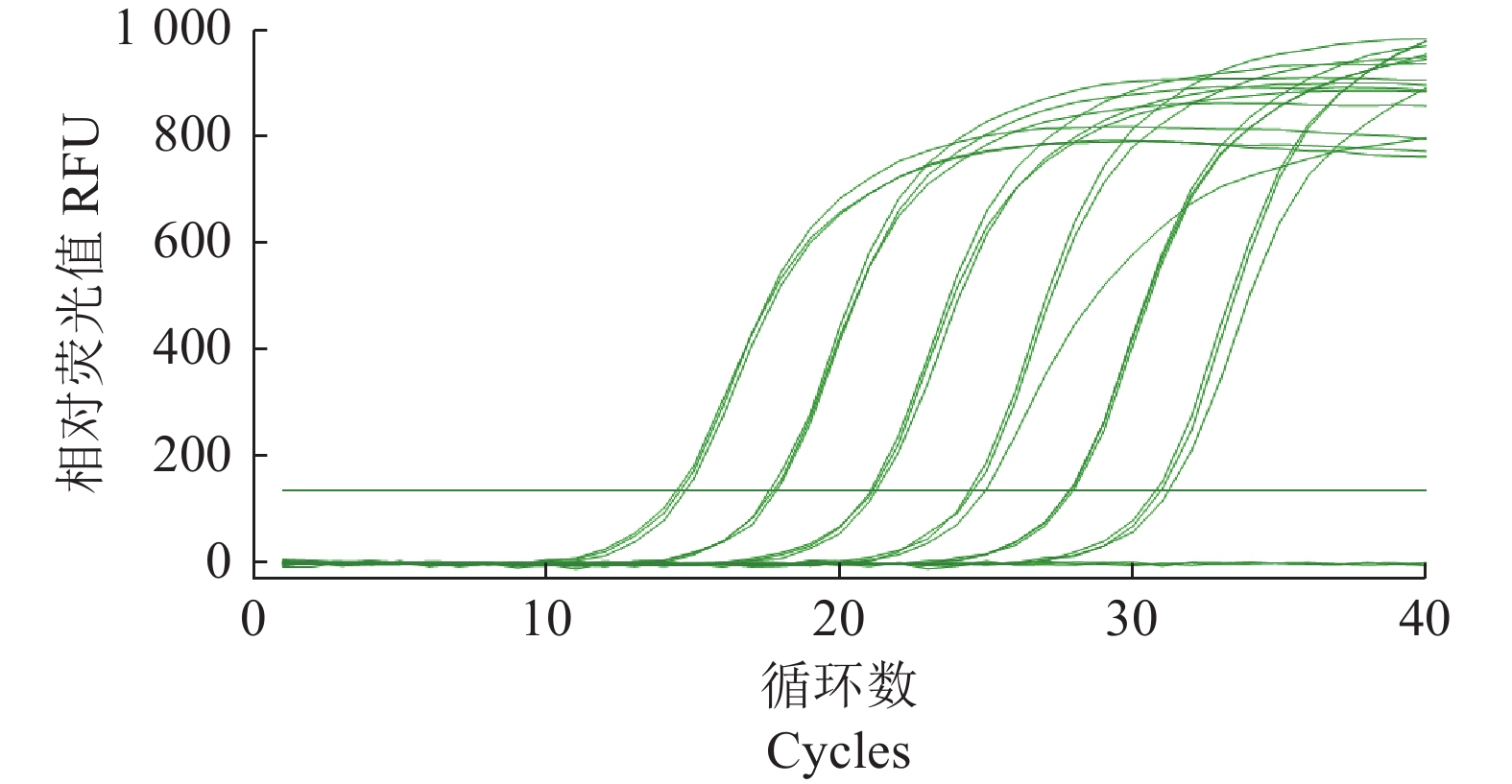
分别以7个不同浓度沙门氏菌DNA为模板进行扩增来验证qPCR体系的灵敏度,结果如图3所示,在每管25 μL qPCR反应体系中当沙门氏菌DNA质量浓度为1×104~1×10−1 pg·μL−1时均出现明显的荧光扩增曲线,3个平行样品间扩增曲线重复性好且Ct值<35,而当沙门氏菌DNA质量浓度为1×10−2 pg·μL−1时,虽然也出现了荧光扩增曲线,但3个平行样品间扩增曲线重复性较差且Ct值>35(图3),表明本qPCR反应体系的检测灵敏度为1×10−1 pg·μL−1的沙门氏菌DNA。
2.4 标准曲线结果
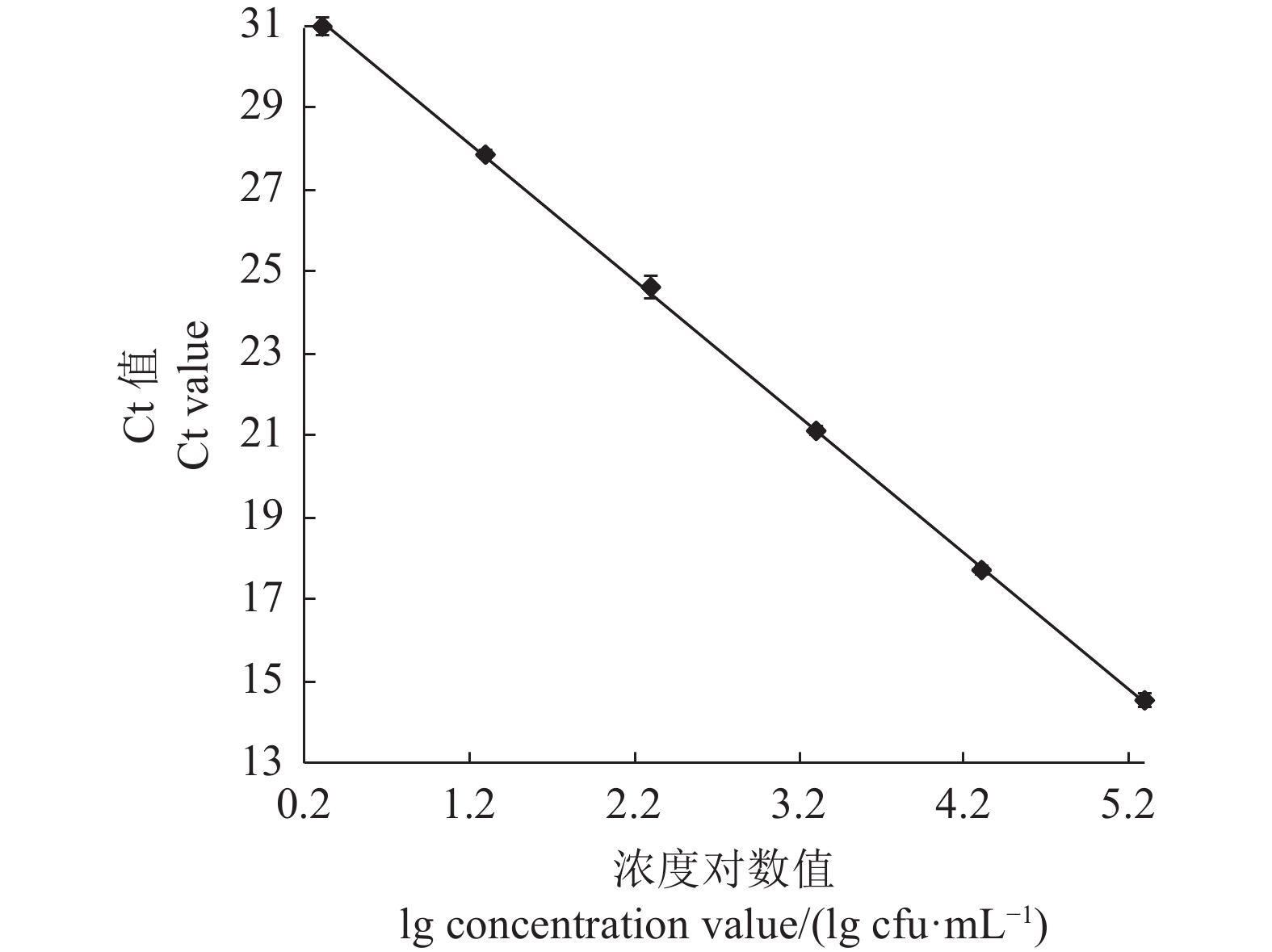
利用优化好的沙门氏菌qPCR反应体系和反应条件对6个10倍梯度稀释的定量标准品进行扩增,建立不同沙门氏菌浓度对数值与Ct值对应关系的定量标准曲线。结果显示,所制作的标准曲线在2×100~2×105 cfu·mL−1的Ct值相差均匀,符合qPCR的Ct值与起始浓度对数值之间的线性关系(图4)。以Ct值为纵坐标,不同沙门氏菌浓度对数值为横坐标,得出起始浓度对数值与Ct值的线性回归方程为Y=−3.3157X+32.097,斜率约为−3.3157,截距为32.097,相关系数R2为0.999 6(图5)。在对样品进行检测时,根据其Ct值和线性方程可以计算该样品沙门氏菌浓度值。
2.5 qPCR方法与国标方法对比验证结果
采用qPCR方法和国标方法《食品安全国家标准 食品微生物学检验 沙门氏菌检验》(GB 4789.4—2016),分别对采自全省的39份灌溉水源样品中沙门氏菌污染情况进行检测,来验证所建立的qPCR方法应用于蔬菜灌溉水源中沙门氏菌污染检测的准确性。结果显示(图6),2种方法均检测到1份灌溉水源沙门氏菌阳性样品,且为同一份灌溉水源样品,其余38份灌溉水源样品检测结果均显示为沙门氏菌阴性,2种方法检测结果一致,但qPCR方法在保证准确性的前提下,将检测时间由5~7 d缩短至6 h内,降低了检测强度,提高了检测效率。此外,qPCR方法检测沙门氏菌阳性灌溉水源样品的Ct值为24,根据上述灌溉水源中沙门氏菌回归方程Y=−3.3157X+32.097,计算该灌溉水源样品中污染的沙门氏菌数为277 cfu·mL−1。
3. 讨论与结论
目前qPCR方法包括探针法和嵌合荧光法,探针法由于探针引物设计要求高且合成费用贵,导致探针法使用成本较高,从而限制了探针法的推广应用。而嵌合荧光法则可以使用普通PCR引物再辅以荧光染料如SYBR Green I,在引物设计上要求相对简单和使用成本上低廉,故在qPCR检测中得到越来越多应用。但SYBR Green I作为一种可结合于双链DNA(dsDNA)双螺旋小沟区域的荧光染料,在与目的扩增条带结合的同时,也会结合引物二聚体和非特异性扩增条带从而产生荧光干扰信号,因此,基于SYBR Green I嵌合荧光法的qPCR方法需要对引物浓度和退火温度进行优化,以防止引物二聚体和非特异性扩增条带的产生,同时借助溶解曲线来排除引物二聚体和非特异性扩增条带的影响。
沙门氏菌侵袭蛋白(invasion protein)基因是一类与沙门氏菌吸附和侵袭肠道上皮细胞相关的基因,包括invA-invE等多个基因,其中以invA基因的研究和应用最为广泛[13-15]。本研究根据沙门氏菌侵袭蛋白A(invA)基因序列设计1对特异性qPCR检测引物,通过对qPCR反应体系和反应条件进行优化后,建立了基于SYBR Green I嵌合荧光法的实时荧光定量PCR检测方法,其对沙门氏菌具有良好特异性,检测灵敏度为1×10−1 pg·μL−1,所制作的qPCR扩增标准曲线在2×100~2×105 cfu·mL−1有较好的线性关系,可用于沙门氏菌的定量分析。采用本qPCR方法与国标方法对39份灌溉水源样品中沙门氏菌污染进行对比检测,发现本qPCR方法与国标方法检测结果一致,表明2种检测方法具有相同的特异性和灵敏度,但在检测时间和检测效率上本qPCR方法明显优于国标方法,且能定量分析灌溉水源中沙门氏菌含量,可为蔬菜产地环境中沙门氏菌污染的防控和蔬菜质量安全的提升提供技术支撑。
-
表 1 供试菌株
Table 1 Salmonella strains applied for experimentation
序号
No.菌株名称
Strains name拉丁学名
Scientific name of strains菌株编号
Strains number菌株来源
Source of strains1 鼠伤寒沙门氏菌 Salmonella typhimurium 1.1174 CGMCC 2 金黄色葡萄球菌 Staphylococcus aureus 25923 ATCC 3 大肠埃希氏菌 Escherichia coli 25922 ATCC 4 阿氏肠杆菌 Enterobacter asburiae 1.876 CGMCC 5 大肠埃希氏菌O157:H7 Escherichia coli O157:H7 10907 CICC 6 单核细胞增生李斯特氏菌 Listeria monocytogenes 1.9136 CGMCC 7 英诺克李斯特氏菌 Listeria innocua 1.2990 CGMCC -
[1] YANG B W, QU D, ZHANG X L, et al. Prevalence and characterization of Salmonella serovars in retail meats of marketplace in Shaanxi, China [J]. International Journal of Food Microbiology, 2010, 141(1/2): 63−72.
[2] OLAIMAT A N, HOLLEY R A. Factors influencing the microbial safety of fresh produce: A review [J]. Food Microbiology, 2012, 32(1): 1−19. DOI: 10.1016/j.fm.2012.04.016
[3] WANG Y, YANG B W, WU Y, et al. Molecular characterization of Salmonella enterica serovar Enteritidis on retail raw poultry in six provinces and two National cities in China [J]. Food Microbiology, 2015, 46: 74−80. DOI: 10.1016/j.fm.2014.07.012
[4] 方敏, 郑华英, 郭爱玲, 等. 自市售生食蔬菜胡萝卜中检出沙门菌报告 [J]. 中国卫生检验杂志, 2006, 16(12):1537. DOI: 10.3969/j.issn.1004-8685.2006.12.067 FANG M, ZHENG H Y, GUO A L, et al. Report on Salmonella detection from raw vegetable carrots on the market [J]. Chinese Journal of Health Laboratory Technology, 2006, 16(12): 1537.(in Chinese) DOI: 10.3969/j.issn.1004-8685.2006.12.067
[5] 李秀桂, 黄彦, 唐振柱, 等. 南宁市生食蔬菜中病原菌污染监测 [J]. 应用预防医学, 2008, 14(6):361−363. LI X G, HUANG Y, TANG Z Z, et al. Monitoring of pathogenic bacteria pollution in raw vegetables in Nanning city [J]. Journal of Applied Preventive Medicine, 2008, 14(6): 361−363.(in Chinese)
[6] 吕新, 陈丽华, 李玥仁. 福州市生食蔬菜沙门氏菌污染状况分析 [J]. 福建农业学报, 2016, 31(3):297−300. LV X, CHEN L H, LI Y R. Salmonella contamination of raw vegetables in Fuzhou [J]. Fujian J Agricl Sci, 2016, 31(3): 297−300.(in Chinese)
[7] SANT'ANA A S, BARBOSA M S, DESTRO M T, et al. Growth potential of Salmonella spp. and Listeria monocytogenes in nine types of ready-to-eat vegetables stored at variable temperature conditions during shelf-life [J]. International Journal of Food Microbiology, 2012, 157(1): 52−58. DOI: 10.1016/j.ijfoodmicro.2012.04.011
[8] ILIC S, RAJIĆ A, BRITTON C J, et al. A scoping study characterizing prevalence, risk factor and intervention research, published between 1990 and 2010, for microbial hazards in leafy green vegetables [J]. Food Control, 2012, 23(1): 7−19. DOI: 10.1016/j.foodcont.2011.06.027
[9] SCALLAN E, HOEKSTRA R M, MAHON B E, et al. An assessment of the human health impact of seven leading foodborne pathogens in the United States using disability adjusted life years [J]. Epidemiology and Infection, 2015, 143(13): 2795−2804. DOI: 10.1017/S0950268814003185
[10] PARKER J S, WILSON R S, LEJEUNE J T, et al. An expert guide to understanding grower decisions related to fresh fruit and vegetable contamination prevention and control [J]. Food Control, 2012, 26(1): 107−116. DOI: 10.1016/j.foodcont.2011.12.025
[11] SCALLAN E, HOEKSTRA R M, ANGULO F J, et al. Foodborne illness acquired in the United States: Major pathogens [J]. Emerging Infectious Diseases, 2011, 17(1): 7−15. DOI: 10.3201/eid1701.P11101
[12] 吕新, 刘兰英, 陈丽华, 等. 可视化环介导恒温扩增法检测蔬菜产地环境灌溉水源中沙门氏菌 [J]. 食品安全质量检测学报, 2021, 12(2):492−498. LV X, LIU L Y, CHEN L H, et al. Determination of Salmonella in irrigated water of vegetable producing environment by visual loop-mediated isothermal amplification method [J]. Journal of Food Safety & Quality, 2021, 12(2): 492−498.(in Chinese)
[13] GONZÁLEZ-ESCALONA N, BROWN E W, ZHANG G D. Development and evaluation of a multiplex real-time PCR (qPCR) assay targeting ttrRSBCA locus and invA gene for accurate detection of Salmonella spp. in fresh produce and eggs [J]. Food Research International, 2012, 48(1): 202−208. DOI: 10.1016/j.foodres.2012.03.009
[14] SIALA M, BARBANA A, SMAOUI S, et al. Screening and detecting Salmonella in different food matrices in southern Tunisia using a combined enrichment/real-time PCR method: Correlation with conventional culture method [J]. Frontiers in Microbiology, 2017, 8: 2416. DOI: 10.3389/fmicb.2017.02416
[15] BAI J F, TRINETTA V, SHI X R, et al. A multiplex real-time PCR assay, based on invA and pagC genes, for the detection and quantification of Salmonella enterica from cattle lymph nodes [J]. Journal of Microbiological Methods, 2018, 148: 110−116. DOI: 10.1016/j.mimet.2018.03.019
[16] 国家卫生和计划生育委员会, 国家食品药品监督管理总局. 食品安全国家标准 食品微生物学检验 沙门氏菌检验: GB 4789.4—2016[S]. 北京: 中国标准出版社, 2017. [17] 李光伟, 邱杨, 肖性龙, 等. 沙门氏菌荧光实时定量PCR检测试剂的研制及应用 [J]. 微生物学通报, 2007, 34(3):496−499. LI G W, QIU Y, XIAO X L, et al. Research and application on detection of Salmonella sp. by FQ-PCR [J]. Microbiology, 2007, 34(3): 496−499.(in Chinese)
[18] 杨滴, 赵宇明, 秦鹏钧, 等. 肉制品中蜡样芽胞杆菌实时荧光PCR检测方法的研究 [J]. 肉类工业, 2017(6):49−52. YANG D, ZHAO Y M, QIN P J, et al. Study on real-time fluorescent PCR detection method of cerea spore bacilli in meat products [J]. Meat Industry, 2017(6): 49−52.(in Chinese)
[19] 包海燕, 岳喜庆, 武俊瑞, 等. 基于实时荧光PCR技术快速检测饲料中的蜡样芽孢杆菌 [J]. 沈阳农业大学学报, 2020, 51(3):349−354. BAO H Y, YUE X Q, WU J R, et al. Rapid detection of Bacillus cereus in feed by real-time PCR [J]. Journal of Shenyang Agricultural University, 2020, 51(3): 349−354.(in Chinese)
[20] 张晓君, 陈丽, 毕可然, 等. 副溶血弧菌的SYBR Green Ⅰ实时定量PCR检测方法建立 [J]. 食品科学, 2012, 33(8):203−206. ZHANG X J, CHEN L, BI K R, et al. Development of SYBR green-based ⅠReal-time quantitative PCR for detection of Vibrio parahaemolyticus [J]. Food Science, 2012, 33(8): 203−206.(in Chinese)
[21] 陈琳, 周青青, 顾青, 等. 实时定量PCR法快速检测水产品中的副溶血性弧菌 [J]. 浙江农业学报, 2019, 31(5):823−828. CHEN L, ZHOU Q Q, GU Q, et al. Detection of Vibrio parahaemolyticus in seafood products by real-time quantitative PCR assay [J]. Acta Agriculturae Zhejiangensis, 2019, 31(5): 823−828.(in Chinese)
[22] 周慧平, 唐连飞, 蔡文杰, 等. 奶粉中阴沟肠杆菌实时荧光PCR快速检测方法建立 [J]. 检验检疫学刊, 2016, 26(3):10−13. ZHOU H P, TANG L F, CAI W J, et al. Rapid detection of Enterobacter cloacae in powdered milk by real-time PCR [J]. Journal of Inspection and Quarantine, 2016, 26(3): 10−13.(in Chinese)
[23] 林艳艳, 邢子伟, 谭翰清. 单核细胞增生李斯特菌hlyA基因实时荧光PCR的建立与研究 [J]. 中国热带医学, 2017, 17(12):1184−1188. LIN Y Y, XING Z W, TAN H Q. Establishment and application of real-time PCR for hlyA gene of Listeria monocytogenes [J]. China Tropical Medicine, 2017, 17(12): 1184−1188.(in Chinese)
-
期刊类型引用(1)
1. 张捷,李献,张瑞,刘雨蒙,明若阳,陈佳,周巍. SYBR Green Ⅰ染料-实时荧光定量聚合酶链式反应法检测发酵乳中的霉菌和酵母含量. 食品安全质量检测学报. 2024(16): 31-38 .  百度学术
百度学术
其他类型引用(1)




 下载:
下载: