Differentiate Transcriptomes of Aerial and Creepy Agaricus bisporus Hyphae
-
摘要:目的
挖掘双孢蘑菌落形态变化可能涉及的重要基因,为双孢蘑菇种质资源评价、新品种选育提供科学依据。
方法以双孢蘑菇主栽品种As2796为试验菌株,收集同一平板上生长的贴生与气生菌丝,提取样品的RNA进行转录组测序分析。
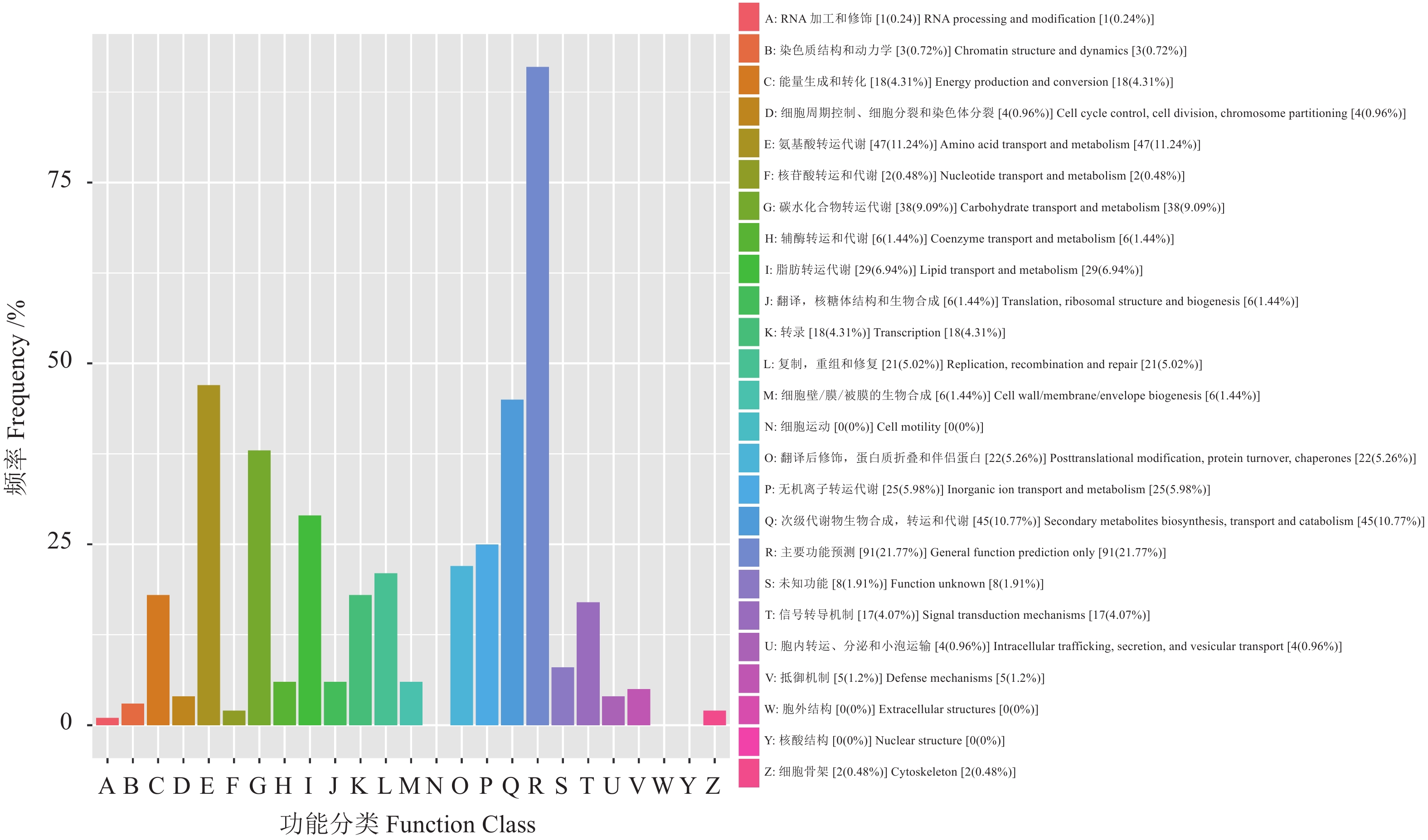
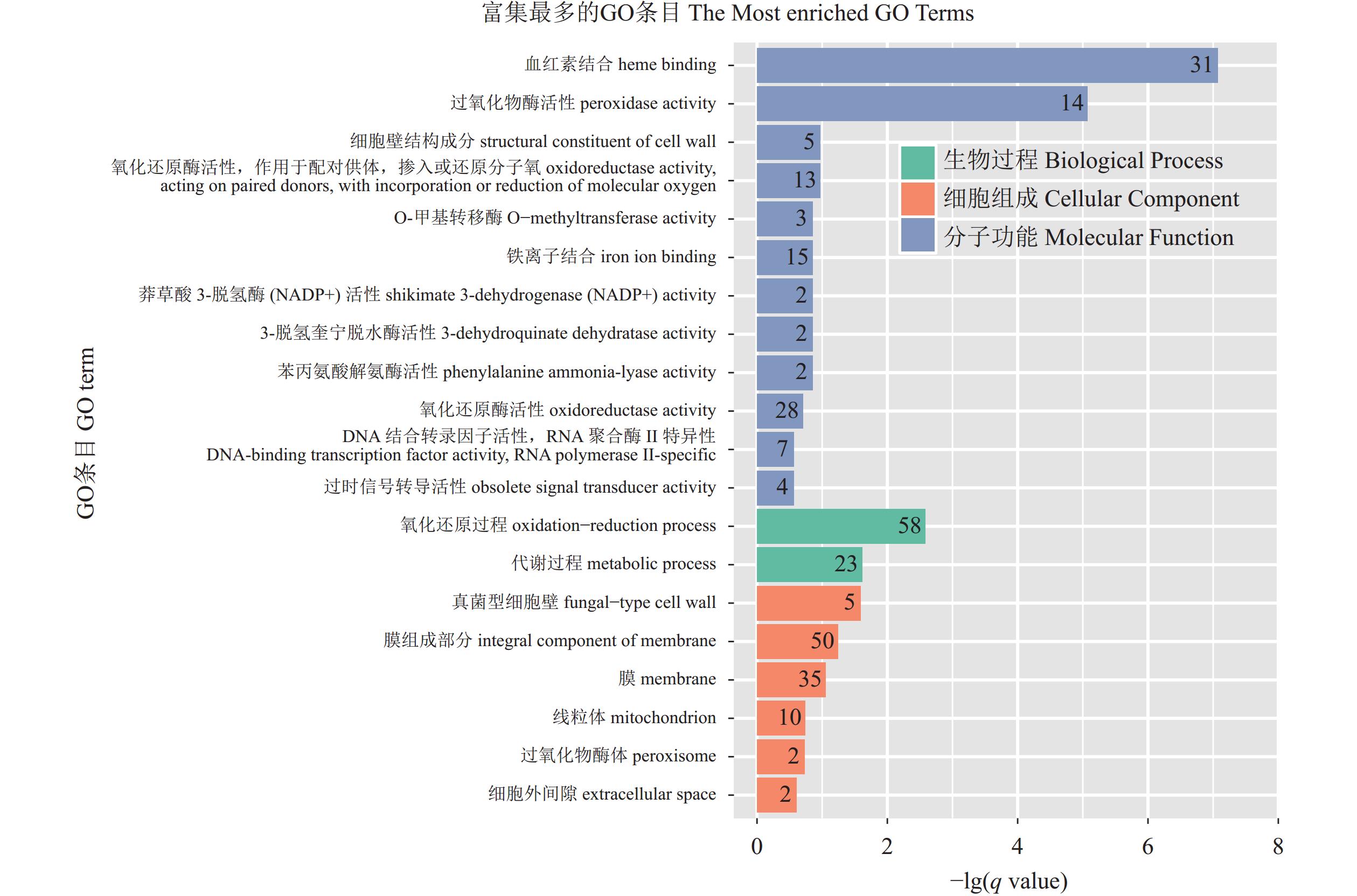
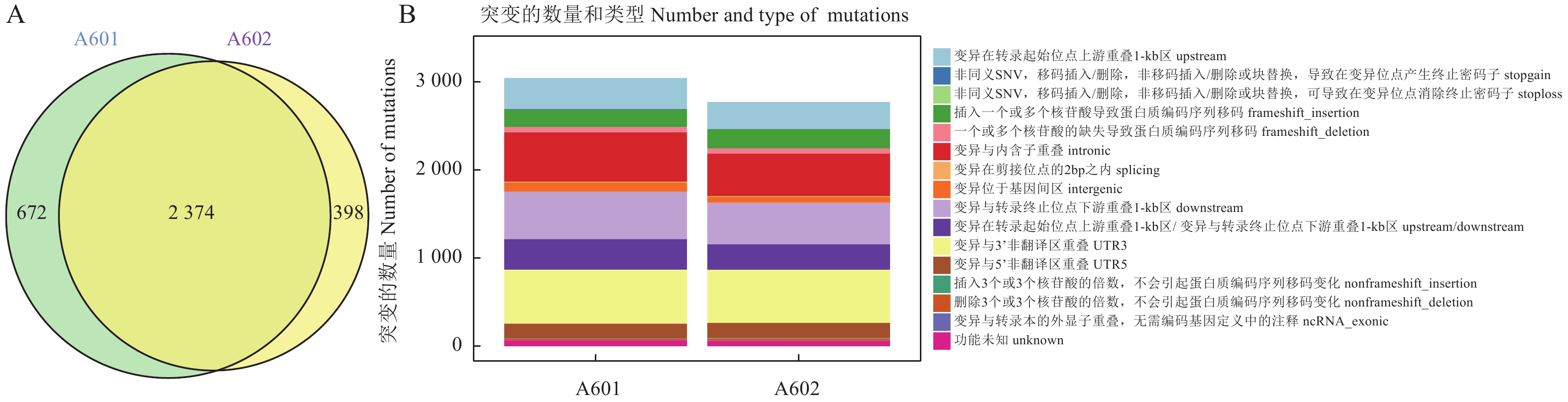
结果通过转录组分析,A601(As2796气生组)与A602(As2796贴生组)共鉴定出965个差异表达基因(DEGs)。GO富集分析结果表明,在真菌细胞壁组成方面有疏水蛋白相关的DEGs富集,其中AGABI2DRAFT_13655、AGABI2DRAFT_136569、AGABI2DRAFT_193057属于ABH3蛋白基因家族,在气生菌丝的表达水平显著高于贴生菌丝,可能与气生菌丝的产生有关。研究还发现了疏水表面结合蛋白A(HsbA)相关基因AGABI2DRAFT_194662在气生菌丝中FPKM表达水平为0,在贴生菌丝中为10.24。通过对DEGs进一步分析,富集到8个可能与脂酶同工酶相关的DEGs,气生菌丝中这些基因的表达水平显著高于贴生菌丝。SNP/InDel分析结果表明,As2796气生组SNP数为110 601,贴生组SNP数为111 188,单核苷酸变异中,C:G>T:A的变异次数较多,共筛选出2 374个共同InDel。
结论成功获得双孢蘑菇不同形态菌丝的转录组数据,并从中挖掘出潜在的相关基因和SNP位点,为双孢蘑菇相关分子标记开发及辅助育种提供参考。
Abstract:ObjectiveKey genes associated with Agaricus bisporus of different mycelial morphology were analyzed. [Methods] As 2796, the main cultivated variety of A. bisporus, with aerial or creepy hyphae grown on same plate were collected for RNA transcriptome sequencing and comparison with reference.
ResultsThere were 965 differentially expressed genes (DEGs) between the aerial hyphae-A601 and the creepy hyphae-A602. According to the GO enrichment analysis, the hydrophobic DEGs were enriched in the fungal cell wall, such as AGABI2DRAFT_13655, AGABI2DRAFT_136569, and AGABI2DRAFT_193057 that belonged to the ABH3 protein gene family, with expressions significantly higher in A601 than in A602. Thus, they were postulated to be related to the production of aerial hyphae. In addition, the hydrophobic surface binding protein A (HsbA)-related gene, AGABI2DRAFT_194662, showed a FPKM expression of 0 in A601, but 10.24 in A602. Furthermore, 8 enriched DEGs related to lipase isoenzymes exhibited a FPKM expression significantly higher in A601 than in A602. The SNP/InDel analysis indicated that the SNPs in A601 was 110 601 as opposed to 111 188 in A602, the most frequent mutation in SNPs was C:G>T:A, and that 2 374 common InDels were identified.
ConclusionThe transcriptomes and possibly associated genes and SNP sites in A. bisporus with different mycelial morphology obtained in this study would be of value for the mushroom germplasm evaluation and breeding.
-
0. 引言
【研究意义】棉花是我国的重要经济作物,2020年棉花产量位列全球第二,为591.0万t,种植面积约占全国耕地总面积的2.64%。中国是棉花消费大国也是进口大国,每年对棉花的需求量很大。氮肥对提高农作物产量起到重要作用,其产量贡献率为30%~50%[1]。大田生产投入过多的氮肥,部分氮肥通过NH3挥发损失和反硝化成为主要的氮素损失途径,造成资源浪费、农田土壤酸化[2]和地表水体富营养化[3]等负面影响。若施入较少的氮肥则影响作物后期的养分吸收与干物质积累,较低的生物量积累使作物产量也下降。因此,明确施氮对棉花产量的影响,分析不同农艺措施与不同土壤条件下施氮的产量效应和氮素吸收量效应的变化趋势,对于提高棉花产量具有重要意义。【前人研究进展】目前,我国已进行了大量有关施氮影响棉花产量的大田试验。李鹏程等[4]的研究表明,增施氮肥360 kg·hm−2时,产量提升698.9 kg·hm−2。同时,施氮的增产效益受多方面因素的影响。李景慧等[5]在新疆库尔勒市(试验土壤有机质含量为11.89 g·kg−1)试验比较了不同施氮量对棉花的产量影响,发现施氮量在384.9~430.4 kg·hm−2时,棉花产量最高。在河南安阳(试验土壤有机质含量为11.84 g·kg−1)种植密度与施氮量互作研究表明,种植密度为5.25 万株·hm−2、施氮量为225.0 kg·hm−2时棉花产量最高[6]。薛晓萍等[7]在两种不同土壤类型下种植棉花,在黏土下施氮量为240 kg·hm−2时,氮素吸收量最高;在砂壤土下施氮量为480 kg·hm−2时,氮素吸收量最高。这说明氮素合理施用可显著提高棉花产量[8-10],但是不同的土壤类型和不同土壤肥力下,施氮对棉花增产和氮素吸收量影响并不一致。Meta分析是整合一个主题对其独立研究进行定量结合和总结的统计方法[11],能够有效分析不同因素对试验目标的影响。【本研究切入点】独立田间试验研究难以解读较大区域施肥对产量和氮素吸收量的影响,且在全国尺度上研究施肥对棉花产量和氮素吸收量的影响及其关键因素的研究鲜有报道。【拟解决的关键问题】本研究以Meta分析为手段,从全国尺度上解读我国棉花种植时段、种植区域、种植密度、土壤类型及其土壤理化性质等条件下施氮对棉花产量和氮素吸收量的影响,以期为棉花氮肥高效利用与管理提供借鉴。
1. 材料与方法
1.1 数据来源
本研究所采用的文献数据来源于Web of Science、中国知网(CNKI)、万方和维普数据库,文献检索所用的关键词主要包括氮(Nitrogen)、施氮量(N application)、棉花(Cotton)、产量(Yield)、氮素吸收量(N uptake)等。为了提高整合分析结果的准确性和代表性,选用的文献必须满足以下标准:(1)试验地点必须在中国大陆区域范围内;(2)试验必须为田间试验,试验材料为棉花,田间试验必须有重复;(3)试验必须包含不施氮和施氮处理;(4)文献报道中至少包含产量和氮素吸收量中的一项;(5)文献报道中应包括试验组和对照组的平均值。文献中的数据有3种呈现方式:表格、折线图和柱状图;表格中的数据直接提取,折线图和柱状图用软件Web Plot Digitizer 4.4[12]将图形数值化后再提取。按照以上标准,筛选出184组棉花产量和103组氮素吸收量相关数据(产量均值、标准差和样本量)。
1.2 Meta分析
1.2.1 响应比及其方差计算
本研究选用响应比(Response ration,RR)为统计学指标,用响应比自然对数(lnRR)来评价施氮对棉花产量和氮素吸收量的影响程度[13],即施氮产量效应值和氮素吸收量效应。如果lnRR大于0,说明施氮对棉花产量和氮素吸收量有正效应,且lnRR值越大说明作用越明显;反之,则表示产生负效应。每项独立研究中响应比及其对数化的计算公式为:
RR=Xt/Xc (1) lnRR=ln(XtXc) (2) 式中,Xt和Xc分别表示处理组(施氮)和对照组(不施氮)的棉花产量和氮素吸收量平均值。lnRR的方差(V)计算公式为:
V=SD2tntX2t+SD2cncX2c (3) 式中,
SD2t 和SD2c 分别为处理组和对照组棉花产量和氮素吸收量的标准差,文献中SD缺失值,根据Andreas Gattingera的方法,采取相对应的试验组和对照组的1/10作为替代[14]。nt和nc分别为处理组和对照组的样本数。权重(Wij)的计算公式为:
Wij=1V (4) 加权响应比(RR++)的计算公式为:
RR++=m∑i=1ki∑j=1WijRRijm∑i=1ki∑j=1Wij (5) 式中, m是分组数(例如,不同的种植区域或种植密度等),ki是第i分组的总比较对数。
总体标准误计算公式为:
S(RR++)=√1m∑i=1ki∑j=1Wij (6) 式中,S(RR++)为总体标准误。
加权响应比(RR++)的95%置信区间计算公式为:
95%CI=RR++±1.96S(RR++) (7) 1.2.2 异质性与发表偏倚检验
本研究通过卡方检验(Chi-square test)来确定不同试验结果间是否存在异质性,并根据异质性情况来选择分析模型。若显著性检验结果为P>0.05,说明不同试验结果间无明显差异,可选择固定效应模型(Fixed effect model,FEM);若P<0.05,则说明不同试验结果存在明显异质性,应选择随机效应模型(Random effect model,REM)。
发表偏倚检验采用失安全系数法(Fail-safe number,Nfs),若Nfs>5n+10(n为数据量),则说明不存在发表偏倚,结论可信[15]。
1.2.3 加权综合响应比及其置信区间
因为纳入本研究的试验数据间存在明显的异质性,因此本研究选择随机效应模型,运用R软件中metafor包[16]来计算各研究的权重、加权综合响应比(RR++)及其95%置信区间(95%CI)。95%CI可用于RR++的假设检验:若95%CI下限>0,说明施氮对棉花的增产效应和氮素吸收效应显著;若95%CI上限<0,说明施氮对棉花产量和氮素吸收量存在显著的负效应;如果95%CI与0重叠,则说明施氮对棉花产量和氮素吸收无明显影响[13]。为更好地描述施氮对产量和氮素吸收量变化的影响,通过以下公式将RR++转换为变化百分率(E):
E=(eRR++−1)×100% (8) 1.2.4 亚组分析与Meta回归分析
为明确异质性来源与大小,分析不同条件下的施氮产量效应和氮素吸收量及其影响因素,本研究根据基础产量(不施氮处理的产量)、氮素吸收量(不施氮处理的氮素吸收量)、种植区域、种植年限、种植密度、土壤条件等特征因素进行亚组分析和Meta回归分析。根据我国种植区域(华中区、西北区、华东区)、种植时段(2002–2007、2008–2013、2014–2019)、土壤类型(砂壤土、壤土、黏土)、种植密度(<5万、5万~10万和>10万株·hm−2)、施氮量(<300、300~450、>450 kg·hm−2)、土壤有机质含量(<10、10~15、>15 g·kg−1)、土壤全氮含量(<1、1~1.6、>1.6 g·kg−1)、土壤碱解氮(<50、50~100、>100 g·kg−1)、土壤速效磷含量(<15、15~30、>30 mg·kg−1)、土壤速效钾含量(<200、200~300、>300 mg·kg−1)来探究其对棉花产量和氮素吸收量响应氮肥的影响。
1.3 统计分析
使用Excel 2019 进行数据合并、分类,使用R语言(4.0)计算相关数据,采用GraphPad Prism 8.0 软件进行图形绘制。
2. 结果与分析
2.1 施氮对棉花产量和氮素吸收量影响的Meta分析
由表1可知,棉花产量和氮素吸收量的失安全系数为196793、121726,5n+10为930、525,失安全系数远大于5n+10,说明本研究结果受发表偏倚的影响不明显,结论具有较高的可信度。由图1可知,施氮使棉花产量平均增加了32.22%(29.30%~38.80%),氮素吸收量平均增加了43.29%(38.80%~47.78%),说明氮肥的施用在不同程度上显著提高了棉花产量和氮素吸收量。
表 1 施氮影响棉花产量和氮素吸收量的Meta分析Table 1. Meta analysis on effects of N-fertilization on yield and N-uptake of cotton plants项目
Item样本量
Sample size显著性检验
Significance test95%置信区间下限
Bottom of 95% confidence interval95%置信区间上限
Top of 95% confidence interval失安全系数
Fail-safe number5n+10 产量 Yield 184 <0.01 0.2930 0.3514 196793 930 氮素吸收量 N uptake 103 <0.01 0.3880 0.4778 121726 525 2.2 棉花产量和氮素吸收量的影响因素
图2所示为不同种植时段、种植地区施氮对棉花产量和氮素吸收量的效应值。由图2可以看出,种植时段对施肥增加棉花产量的提高幅度具体体现为:2002–2007年提高了20%,2008—2013年提高了36%,2014–2019年提高了28%;施肥较不施肥处理对氮素吸收量的提高幅度具体体现为:2002—2007年提高了34%,2008–2013年提高了51%,2014–2019年提高了33%。
施氮显著提高了各个区域棉花产量(图2),且在不同区域间的增产效应和氮素吸收效应存在明显差异。其中,西北区的施氮产量增幅最高为45%,但西北区的氮素吸收量增幅最低为35%;华东区增幅分别为33%和38%,华中区产量增幅较低为25%,但华中区的氮素吸收量增幅最高为67%。
图3所示为不同土壤类型、种植密度对施氮棉花产量和氮素吸收量效应值影响。由图3可知,黏土施氮增产效果最高(40%),其次为壤土(36%)和砂壤土(25%)。就氮素吸收量而言,黏土氮素吸收效应最高(56%),壤土次之(39%),砂壤土最低(28%)。
种植密度显著影响棉花产量和氮素吸收量(图3)。当种植密度小于5 万株·hm−2时,施氮的产量增幅为40%,种植密度越大,棉花产量越低。对氮素吸收量而言,种植密度小于5 万株·hm−2时提高了62%,明显高于5万~10万株·hm−2(35%)和>10万株·hm−2(37%)的密度条件处理。
图4所示为不同施氮量、土壤有机质含量对棉花产量和氮素吸收量的效应值。由图4可知,施氮量在300~450 kg·hm−2时,施氮效果最显著,增产效应为38%,氮素吸收量提升了58%;其次为施氮量>450 kg·hm−2时,棉花产量涨幅为30%,氮素吸收量提升了54%;最后为施氮量<300 kg·hm−2时,棉花产量增长了27%,氮素吸收量增长了31%。
随着土壤有机质含量的增加,施肥较不施肥处理对棉花产量提高的幅度呈增高趋势,但氮素吸收量呈先升高再降低的趋势(图4),在有机质含量为10~15 g·kg−1时,氮素吸收量增幅为48%,明显高于有机质含量为>15 g·kg−1(40%)和<10 g·kg−1(21%)。
图5所示为不同土壤碱解氮含量、土壤全氮含量下施氮对棉花产量和氮素吸收量的效应值。由图5可知,在土壤碱解氮含量50~100 g·kg−1时施氮增产效应最高(37%),土壤碱解氮含量>100 g·kg−1时施氮的增产效应次之(34%),土壤碱解氮含量<50 g·kg−1时施氮的增产效应最低(32%)。不同于产量的增加趋势,在土壤碱解氮含量>100 g·kg−1(46%)和<50 g·kg−1(44%)时氮素吸收效应明显高于土壤速效氮含量50~100 g·kg−1(19%)时氮素吸收效应。
随着土壤全氮含量的增加,施肥较不施肥处理对棉花产量提高幅度差异不大,但氮素吸收量提高幅度差异较大(图5),土壤全氮含量为>1.6 g·kg−1、1~1.6 g·kg−1和<1 g·kg−1时增幅为51%、28%和44%。
图6所示为不同土壤速效磷、速效钾含量对施氮棉花产量和氮素吸收量的效应值。土壤速效磷含量15~30 mg·kg−1时增产效应最高(37%),>30 mg·kg−1的增产效应次之(36%),<15 mg·kg−1时增产效益最低(27%);与产量的增加趋势相同,土壤速效磷含量15~30 mg·kg−1时氮素吸收量增效最高(50%),土壤速效磷含量>30 mg·kg−1次之(44%),<15 mg·kg−1仅为21%。
土壤速效钾含量>300 mg·kg−1时增产效应为45%,明显高于速效钾含量200~300 mg·kg−1(22%)及<200 mg·kg−1(25%)的增产效应(图6)。随着土壤速效钾含量的升高,氮素吸收量呈先降低后增加的趋势。具体表现为:土壤速效钾含量为<200 mg·kg−1时提高的幅度为49%,200~300 mg·kg−1提高的幅度为26%,>300 mg·kg−1提高的幅度为39%。
2.3 棉花产量和氮素吸收量影响因素的异质性分析
表2所示为产量主要影响因素的Meta回归分析。从表2可看出,种植时间、种植区域、土壤类型、种植密度、施氮量、速效钾含量等因素引起的异质性较大,说明它们对施氮的增产效应存在明显差异且有显著影响;而土壤有机质含量、全氮含量、碱解氮含量、速效磷含量引起的异质性比较小且未达到显著水平,说明这4个因素对施氮的增产效果影响较小。
表 2 产量主要影响因素的Meta回归分析Table 2. Meta regression analysis on main factors affecting cotton yield产量影响因素
Factors affecting yield自由度
df异质性统计量
Qm显著性检验
PQM种植时段 Planting time 2 13.7413 <0.01 种植区域 Planting region 2 26.1813 <0.01 土壤类型 Soil type 2 21.3611 <0.01 种植密度 Planting density 2 35.0722 <0.01 施氮量 N application 2 11.8923 <0.01 有机质含量 SOM content 2 2.8682 0.2383 全氮含量 Total N content 2 0.0427 0.9789 碱解氮含量 Available N content 2 0.7656 0.6819 速效磷含量 Available P content 2 8.8255 0.0121 速效钾含量 Available K content 2 60.2263 <0.01 表3所示为氮素吸收量主要影响因素的Meta回归分析。从表3可看出,只有土壤全氮含量引起的异质性比较小且未达到显著水平,说明土壤全氮量对施氮效果影响较小。其余因素引起的异质性较大,说明它们对施氮的氮素吸收量效应密切相关且有显著影响。
表 3 氮素吸收量主要影响因素的Meta回归分析Table 3. Meta regression analysis on main factors affecting N-uptake of cotton plantse氮素吸收量影响因素
Factors affecting N uptake自由度
df异质性统计量
Qm显著性检验
PQM种植时段 Planting time 2 17.5224 <0.01 种植区域 Planting region 2 48.5255 <0.01 土壤类型 Soil type 2 13.0509 <0.01 种植密度 Planting density 2 36.2180 <0.01 施氮量 N application 2 33.7026 <0.01 有机质含量 SOM content 2 11.3464 <0.01 全氮含量Total N content 2 7.9870 0.0184 碱解氮含量 Available N content 2 10.1454 <0.01 速效磷含量 Available P content 2 9.9292 <0.01 速效钾含量 Available K content 2 10.4530 <0.01 3. 讨 论
3.1 施氮对棉花产量和氮素吸收量的整体影响分析
Meta分析结果表明,氮肥的施用可以显著提高我国棉花产量和氮素吸收量,增幅分别为32.22%和43.29%。董合林[17]研究认为棉田施用氮肥产量增长幅度为6.0%~114.7%。施肥增加作物产量的原因主要是施肥补充土壤养分的不足。棉花种植因生态区域、生态气候、栽培习惯不一致,产量差异较大[9],施氮显著提高了各种植区域棉花产量和氮素吸收量,棉花产量和氮素吸收量表现出不同的显著性。西北地区土壤较为贫瘠,土壤氮素供应能力较弱,施氮增加氮素养分供给,增产幅度较高。任意等[18]研究也表明西北地区因水土流失、降水稀少、气候干旱、土壤水分含量低等环境因素限定了养分的可利用性,所以施用的氮肥较难吸收并转化为产量,进而产量提升幅度较大,这与本文研究结果一致。
3.2 各类影响因素分析
就种植时段而言,不同试验时间的增产效应和氮素吸收量存在显著差异,近十年由于棉花品种改良、生产技术更新,较2008年以前棉花产量和氮素吸收量增幅较大。
就种植密度而言,娄善伟等[10]研究发现种植密度影响了棉株养分累积进而影响产量的形成。氮肥的施用与密度存在密切的交互作用[6]。本研究表明,种植密度对棉花施氮产量效应和氮素吸收量有显著影响,随着密度的增加,增产效应和氮素吸收量存在下降趋势。这与广大研究者研究结果一致[19]。
就施氮量而言,赵新华等[20]提出施氮量过低以至于棉铃干物质和氮的累积也较低。薛晓萍等[21]研究表明,棉花取得了较高的生物量和氮素累积量,但因为营养体内氮素积累量较多,营养生长较旺盛,干物质与养分向生殖器官转移量少,生殖器官比例下降,由此导致经济产量降低。这与本文研究结果一致,棉花产量随着施氮量的增加先升高后降低,氮素吸收与产量趋势相同。本文研究结果为当施氮量大于450 kg·hm−2时,施氮量对棉花产量增长幅度较小。养分含量较高易使棉花营养生长和生殖生长失调,同化物更多的去向茎叶分配,导致茎叶徒长,进而降低产量[22]。
施入土壤的养分能否被植物高效吸收利用与土壤类型和土壤理化性质关系密切[23]。本研究结果表明,壤土、砂壤土和黏土施氮均显著增加了棉花产量,其黏土施肥的增产效果高于砂壤土和壤土,这与徐国春等[24]的研究结果一致。在黏土条件下黏土的氮素吸收量的响应最高,这可能是由于黏土具有较好的保肥能力[25],且颗粒较小、养分吸附位点多[26]等原因。本研究结果表明在砂壤土条件下施氮对棉花产量和氮素吸收量的效应都较小,主要是因为砂壤土养分含量少,养分固定位点较少,施入的养分易随着水分淋失[27]。
土壤有机质作为衡量土壤肥力的重要指标,有机质含有土壤中80%以上的氮素含量,其影响氮素的供求关系[28]。本研究结果表明,有机质含量越高,施氮对棉花产量的效应越大。所以在有机质含量较低的土壤条件下,尽可能补充更多的外源氮素才能满足棉花高产的需求。刘德平等[29]研究指出氮磷有明显的互作关系,适宜的氮磷含量更好地提升作物产量。本研究结果表明土壤速效磷含量为15~30 mg·kg−1时,棉花产量最高。
3.3 Meta不确定性分析
因为Meta分析对数据要求较高,不足以纳入所有的相关文献,使本研究数据库的规模受到限制[25]。本研究没有考虑水肥一体化、测土配方等特殊施肥技术,也没有考虑到土壤质地、施肥方式、年降雨量和积温等对棉花产量和氮素吸收量的影响,没有涉及水分利用率,也未涉及水分管理模式对棉花产量和氮素吸收量的影响,同时由于不同文献中存在不同差异,这些都增加分析的不确定性。尽管如此,本研究结果依然在一定程度上阐明了施氮对我国棉花产量和氮素吸收量的影响,可以为棉花施氮效果提高和氮素营养综合管理提供借鉴。
4. 结论
利用Meta分析定量地研究了施氮肥对中国棉花产量和氮素吸收量的影响,结果显示施入氮肥对提高我国棉花产量和氮素吸收量有重要作用,施氮的平均增产效应为32.22%,氮素吸收量效应值平均增幅为43.29%。施氮肥对棉花产量的增产效应与种植时段、种植区域、土壤类型、种植密度、施氮量和土壤速效钾含量密切相关;施氮肥对棉花氮素吸收量的增长效应与种植时段、种植区域、土壤类型、种植密度、施氮量、土壤有机质含量、土壤碱解氮含量、土壤速效磷含量和土壤速效钾含量密切相关。
-
表 1 样品测序数据及与参考基因组的序列比对结果
Table 1 Statistics of experimental data and alignment with reference on genome sequences
编号
ID样品
Sample总Read数
Total clean reads比对到参考基因组的Reads数目
Mapped reads比对到参考基因组的Reads百分比
Mapped reads/%GC含量
GC content/%≥Q30的百分比
≥Q30/%A601 As2796气生菌丝 42 155 674 25 410 008 60.28 49.12 92.00 A602 As2796贴生菌丝 45 225 288 28 324 702 62.63 49.28 92.37 表 2 新基因功能注释结果统计
Table 2 Number of new genes with functional annotation
用于功能注释的数据库
Annotated database获得注释的新基因数目
Annotated gene number长度在≥300~ 1000 的新基因数目
Gene number (300≤gene length<1000 )长度≥ 1000 的新基因数目
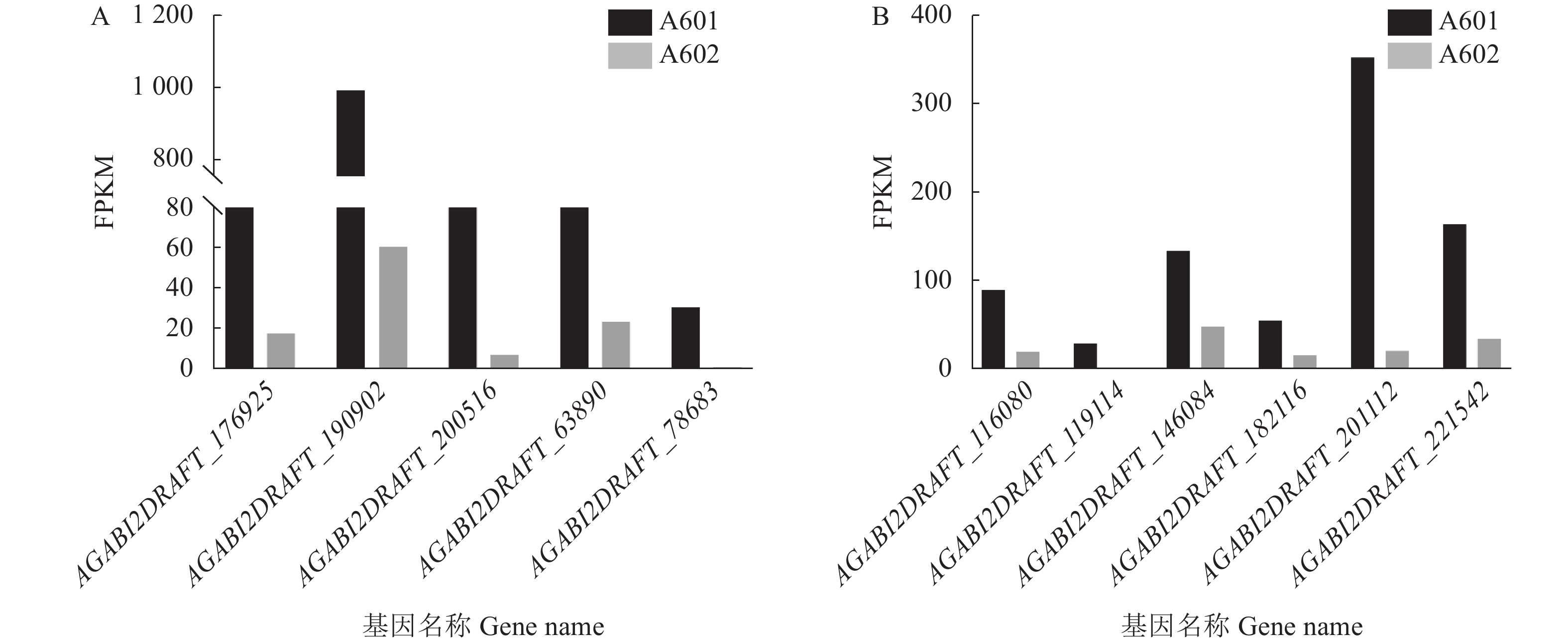
Gene number (gene length≥1000 )COG 15 12 3 GO 21 13 7 KEGG 38 21 14 KOG 20 12 8 Pfam 34 20 12 Swissprot 24 15 8 nr 132 70 57 All 139 76 57 表 3 部分差异表达基因
Table 3 Partial DEGs
编号
No.基因ID
Gene ID相对表达量FPKM
Relative expression FPKMLog2FC Pfam蛋白注释
Pfam protein annotation基因注释
Gene annotationA601 A602 1 AGABI2DRAFT_194497 15.6653 0.0000 −6.78 — — 2 AGABI2DRAFT_177536 20.5777 0.0000 −4.47 — 假设蛋白OH76DRAFT_1359727 3 AGABI2DRAFT_120985 1.0452 0.0000 −3.64 — 假设蛋白AN958_00580 4 AGABI2DRAFT_203489 2.3380 0.0001 −3.85 — 假设蛋白 AN958_02967 5 AGABI2DRAFT_44066 6.2585 0 −3.75 HhH-GPD超家族碱基
切除DNA修复蛋白假设蛋白 AN958_02739 6 AGABI2DRAFT_113583 0.0000 4.2489 4.73 — 假设蛋白 AN958_04089 7 AGABI2DRAFT_117617 0.0008 6.9437 3.30 — 假设蛋白 AN958_03520 8 AGABI2DRAFT_179131 0.0010 2.5569 4.28 — 假设蛋白 AN958_00324 9 AGABI2DRAFT_194662 0 10.2374 4.66 疏水表面结合蛋白A 假设蛋白AN958_09554 10 AGABI2DRAFT_136569 196.301 32.0293 −2.58 真菌疏水蛋白 假定的疏水蛋白 [双孢蘑菇] 11 AGABI2DRAFT_193057 999.26 189.163 −2.38 真菌疏水蛋白 假定的疏水蛋白 [双孢蘑菇] 12 AGABI2DRAFT_136557 19733 3086.17 −2.67 真菌疏水蛋白 假定的疏水蛋白 [双孢蘑菇] 13 AGABI2DRAFT_143464 100.031 896.356 3.21 真菌疏水蛋白 假定的疏水蛋白 [双孢蘑菇] 14 AGABI2DRAFT_178707 31.7555 666.848 4.35 真菌疏水蛋白 假定的疏水蛋白 [双孢蘑菇] 表 5 SNP突变统计表
Table 5 SNP mutation statistics
编号 ID T∶A>G∶C C∶G>G∶C T∶A>A∶T C∶G>T∶A C∶G>A∶T T∶A>C∶G 转化 Ts 颠换 Tv 转换/颠换比 ts/tv A601 6312 5221 7641 42864 6405 42158 85022 25579 3.32 A602 6276 5239 7586 43273 6338 42476 85749 25439 3.37 表 6 全基因组和编码区InDel长度分布表
Table 6 Length distribution of genome-wide and coding region InDel sites
编号
ID区域
Region−10 −9 −8 −7 −6 −5 −4 −3 −2 −1 1 2 3 4 5 6 7 8 9 10 A601 基因组
Genome11 2 0 0 1 2 0 6 106 1235 1559 121 1 0 0 2 0 0 0 0 A601 外显子
Exonic7 1 0 0 1 0 0 3 2 99 221 14 1 0 0 2 0 0 0 0 A602 基因组
Genome14 3 0 0 1 2 0 11 100 1080 1434 121 3 0 0 3 0 0 0 0 A602 外显子
Exonic9 1 0 0 1 0 0 7 2 100 229 15 3 0 0 3 0 0 0 0 -
[1] BERNAŚ E, JAWORSKA G. Vitamins profile as an indicator of the quality of frozen Agaricus bisporus mushrooms [J]. Journal of Food Composition and Analysis, 2016, 49: 1−8. DOI: 10.1016/j.jfca.2016.03.002
[2] OWAID M N, BARISH A, ALI SHARIATI M. Cultivation of Agaricus bisporus (button mushroom) and its usages in the biosynthesis of nanoparticles [J]. Open Agriculture, 2017, 2(1): 537−543. DOI: 10.1515/opag-2017-0056
[3] MUSZYŃSKA B, KAŁA K, ROJOWSKI J, et al. Composition and biological properties of Agaricus bisporus fruiting bodies- a review [J]. Polish Journal of Food and Nutrition Sciences, 2017, 67(3): 173−181. DOI: 10.1515/pjfns-2016-0032
[4] 王翠, 郭仲杰, 尤洁, 等. 双孢蘑菇产、质量性状相关分子标记的初步研究 [J]. 福建农业科技, 2018, 49(7):1−5. WANG C, GUO Z J, YOU J, et al. A preliminary study on molecular markers related to yield and quality traits of Agaricus bisporus [J]. Fujian Agricultural Science and Technology, 2018, 49(7): 1−5. (in Chinese)
[5] DAS B, DE B, CHETREE R, et al. Medicinal aspect of mushrooms: A view point[J]. Herbal Medicine in India: Indigenous Knowledge, Practice, Innovation and its Value, 2020: 509-532.
[6] ZHANG M, ZHAO L, TANG F, et al. Chemical structures, biological activities, and biosynthetic analysis of secondary metabolites from Agaricus mushrooms: A review [J]. Journal of Agricultural and Food Chemistry, 2024, 72(22): 12387−12397. DOI: 10.1021/acs.jafc.4c01861
[7] 柯斌榕, 兰清秀, 卢政辉, 等. 福建省双孢蘑菇栽培技术的变革与发展 [J]. 食药用菌, 2017, 25(1):12−19. KE B R, LAN Q X, LU Z H, et al. Innovation and development of Agaricus bisporus cultivation techniques in Fujian province [J]. Edible and Medicinal Mushrooms, 2017, 25(1): 12−19. (in Chinese)
[8] 林杰, 林莫翎. 蘑菇异型菌丝与栽培技术的相关性 [J]. 福建农业, 2007(11):20−21. LIN J, LIN M L. Correlation between heteromorphic hyphae of mushrooms and cultivation techniques [J]. Fujian Agriculture, 2007(11): 20−21. (in Chinese)
[9] 詹才新, 凌霞芬. 双孢蘑菇菌落形态和产质量性状间相关性研究 [J]. 食用菌学报, 1997, 4(3):7−12. ZHAN C X, LING X F. Studies on correlation between colonial morphology and characters of productivity and quality of Agaricus bisporus [J]. Acta Edulis Fungi, 1997, 4(3): 7−12. (in Chinese)
[10] 李洪荣. 双孢蘑菇不同菌落形态的同工酶分析 [J]. 福建轻纺, 2011(4):43−45. LI H R. Isozyme analysis of different colony morphology of Agaricus bisporus [J]. The Light & Textile Industries of Fujian, 2011(4): 43−45. (in Chinese)
[11] CAI Z X, CHEN M Y, LU Y P, et al. Metabolomics and transcriptomics unravel the mechanism of browning resistance in Agaricus bisporus [J]. PLoS One, 2022, 17(3): e0255765. DOI: 10.1371/journal.pone.0255765
[12] LU Y P, GUO Z J, KE B R, et al. Genome-wide association study and transcriptome analysis provide candidate genes for agronomic traits of Agaricus bisporus [J]. Horticulturae, 2024, 10(7): 691. DOI: 10.3390/horticulturae10070691
[13] YANG X M, YANG K X, WANG X H, et al. Transcriptomic analysis reveals the mechanism of bacterial disease resistance of postharvest button mushroom (Agaricus bisporus) [J]. Physiological and Molecular Plant Pathology, 2022, 122: 101903. DOI: 10.1016/j.pmpp.2022.101903
[14] 秦煜, 郑向丽, 林钟员. 基于转录组测序的红萍SSR和SNP特征分析 [J]. 福建农业科技, 2023, 54(6):44−48. QIN Y, ZHENG X L, LIN Z Y. Characteristic analysis of SSR and SNP in Azolla imbircata based on transcriptome sequencing [J]. Fujian Agricultural Science and Technology, 2023, 54(6): 44−48. (in Chinese)
[15] 陈美元. 双孢蘑菇As2796全长cDNA文库的构建及鉴定 [J]. 福建农业学报, 2012, 27(11):1201−1204. CHEN M Y. Construction and identification of full-length cDNA library of Agaricus bisporus As2796 [J]. Fujian Journal of Agricultural Sciences, 2012, 27(11): 1201−1204. (in Chinese)
[16] 王泽生. 蘑菇杂交株As2796的种性与栽培技术要点 [J]. 食用菌, 1993, 15(5):9. WANG Z S. Characteristics and cultivation techniques of mushroom hybrid strain As2796 [J]. Edible Fungi, 1993, 15(5): 9. (in Chinese)
[17] KIM D, PERTEA G, TRAPNELL C, et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions [J]. Genome Biology, 2013, 14(4): R36. DOI: 10.1186/gb-2013-14-4-r36
[18] 顾鑫, 杨晓贺, 姚亮亮, 等. 大豆灰斑病菌Race15的全基因组测序分析 [J]. 大豆科学, 2021, 40(4):466−475. GU X, YANG X H, YAO L L, et al. Whole-genome sequencing and analysis of Cercospora sojina race 15 [J]. Soybean Science, 2021, 40(4): 466−475. (in Chinese)
[19] JIANG H, WONG W H. Statistical inferences for isoform expression in RNA-Seq [J]. Bioinformatics, 2009, 25(8): 1026−1032. DOI: 10.1093/bioinformatics/btp113
[20] ANDERS S, HUBER W. Differential expression analysis for sequence count data[J]. Nature Precedings, 2010.
[21] MCKENNA A, HANNA M, BANKS E, et al. The Genome Analysis Toolkit: A MapReduce framework for analyzing next-generation DNA sequencing data [J]. Genome Research, 2010, 20(9): 1297−1303. DOI: 10.1101/gr.107524.110
[22] WANG K, LI M, HAKONARSON H. ANNOVAR: Functional annotation of genetic variants from next-generation sequencing data Nucleic Acids Research[J]. ANNOVAR: Functional annotation of genetic variants from next-generation sequencing data Nucleic Acids Research, 2010: 38.
[23] 王秉峰. 匍匐型与气生型双孢蘑菇菌株品比试验 [J]. 中国食用菌, 2010, 29(5):19−20. WANG B F. The comparison test of different strains of prostrate and aerial-type Agaricus bisporas [J]. Edible Fungi of China, 2010, 29(5): 19−20. (in Chinese)
[24] WANG H C, WANG Z S. The prediction of strain characteristecs of Agaricus bisporus by the application of isozyme electrophoresis [J]. Fujian Mushroom, 1991(1): 38−48. (in Chinese).
[25] WANG Z S, WANG H C. Isozyme patterns and characteristics of hybrid strains of Agaricus bisporus [J]. Microl. Neotrop. Apl, 1990(3): 19−29.
[26] 金建康, 徐鹤林. 双孢蘑菇气生型与匍匐型的比较研究 [J]. 中国食用菌, 1990, 9(5):3−4. JIN J K, XU H L. Comparative study on aerial type and creeping type of Agaricus bisporus [J]. Edible Fungi of China, 1990, 9(5): 3−4. (in Chinese)
[27] 杜炫星, 牛敏敏, 赵清, 等. 李白盾蚧转录组分析及 SSR 位点开发 [J]. 环境昆虫学报, 2023, 45(5):1318−1332. DU X X, NIU M M, ZHAO Q, et al. Analysis of the transcriptome and development of SSR Loci in Pseudaulacaspis prunicola (Maskell) [J]. Journal of Environmental Entomology, 2023, 45(5): 1318−1332. (in Chinese).
[28] MORIN E, KOHLER A, BAKER A R, et al. Genome sequence of the button mushroom Agaricus bisporus reveals mechanisms governing adaptation to a humic-rich ecological niche [J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(43): 17501−17506.
[29] LINDER M B, SZILVAY G R, NAKARI-SETÄLÄ T, et al. Hydrophobins: The protein-amphiphiles of filamentous fungi [J]. FEMS Microbiology Reviews, 2005, 29(5): 877−896. DOI: 10.1016/j.femsre.2005.01.004
[30] LUGONES L G, BOSSCHER J S, SCHOLTMEYER K, et al. An abundant hydrophobin (ABH1) forms hydrophobic rodlet layers in Agaricus bisporus fruiting bodies[J]. Microbiology, 1996, 142 ( Pt 5): 1321-1329.
[31] LUGONES L G, WÖS H A B, WESSELS J G H. A hydrophobin (ABH3) specifically secreted by vegetatively growing hyphae of Agaricus bisporus (common white button mushroom)[J]. Microbiology, 1998, 144 ( Pt 8): 2345-2353.
[32] OHTAKI S, MAEDA H, TAKAHASHI T, et al. Novel hydrophobic surface binding protein, HsbA, produced by Aspergillus oryzae [J]. Applied and Environmental Microbiology, 2006, 72(4): 2407−2413. DOI: 10.1128/AEM.72.4.2407-2413.2006
[33] 张烨. 球孢白僵菌疏水表面结合蛋白HsbA的克隆及功能研究[D]. 北京: 中国农业科学院, 2013 ZHANG Y. cDNA clone and functional research on hydrophobic surface binding protein HsbA in Beauveria bassiana [D]. Beijing: Chinese Academy of Agricultural Sciences, 2013. (in Chinese)
[34] PARK J, LEE S, CHOI J, et al. Fungal cytochrome P450 database [J]. BMC Genomics, 2008, 9: 402. DOI: 10.1186/1471-2164-9-402
[35] HLAVICA P. Evaluation of structural features in fungal cytochromes P450 predicted to rule catalytic diversification [J]. Biochimica et Biophysica Acta, 2013, 1834(1): 205−220. DOI: 10.1016/j.bbapap.2012.09.012
[36] SMIDA A D, VALDERRAMA X P, AGOSTINI M C, et al. Cadmium stimulates transcription of the cytochrome p450 side chain cleavage gene in genetically modified stable porcine granulosa cells [J]. Biology of Reproduction, 2004, 70(1): 25−31. DOI: 10.1095/biolreprod.103.019000
[37] ZHANG M, LIU X C, YUAN L Y, et al. Transcriptional profiling in cadmium-treated rice seedling roots using suppressive subtractive hybridization [J]. Plant Physiology and Biochemistry, 2012, 50: 79−86. DOI: 10.1016/j.plaphy.2011.07.015
[38] WANG L L, LI H B, WEI H L, et al. Identification of cadmium-induced Agaricus blazei genes through suppression subtractive hybridization [J]. Food and Chemical Toxicology, 2014, 63: 84−90. DOI: 10.1016/j.fct.2013.10.036
[39] 宋媛, 胡秋辉, 苏安祥, 等. 转录组学解析茉莉酸甲酯对双孢蘑菇个体大小的影响机制 [J]. 食品科学, 2021, 42(4):130−137. SONG Y, HU Q H, SU A X, et al. Transcriptomic analysis of the mechanism underlying the effect of methyl jasmonate on the size of Agaricus bisporus [J]. Food Science, 2021, 42(4): 130−137. (in Chinese)
[40] 刘俊敏. 镉胁迫对双孢蘑菇抗氧化系统的影响及耐受性相关基因的筛选与鉴定[D]. 厦门: 厦门大学, 2018. LIU J M. Cadmium stress on the effect of antioxidant system in Agaricus bisporus and the identification of differentially expressed genes [D]. Xiamen: Xiamen University, 2018. (in Chinese)
[41] XU K H, ZHAO Y, ZHAO Y, et al. Soybean F-box-like protein GmFBL144 interacts with small heat shock protein and negatively regulates plant drought stress tolerance [J]. Frontiers in Plant Science, 2022, 13: 823529. DOI: 10.3389/fpls.2022.823529
[42] MÜLLER-STARCK G. Isozymes[M]//Molecular Tools for Screening Biodiversity. Dordrecht: Springer Netherlands, 1998: 75-81.
[43] 时如霞, 曾琴, 赵丹. 基于楠木转录组的SSR、SNP、Indel分子标记技术特征分析 [J]. 山地农业生物学报, 2023, 42(4):83−87. SHI R X, ZENG Q, ZHAO D. SSR, SNP and InDel characterization of Phoebe zhennan based on the transcriptome sequence [J]. Journal of Mountain Agriculture and Biology, 2023, 42(4): 83−87. (in Chinese)
[44] 贾定洪, 王波, 何晓兰, 等. 基于基因组重测序发掘金针菇 InDel, SV 及丰度 SNP 标记 [J]. 菌物学报, 2024, 43(7):230228. JIA D H, WANG B, HE X L, et al. Exploitation of InDel, SV and abundance SNP markers in Flammulina filiformis based on genome resequencing [J]. Mycosystema, 2024, 43(7): 23022. (in Chinese).
[45] 施肖堃, 蔡志欣, 郭仲杰, 等. 18个双孢蘑菇核心种质的重测序初步分析 [J]. 福建农业学报, 2019, 34(10):1167−1172. SHI X K, CAI Z X, GUO Z J, et al. A preliminary report on resequencing 18 representative strains of Agaricus bisporus [J]. Fujian Journal of Agricultural Sciences, 2019, 34(10): 1167−1172. (in Chinese).
-
期刊类型引用(3)
1. 王嘉琳,张世柏,王涛,赵竹青,石磊. 我国油菜、大豆和烤烟硼肥增产提质的整合分析. 中国土壤与肥料. 2024(12): 176-184 .  百度学术
百度学术
2. 朱盼盼,石磊. 我国主要粮食作物施用锌肥增产效果的整合分析. 中国土壤与肥料. 2023(06): 213-218 .  百度学术
百度学术
3. 程艳莉,张芬,刘发波,方林发,孙铭,梁涛,刘敦一,陈新平,王孝忠. 滴灌施肥对不同类型蔬菜和果树产量、水氮利用效率和品质的影响. 植物营养与肥料学报. 2023(09): 1677-1688 .  百度学术
百度学术
其他类型引用(3)














 下载:
下载: