Vector Construction and Immunogenicity of S and N Gene DNA Vaccine for TGEV
-
摘要:目的
构建猪传染性胃肠炎病毒(transmissible gastroenteritis virus, TGEV)S、N基因的DNA疫苗载体,并进行免疫原性试验,为猪传染性胃肠炎(transmissible gastroenteritis, TGE)的防控和DNA疫苗研究提供技术支撑和基础数据。
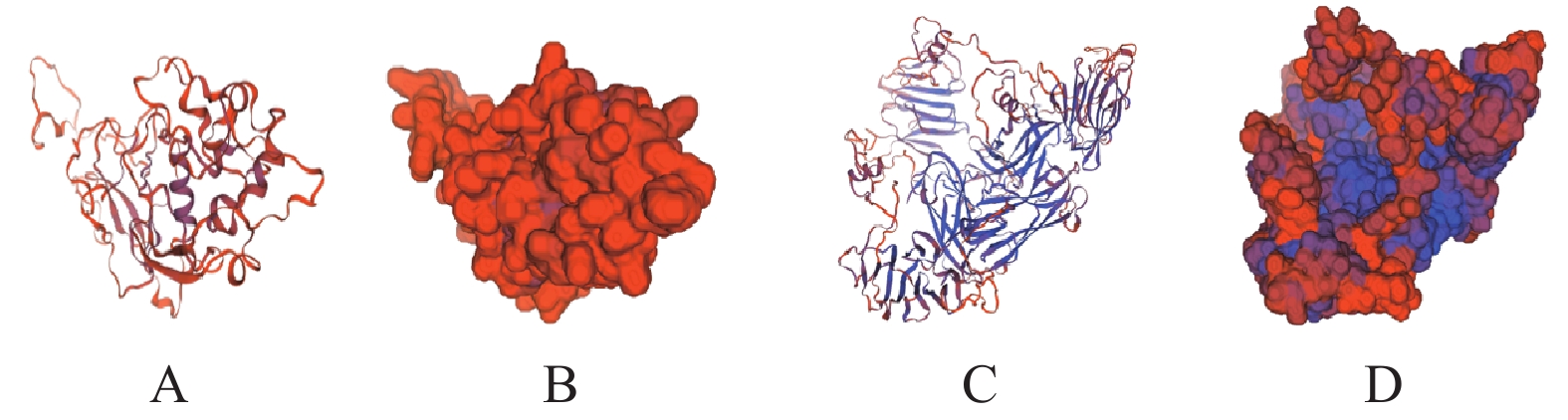
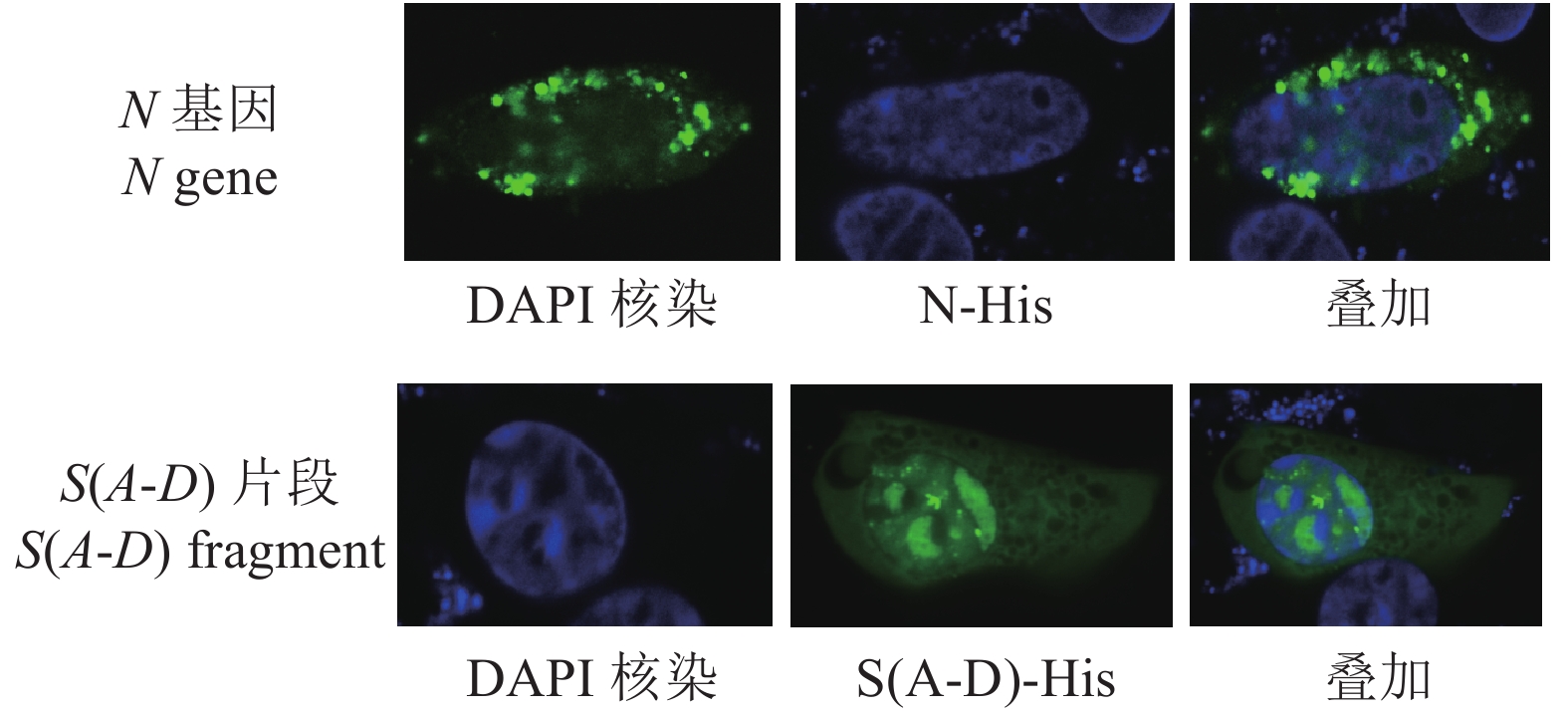
方法扩增S基因的A位点、D位点和N基因,并将N基因(单独)、A位点和D位点(融合)克隆至pCDNA3.1-His-C构建重组疫苗载体,运用生物信息学软件预测分析S(A-D)蛋白、N蛋白二级结构组成、三级构像、亚细胞定位和优势B细胞抗原表位。将构建成功的重组载体分别转染至PK-15细胞进行间接免疫荧光试验,运用共聚焦检测重组蛋白的表达分布情况。将重组疫苗载体单独或联合免疫小鼠,运用间接ELISA检测IgG抗体水平。
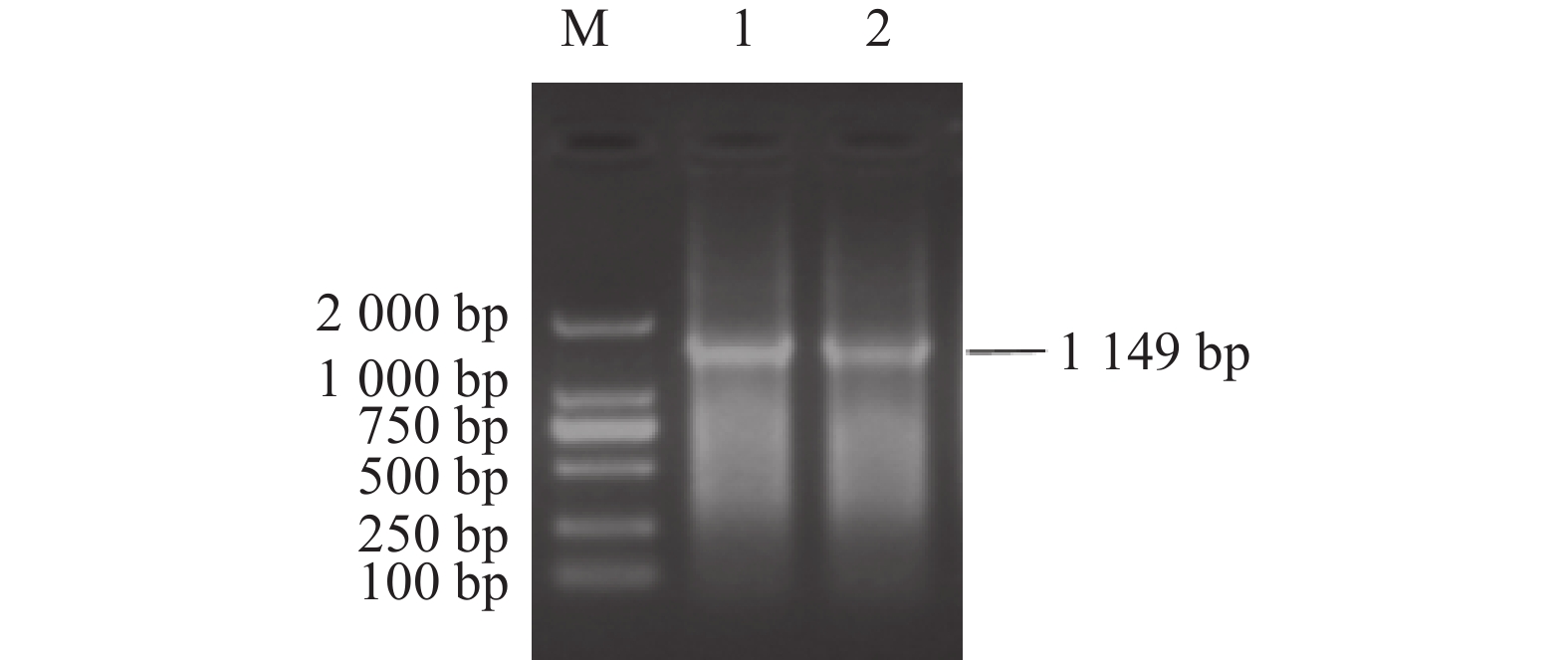
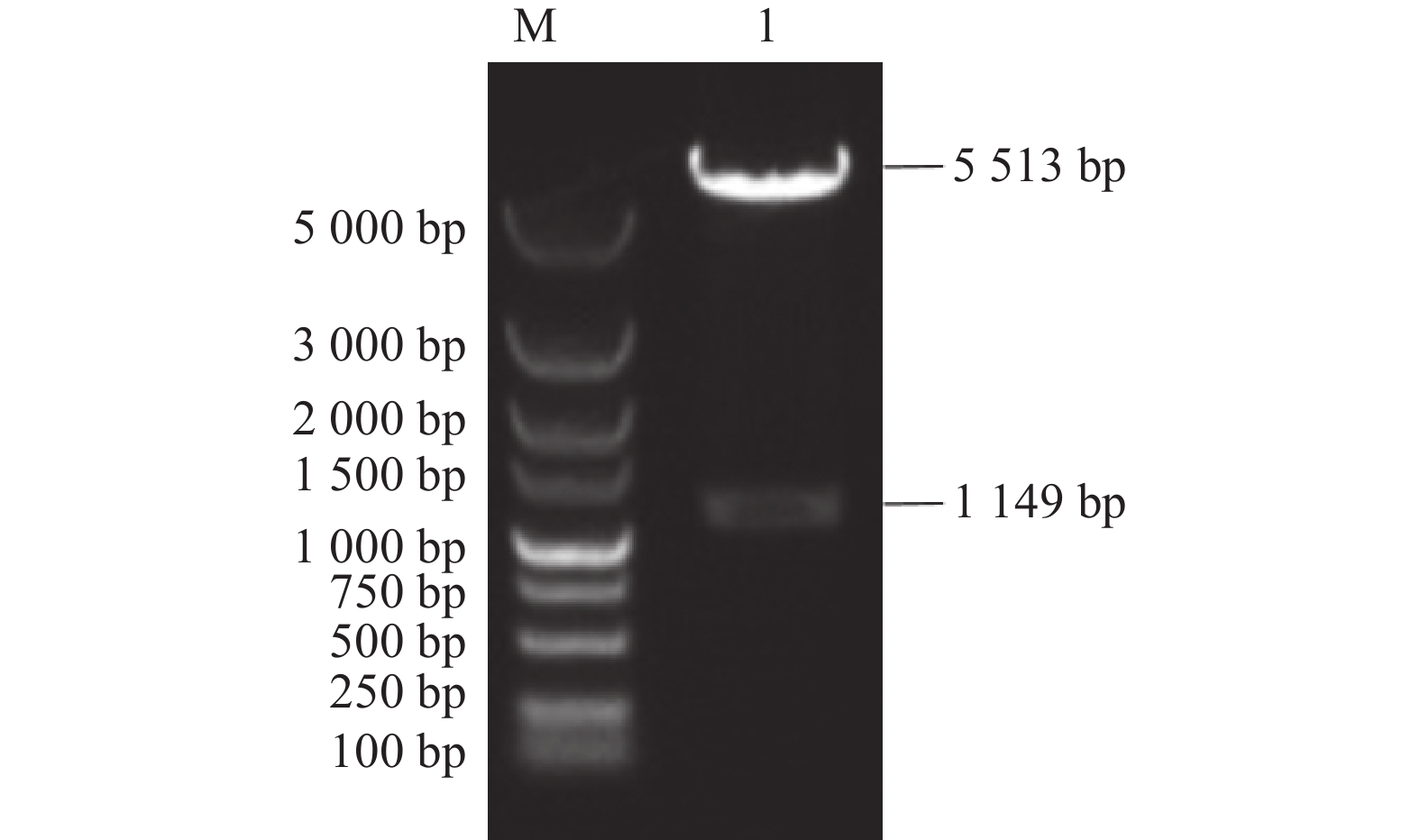
结果扩增出S基因的A位点、D位点和N基因,大小分别为498、606、
1149 bp。构建了A位点与D位点(融合)、N基因(单独)的DNA疫苗重组载体p-S(A-D)-His和p-N-His。生物信息学软件预测分析发现TGEV感染宿主细胞时N蛋白主要定位于细胞核和线粒体,S(A-D)蛋白主要定位于细胞质和线粒体,S(A-D)蛋白具有7个优势B细胞抗原表位,N蛋白具有8个优势B细胞抗原表位。重组载体p-S(A-D)-His和p-N-His均在PK-15细胞内成功表达,且S(A-D)-His和N-His在PK-15细胞核和细胞质中均有分布。重组疫苗载体免疫小鼠后,免疫效果由高至低依次为p-N-His>p-S(A-D)-His + p-N-His>p-S(A-D)-His。结论本研究构建了TGEV的 S、N基因的DNA疫苗载体,免疫小鼠后均产生了较强的特异性抗体,为TGEV的核酸疫苗的研制提供了基础材料和依据。
Abstract:ObjectiveDNA vaccine vector of S and N genes of porcine transmissible gastroenteritis virus (TGEV) was constructed with the vaccine immunogenicity determined to pave the way for studying, preventing, and controling TGE.
MethodA and D sites on S and N from a TGEV were amplified. The N gene alone as well as the A and D sites fusion were cloned into the vaccine vector pCDNA3.1-His-C. Bioinformatics software was used to predict and analyze the secondary structure, tertiary configuration, subcellular localization, and dominant B cell epitope of S (A-D) and N proteins. The recombinant vectors were transfected into PK-15 cells, and expression distribution of N and the A and D sites fusion detected by indirect immunofluorescence and confocal detection. Mice were immunized with the single or combined recombinant vaccine vector to detect the IgG antibody using indirect ELISA.
ResultThe A and D sites of the S were 498 bp and 606 bp, respectively, and the N,
1149 bp in length. The nucleic acid vaccine expression vectors p-S (A-D)-His and p-N-His for the A and D sites (fusion) and N were constructed. Bioinformatics software predicted that, when TGEV infected the host cells, N protein was mainly located in the nucleus and mitochondria and S (A-D) largely in the cytoplasm and mitochondria, while S (A-D) had 7 and N, 8 dominant B cell epitopes. All p-S (A-D)-His and p-N-His were successfully expressed in PK-15 cells distributed in the nucleus and cytoplasm. The immunized mice showed an effect of immunity in the order of p-N-His>p-S (A-D)-His + p-N-His>p-S (A-D)-His.ConclusionThe DNA vaccine vectors of S and N of TGEV were successfully constructed. Strong specific antibodies were generated in lab mice after the immunization.
-
0. 引言
【研究意义】秀珍菇学名肺形侧耳[Pleurotus pulmonarius(Fr.)Quél.],因朵型小巧,又名袖珍菇,是中温型结实食用菌[1]。秀珍菇不仅营养丰富,富含人体必需氨基酸、蛋白质等,风味独特[2-6],还具有抗氧化、抗肿瘤、降血压、降血糖和提高免疫力等功能[7-10]。与平菇(糙皮侧耳)相比,因采收时成熟度小,质地更为鲜嫩,口感更为滑嫩,深受消费者喜爱。木霉是秀珍菇栽培上的重要竞争性杂菌,其主要与秀珍菇菌丝竞争营养,由于木霉生长快,繁殖能力强,繁殖系数高,一旦出现,极易爆发,严重时导致大量菌包废弃,造成较大经济损失[11-12]。研究秀珍菇不同菌株适宜生长条件,阐明秀珍菇与木霉共培养特性,对秀珍菇栽培菌种的选择及温度管理具有重要指导意义。【前人研究进展】秀珍菇自1998年由我国台湾地区引入大陆,并在福建罗源试种150万袋获得成功后,在福建、浙江、河南、山东、安徽、江苏等地开始大面积栽培[13-16]。秀珍菇栽培地区和面积的增加也逐渐提高了产量,2019年全国秀珍菇产量超过30万t,较上一年增长20.27%[17]。目前我国虽然已认定了多个秀珍菇品种,但许多栽培者偏好于 “台秀”57菌株[18-19]。温度是食用菌正常生长的必要条件,主要影响食用菌菌丝的生长速度,以及子实体的分化数量和质量[20]。培养基质为食用菌的生长提供必需的营养物质,培养基质是否适宜决定了菌株菌丝和子实体的质量。秀珍菇常用PDA培养基培养,菌丝生长良好[21]。冯志勇等研究发现,秀珍菇菌丝生长的最适碳源、氮源组合是酵母粉和可溶性淀粉,菌丝粗壮,生长速度快[22]。【本研究切入点】采用传统方式栽培,秀珍菇菌性变化明显,在生产上易引起菌棒满菌后退菌、开袋出菇少,甚至不出菇、易感染霉菌等现象,导致秀珍菇栽培的不稳定性,使生产者遭受经济损失。2015–2018年全国秀珍菇产值曾一度下降[23]。据栽培者经验判断,产生这些问题的主要原因可能是菌种退化,导致秀珍菇菌种退化以及秀珍菇菌丝易受木霉病菌侵染的原因有待深入研究。【拟解决的关键问题】旨从温度和培养基质方面着手,观察秀珍菇菌丝在不同培养基和温度条件下,菌丝形态和生长情况。筛选出秀珍菇菌丝生长的适宜温度和培养基条件;探寻秀珍菇与木霉菌丝适宜生长的温度差异,为秀珍菇科学栽培提供理论支撑。
1. 材料与方法
1.1 试验材料
1.1.1 供试菌株
X98ly-13、Y710-14、Xd-13菌株保存于福建省农业科学院食用菌研究所,X903-1与X86-1是多孢分离获得的新材料,亲本分别为X903与X86。木霉菌株(T001)分离自秀珍菇菌包,并经形态学鉴定为哈茨木霉(Trichoderma harzianum)。
1.1.2 培养基配方
马铃薯琼脂葡萄糖培养基(PDA):马铃薯(200 g)、琼脂粉(20 g)、葡萄糖(20 g)、水(1 000 mL)。马铃薯琼脂葡萄糖+淀粉培养基(PDSA):马铃薯(200 g)、琼脂粉(20 g)、葡萄糖(20 g)、可溶性淀粉(5 g)、水(1 000 mL)。 马铃薯琼脂葡萄糖+酵母培养基(PDYA):马铃薯(200 g)、琼脂粉(20 g)、葡萄糖(20 g)、酵母粉(5 g)、水(1 000 mL)。马铃薯琼脂葡萄糖+淀粉+酵母培养基(PDSYA):马铃薯(200 g)、琼脂粉(20 g)、葡萄糖(20 g)、酵母粉(2.5 g)、可溶性淀粉(2.5 g)、水(1 000 mL)。
1.2 试验方法
1.2.1 不同温度对秀珍菇和木霉菌丝生长的影响
将秀珍菇和木霉菌丝块接种在PDA培养基质中,分别放在不同温度条件(25、28、30、32、34、36 ℃)的培养箱中,记录萌发时间,平板菌丝生长速度以平板中菌落半径日增长量计算。
1.2.2 PDA与半合成培养基对秀珍菇菌丝生长的影响
将秀珍菇菌株分别接种在PDA、PDSA、PDYA和PDSYA培养基,记录萌发时间,菌丝生长速度以平板中菌落半径日增长直径计算。
1.2.3 秀珍菇与木霉共培养
将秀珍菇和木霉菌块分别接在距培养皿边缘2 cm的两端 ,每个处理重复3次,标上菌株号,在不同温度的恒温箱中培养,定期观察。
1.3 数据分析
通过Excel 2007和DPS 7.05,对数据进行记录和差异显著性分析。
2. 结果与分析
2.1 温度对秀珍菇菌丝生长及与木霉共培养的影响
秀珍菇菌株在不同温度条件下的生长速度情况见表1。5个菌株在25、28、30 ℃萌发时间均为24 h。当温度超过30 ℃,随着温度逐渐升高,萌发时间逐渐加长。34 ℃ 5个菌株的萌发时间均为72 h以上。X98ly-13、Xd-13、903-1、86-1等4个菌株在25、28、30 ℃菌丝生长速度无显著性差异,但与30和32 ℃有极显著差异。Y710-14菌株菌丝生长最快的温度为30 ℃,与其他4个温度有极显著差异。从满皿时间来看,菌丝最快满皿的菌株为Xd-13,在25、28 ℃,均为5 d。Y710-14、Xd-13菌丝在30 ℃,均为5.33 d。86-1菌株菌丝生长最慢,在32、34 ℃,满皿时间均超过20 d。36 ℃时,秀珍菇菌丝长期处于萌发状态,生长非常缓慢。
表 1 温度对秀珍菇菌丝生长速度的影响Table 1. Effects of temperature on mycelial growth of P. pulmonarius菌株
Strain温度
Temperature/
℃萌发时间
Germination
time/h菌丝生长速度
The growth rate of
mycelia
/(mm·d−1)满皿时间
Full petri
dish time/dX98ly-13 25 24 8.63±0.01 Aa 6 28 24 8.25±0.01 Aab 6 30 24 7.96±0.05 ABbc 6 32 24 7.50±0.01 Bc 6.33 34 72 0.92±0.02 Cd >15 Y710-14 25 24 7.42±0.06 Bc 6.67 28 24 8.00±0.03 Bb 6 30 24 8.83±0.01 Aa 5.33 32 24~48 6.46±0.01 Cd 8.33 34 72 0.83±0.03 De >15 Xd-13 25 24 8.88±0.02 Aa 5 28 24 7.63±0.01 Aa 5 30 24 8.38±0.03 ABb 5.33 32 24~48 7.75±0.03 Bc 6.67 34 72 0.92±0.01 Cd >15 903-1 25 24 7.86±0.08 Aa 6 28 24 7.71±0.01 Aa 6 30 24 7.71±0.02 Aa 6 32 24~48 6.29±0.06 Bb 7.67 34 72 0.67±0.01 Cc >15 86-1 25 24 5.38±0.04 Aa 9 28 24 4.46±0.09 ABa 10 30 24 4.38±0.09 ABa 11.33 32 24~48 3.08±0.05 Bb 20.33 34 72 0.46±0.01 Cc >21 注:表中不同大小写字母表示同一菌株在不同温度条件下菌丝生长速度有极显著差异(P<0.01)和显著差异(P<0.05)。
Note: Data with different uppercase and lowercase letters on same column indicate significant differences at P<0.01 and P<0.05, respectively.试验结果表明,木霉在不同温度下菌丝的生长速度有差异(表2)。6种温度条件,25~32 ℃,木霉菌丝生长势旺盛,其中28 ℃木霉菌丝生长最快,约2 d长满90 mm培养皿,随着温度升高,菌丝生长速度变慢,34 ℃,木霉菌丝生长速度明显减弱,约3 d长满90 mm培养皿。36 ℃,木霉菌丝生长非常缓慢,菌丝泛黄,生长势最弱。同时测定了Xd-13菌丝、木霉菌丝在不同温度下的生长情况及秀珍菇菌丝与木霉菌丝的共培养情况(图1)。结果表明,在相同条件下,木霉菌丝的生长速度与秀珍菇菌丝的生长速度有极显著差异,秀珍菇菌丝的生长速度明显慢于木霉菌丝,且秀珍菇菌丝对木霉菌丝无拮抗现象,秀珍菇菌丝很快被完全覆盖,失去竞争力。
表 2 木霉在不同温度条件下菌丝生长情况Table 2. Mycelial growth of T. harzianum at different temperatures温度
Temperature/
℃萌发时间
Germination
time/h菌丝生长速度
Growth rate of mycelia/
(mm·d−1)满皿
时间
Full petri
dish time/d覆盖秀珍菇
菌丝时间
Infect mycelia of
Pleurotus
pulmonarius
time/h25 <24 19.33±0.03 Bb 2.21 48 28 <24 20.5±0.05 Aa 2.21 48 30 <24 19.33±0.03 Bb 2.5 48 32 24 18.5±0.05 Bc 3 48~54 34 24~48 14.67±0.03 Cd 3 54~72 36 72 3.17±0.03 De / / 注:表中不同大小写字母表示菌株在不同温度条件下菌丝生长速度有极显著差异(P<0.01)和显著差异(P<0.05)。
Note: Data with different uppercase and lowercase letters on same column indicate significant differences at P<0.01 and P<0.05, respectively.![]() 图 1 秀珍菇(XD-13)木霉(T001)菌丝在不同温度条件下生长及共培养情况注:①A:秀珍菇菌丝;B:木霉菌丝;C:秀珍菇和木霉菌丝共培养;②图中平皿从左到右处理温度依次为25、28、32 ℃(上排),34、36 ℃(下排)。Figure 1. Mycelial growth of P. pulmonarius (XD-13) and T. harzianum (T001) in cocultivation at different temperaturesNote: ① A: mycelia of P. pulmonarius; B: mycelia of T. harzianum; C: mycelia of P. pulmonarius and T. harzianum. ② Plate temperatures from left to right at 25 ℃, 28 ℃, and 32 ℃ on upper row; 34 ℃ and 36 ℃ on bottom row.
图 1 秀珍菇(XD-13)木霉(T001)菌丝在不同温度条件下生长及共培养情况注:①A:秀珍菇菌丝;B:木霉菌丝;C:秀珍菇和木霉菌丝共培养;②图中平皿从左到右处理温度依次为25、28、32 ℃(上排),34、36 ℃(下排)。Figure 1. Mycelial growth of P. pulmonarius (XD-13) and T. harzianum (T001) in cocultivation at different temperaturesNote: ① A: mycelia of P. pulmonarius; B: mycelia of T. harzianum; C: mycelia of P. pulmonarius and T. harzianum. ② Plate temperatures from left to right at 25 ℃, 28 ℃, and 32 ℃ on upper row; 34 ℃ and 36 ℃ on bottom row.2.2 培养基对秀珍菇菌丝生长的影响
温度为25 ℃时,5个秀珍菇菌株在四种培养基上的生长速度均无显著性差异,萌发时间均为24 h,生长情况见图2。X98ly-13菌株在PDYA培养基中的生长速度稍快于PDSYA培养基,在PDSA培养基中菌丝生长速度最慢。添加淀粉或酵母粉的培养基均可促进Y710-14、Xd-13和903-1菌株菌丝的生长,其中PDYA培养基对Y710-14菌丝日生长速度最快,PDSA培养基对Xd-13菌丝生长促进作用最明显,PDSYA培养基对903-1菌株菌丝的促进作用最大。5个秀珍菇菌株在PDYA培养基中的生长势最强,其次为PDSYA培养基,PDSA和PDA培养基中的菌丝生长势正常。加淀粉时秀珍菇菌丝普遍比正常菌丝更白,呈现乳白色。
![]() 图 2 5个菌株在不同培养基条件下菌丝的生长情况注:①A:X98ly-13菌株;B:Y710-14菌株;C:XD-13菌株;D:903-1菌株;E:86-1菌株。②平皿从左至右依次为PDA、PDSA、PDYA、PDSYA培养基。Figure 2. Mycelial growth of 5 strains of P. pulmonarius cultured on different mediaNote: ①A:X98ly-13; B: Y710-14; C: XD-13; D: 903-1; E: 86-1. ② Plates from left to right represent culture media PDA, PDSA, PDYA, and PDSYA.
图 2 5个菌株在不同培养基条件下菌丝的生长情况注:①A:X98ly-13菌株;B:Y710-14菌株;C:XD-13菌株;D:903-1菌株;E:86-1菌株。②平皿从左至右依次为PDA、PDSA、PDYA、PDSYA培养基。Figure 2. Mycelial growth of 5 strains of P. pulmonarius cultured on different mediaNote: ①A:X98ly-13; B: Y710-14; C: XD-13; D: 903-1; E: 86-1. ② Plates from left to right represent culture media PDA, PDSA, PDYA, and PDSYA.25 ℃条件下,秀珍菇不同菌株在4种培养基中菌丝的生长速度情况如表3所示,Xd-13菌丝生长速度均最快;86-1菌株生长速度最慢,与其他4个菌株的菌丝生长速度有极显著差异。PDSA培养基条件下,Y710-14菌株菌丝生长速度与Xd-13和X98ly-13无显著性差异,与903-1菌株有极显著差异。PDA和PDSYA培养基条件下,X98ly-13菌株菌丝的生长速度稍快于Y710-14菌株,PDSA和PDYA培养基条件下,X98ly-13菌株菌丝的生长速度稍慢于 Y710-14菌株,但它们之间均无显著性差异。
表 3 秀珍菇不同菌株在4种培养基中的菌丝生长情况分析Table 3. Mycelial growth of 5 strains of P. pulmonarius on 4 different culture media培养基
Culture
medium菌株
Strain菌丝生长速度
The growth rate
of mycelia/
(mm·d−1)菌丝特征
Mycelia characteristicsPDA X98ly-13 8.42±0.38 Aa 生长速度快、粗壮浓密、洁白 Y710-14 8.05±1.04 Aa 生长速度较快、浓密、洁白 XD-13 8.67±0.90 Aa 生长速度快、气生菌丝多、洁白 903-1 7.72±0.36 Aa 生长速度中等、较密、洁白 86-1 6.06±0.88 Bb 生长速度慢、粗壮浓密、洁白 PDSA X98ly-13 7.78±1.20 Bb 生长速度快、粗壮浓密、乳白 Y710-14 8.44±0.46 ABab 生长速度快、浓密、乳白 XD-13 9.28±0.51 Aa 生长速度快、气生菌丝多、乳白 903-1 7.56±0.58 Bb 生长速度中等、较密、乳白 86-1 6.00±0.56 Cc 生长速度慢、粗壮浓密、乳白 PDYA X98ly-13 8.58±0.66 Aab 生长速度快、粗壮浓密、洁白 Y710-14 8.78±0.75 Aa 生长速度快、浓密、洁白 XD-13 8.89±0.82 Aa 生长速度快、气生菌丝多、洁白 903-1 7.83±0.35 Ab 生长速度正常、较密、洁白 86-1 6.22±0.62 Bc 生长速度慢、粗壮浓密、洁白 PDSYA X98ly-13 8.22±0.71 Aa 生长速度快、粗壮浓密、洁白 Y710-14 8.11±0.49 Aa 生长速度快、浓密、洁白 XD-13 8.75±0.69 Aa 生长速度快、气生菌丝多、洁白 903-1 8.05±0.81 Aa 生长速度正常、较密、洁白 86-1 6.72±0.36 Bb 生长速度慢、粗壮浓密、洁白 注:表中不同大、小写字母表示不同菌株在相同培养基条件下菌丝生长速度有极显著差异(P<0.01)和显著差异(P<0.05)。
Note: Means within a column followed by different uppercase and lowercase letters indicate significant difference at P<0.01 or P<0.05, respectively.3. 讨论与结论
不同温度对5种秀珍菇菌株的菌丝生长均有不同程度的影响,同一温度条件,不同菌株生长速度有差异。其中4个菌株菌丝适宜生长温度范围均为25~30 ℃,在此温度范围菌丝生长速度无显著性差异,但与32 ℃和34 ℃条件有极显著差异,最适生长温度为25 ℃,这与王再明等的研究结果一致[19, 20],Y710-14菌株菌丝最适生长温度为30 ℃。试验结果表明,5个菌株在32 ℃,菌丝生长速度变慢,34 ℃秀珍菇菌丝生长非常缓慢。可见,秀珍菇菌株菌丝正常生长的临界温度介于32~34 ℃。这为秀珍菇生产上菌包包心温度的控制提供重要参考。Y710-14菌株菌丝最适生长温度较高,推断其应为中温偏高型菌株。秀珍菇常规栽培的品种为中温型菌株,为适应夏季规模化生产,雷潇等筛选了适宜湖南夏季设施化栽培的秀珍菇优良菌株26和秀58高温品种[24]。可见,不同秀珍菇菌株菌丝生长对温度要求有差异,温度是影响秀珍菇菌丝生长的重要因子,在生产上应根据地区气候条件和种植模式情况,选择适宜的秀珍菇菌株。秀珍菇菌丝生长速度的快慢除了与温度有关,可能还与菌株自身特性有关系,菌株菌丝生长速度快慢是否影响栽培出菇的产量与品质,有待更进一步探究。
吴小平等曾通过PDA平板筛选,发现香菇(Lentinula edodes)、鲍鱼菇(Pleurotus cystidiosus)和杏鲍菇(Pleurotus cryngi)等食用菌菌丝对木霉都没有抗性,但发现秀珍菇部分菌株对木霉有抗性[25]。本试验结果表明秀珍菇(XD-13)菌丝对木霉(T001)无拮抗作用。由此推断,在生产中遇上高温高湿天气,菇房、菌包中潜在的木霉病菌大量快速繁殖,当菌包温度介于32~34 ℃,秀珍菇菌丝与木霉菌丝之间的生长势相差更大,秀珍菇菌丝很快就失去竞争力,于是出现“绿包”现象,造成经济损失。要想避免秀珍菇生产过程中“绿包”现象的发生,首先菌包灭菌要彻底,保持菇房环境整洁卫生,其次可筛选出对木霉有抗性的秀珍菇新品种。
半合成培养基培养试验表明,秀珍菇同一菌株在3种添加淀粉或酵母粉的不同培养基上生长速度与对照PDA培养基无显著性差异。但添加酵母粉及酵母粉与淀粉组合的培养基,菌丝的生长势明显增强,菌丝更浓密,这与冯志勇等的研究结果一致[22],且添加淀粉培养基的菌丝普遍比正常菌丝白,呈现乳白色。有关营养物质的添加浓度,经添加营养物质培养的秀珍菇菌株活力是否更强,保藏时间是否更长,在栽培出菇阶段产量和品质表现等,有待深入研究。
-
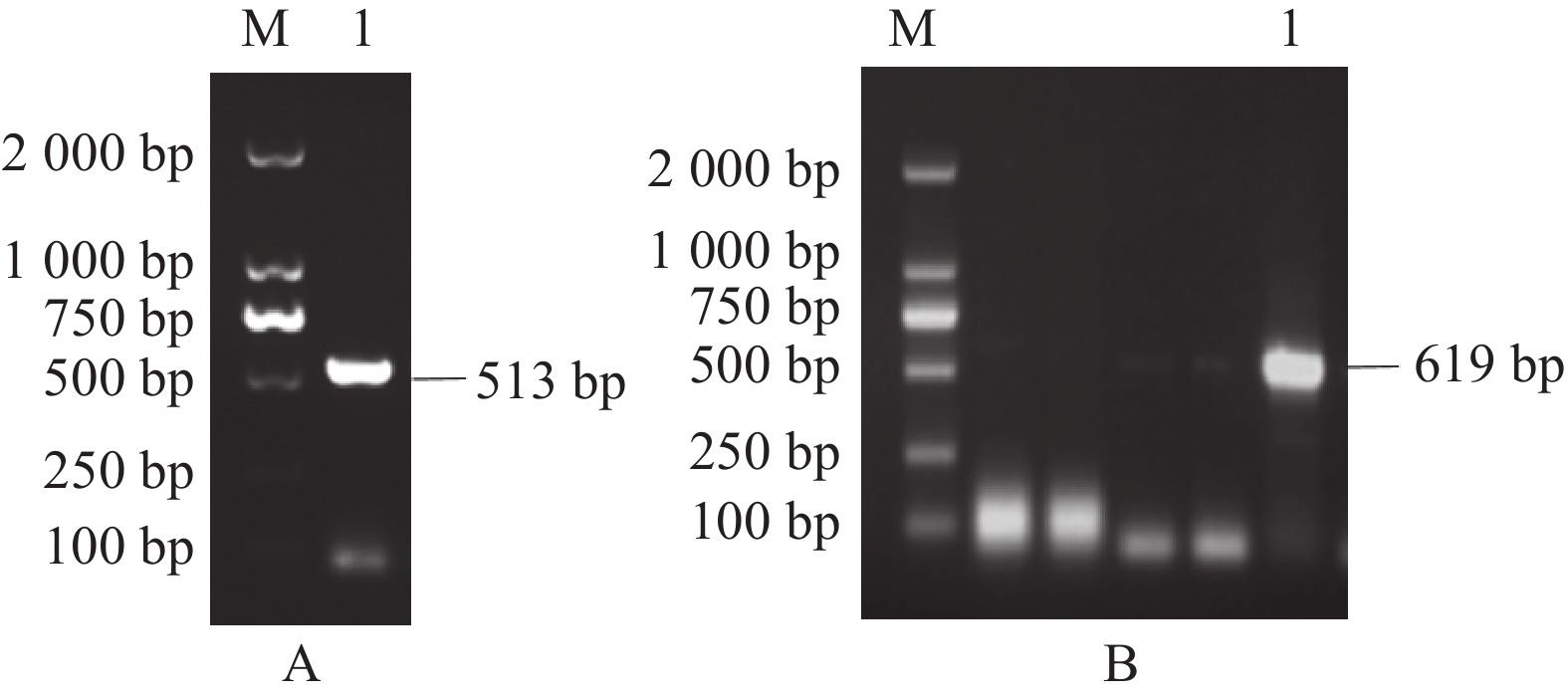
图 2 TGEV S基因A、D位点扩增结果
A:A位点扩增结果;M为DNA分子质量标准DL2000,1为扩增的A位点。B:D位点扩增结果;M为DNA分子质量标准DL2000,1为扩增的D位点。
Figure 2. Amplified A and D sites of TGEV S gene
A: amplification results of A site; M was DNA Marker DL2000, 1 was the PCR-amplified A site. B: amplification results of D site; M was DNA Marker DL2000, 1 was the PCR-amplified D site.
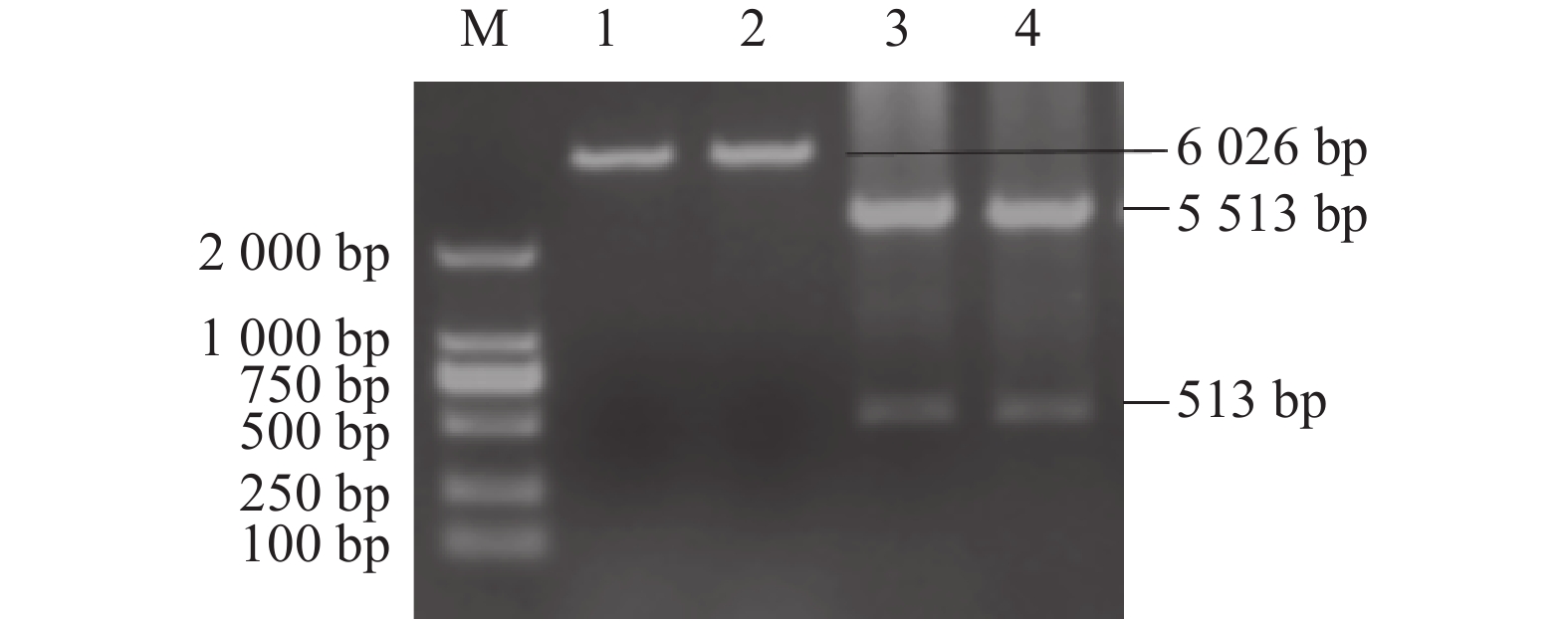
图 4 S基因A位点重组载体p-S(A)-His的构建与鉴定
M:DNA分子质量标准DL2000;1、2:重组载体p-S(A)-His的单酶切;3、4:重组载体p-S(A)-His的双酶切。
Figure 4. Construction and identification of p-S (A)-His and p-S (D)-His at A and D sites of S gene
M: DNA marker DL2000; M2: DNA marker DL10000; 1: single enzyme digestion of recombinant vector p-S (A)-His; 2: single enzyme digestion of recombinant vector p-S (D)-His; 3 and 4: double digestion of recombinant vector p-S (A)-His.
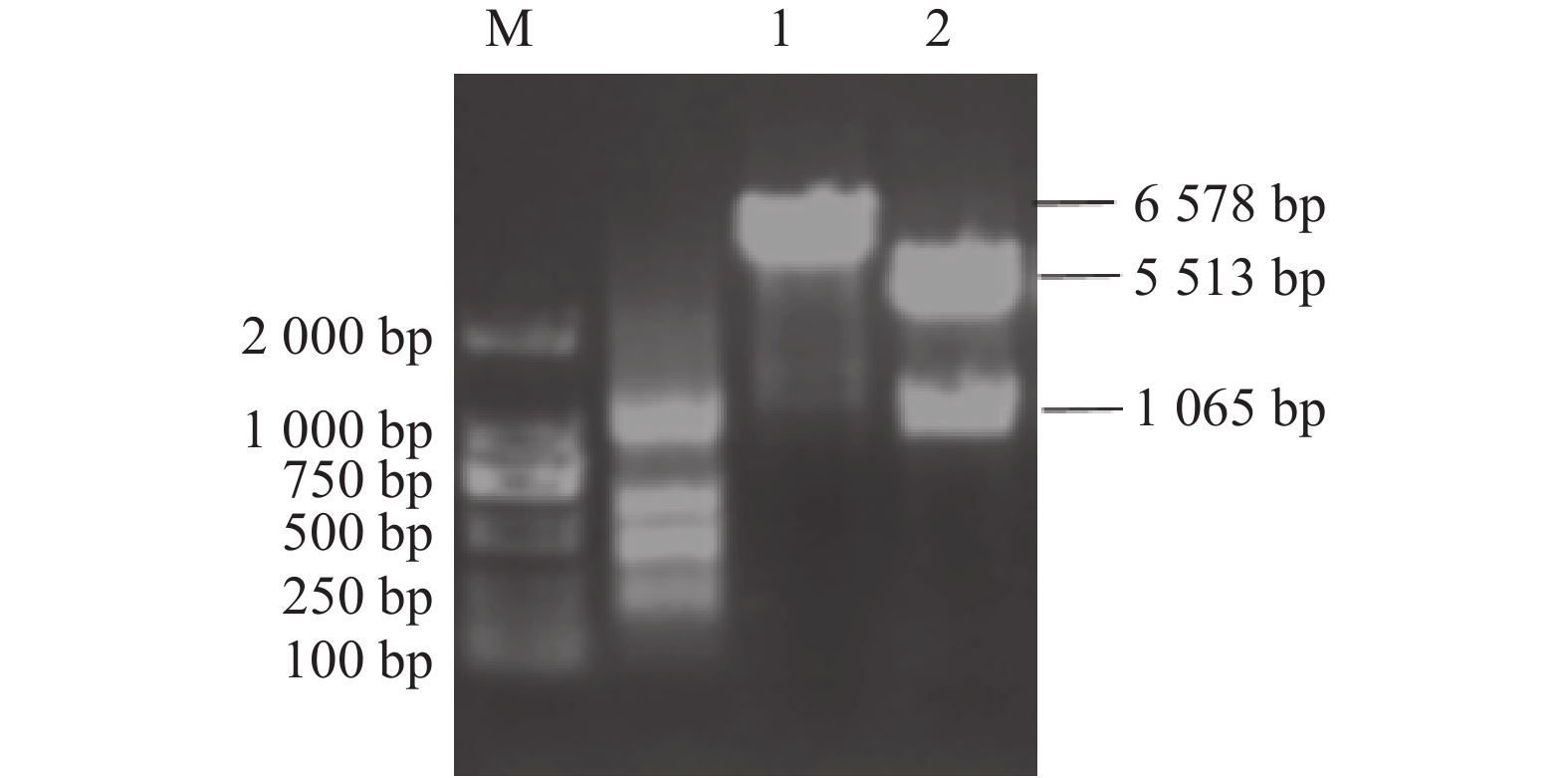
图 5 重组载体p-S(A-D)-His的构建与鉴定
M:DNA分子质量标准DL2000; 1:重组载体p-S(A-D)-His的单酶切;2:重组载体p-S(A-D)-His的双酶切。
Figure 5. Construction and identification of recombinant vectors p-S (A-D)-His
M: DNA marker DL2000; 1: single enzyme digestion of recombinant vector p-S(A-D)-His; 2: double digestion of recombinant vector p-S (A-D)-His.
表 1 S基因A位点、D位点和N基因扩增引物序列
Table 1 Sequences of primers for amplifications of A and D sites in S gene and N gene
基因
Gene引物
Primers序列(5′−3′)
Sequence(5′−3′)酶切位点
Restriction enzyme cutting siteS基因A位点
A-site in S geneP1 CGCGGATCCATGTTAGTTACCAAACAGCCGT Bam H I P2 CCGGAATTCTATTGTCCAGAAAACGTCAC Eco R I S基因D位点
D-site in S geneP3 CCGGAATTCAAGTTGAAAACACAGCTATT Eco R I P4 TGCTCTAGA ACTATTATCAGACGGTACACC Xba I N基因
N GeneP5 CGCGGATCCATGGCCAACCAGGGAC Bam H I P6 CCGGAATTCGTTCGTTACCTCATCAATT Eco R I 表中加下划线的碱基序列为酶切位点序列。
Cleavage sites are underlined.表 2 小鼠分组及免疫程序
Table 2 Groups and procedures of mice immunization
组别
Group疫苗载体种类
Vaccine carrier type免疫剂量
Immunizing dose/μg小鼠数量
Number of mice免疫时间
Immune frequency免疫位置
Immune site1 p-S(A-D)-His 240 6 第1、7、21天 脚底板 2 p-N-His 240 6 第1、7、21天 脚底板 3 p-S(A-D)-His + p-N-His 120+120 6 第1、7、21天 脚底板 4 pCDNA3.1-His-C 240 6 第1、7、21天 脚底板 表 3 S(A-D)蛋白和N蛋白亚细胞定位预测
Table 3 Predicted subcellular localization of S (A-D) and N proteins
组别
Group亚细胞定位
Subcellular localization可能性
Possibility/%S(A-D)蛋白
S(A-D) protein细胞质 Cytoplasm 34.8 线粒体 Mitochondria 17.4 细胞核 Cell nucleus 13.0 质膜 Plasmalemma 13.0 内质网 Endoplasmic reticulum 8.7 N蛋白
N protein细胞核 Cell nucleus 65.2 线粒体 Mitochondria 17.4 细胞质 Cytoplasm 13.0 溶酶体 Lysosome 4.3 表 4 S(A-D)蛋白和N蛋白的B细胞抗原表位预测
Table 4 Predicted B cell epitope of S (A-D) and N proteins
蛋白
Protein序号
Number起始位点
Start site结束位点
End site序列
Amino acid sequenceS(A-D)蛋白
S(A-D)protein1 33 43 FDQCNGAVLNN 2 55 62 TTNVQSGK 3 86 101 DSSFFSYGEIPFGVTD 4 195 211 NLNNGFYPVSSSEVGLV 5 233 250 LGMKRSGYGQPIASTLSN 6 281 295 ALWDNIFKRNCTDVL 7 306 318 CPFSFDKLNNYLT N蛋白
N protein1 4 32 QGQRVSWGDESTKTRGRSNSRGRKSNNIP 2 43 87 QGSKFWNLCPRDFVPNGIGNRDQQIGYWNRQTRYRMVKGQRKELP 3 101 108 ADAKFKDK 4 118 146 DGAMNKPTTLGSRGANNESKALKFDGKVP 5 150 189 QLEVNQSRDNSRSRSQSRSRSRNRSQSRGRQQSNNKKDDS 6 201 244 LGVDTEKQQQRSRSKSKERSNSKTRDTTPKNENKHTWKRTAGKG 7 251 271 GARSSSANFGDSDLVANGSSA 8 316 378 DPKTEQFLQQINAYARPSEVAKEQRKRKSRSKSAERSEQEVVPDALIENYTDVFDDTQVEIID 表 5 免疫小鼠抗TGEV血清IgG间接ELISA检测
Table 5 Anti-TGEV serum IgG in immunized mice detected by indirect ELISA
组别
Group疫苗载体种类
Vaccine carrier type免疫后不同时间抗TGEV血清IgG水平(OD450 nm)
Anti-TGEV serum IgG levels at different time after immunization (OD450 nm)0天
0 days14天
14 days28天
28 days42天
42 days1 p-S(A-D)-His 0.133±0.011 a 0.218± 0.0075 b0.243± 0.0060 c0.401± 0.0100 c2 p-N-His 0.138±0.016 a 0.244± 0.0025 a0.344± 0.0070 a0.504± 0.0141 a3 p-S(A-D)-His + p-N-His 0.142± 0.0050 a0.225± 0.0021 b0.300± 0.0050 b0.471± 0.0075 b4 pCDNA3.1-His-C 0.134± 0.0089 a0.144± 0.0076 c0.141± 0.0069 c0.139± 0.0020 d同列数据小写字母完全不同表示差异显著(P<0.05),含相同小写字母表示差异不显著(P>0.05)。
Data with different lowercase letters on same column indicate significant differences at P<0.05; those with same lowercase letters indicate no significant differences at P>0.05. -
[1] 张羽欣, 王树茂, 段宏勇, 等. 猪传染性胃肠炎病毒TaqMan实时荧光定量PCR检测方法的建立与应用 [J]. 中国兽医科学, 2024, 54(4):479−484. ZHANG Y X, WANG S M, DUAN H Y, et al. Establishment and application of TaqMan real-time quantitative PCR for detection of porcine transmissible gastroenteritis virus [J]. Chinese Veterinary Science, 2024, 54(4): 479−484. (in Chinese)
[2] JI Z Y, DONG H, JIAO R X, et al. The TGEV membrane protein interacts with HSC70 to direct virus internalization through clathrin-mediated endocytosis [J]. Journal of Virology, 2023, 97(4): e0012823. DOI: 10.1128/jvi.00128-23
[3] PU J N, CHEN D W, TIAN G, et al. All-trans retinoic acid attenuates transmissible gastroenteritis virus-induced inflammation in IPEC-J2 cells via suppressing the RLRs/NF-κB signaling pathway [J]. Frontiers in Immunology, 2022, 13: 734171. DOI: 10.3389/fimmu.2022.734171
[4] NIU Z, XU S S, ZHANG Y L, et al. Transmissible gastroenteritis virus nucleocapsid protein interacts with Na+/H+ exchanger 3 to reduce Na+/H+ exchanger activity and promote piglet diarrhea [J]. Journal of Virology, 2022, 96(22): e0147322. DOI: 10.1128/jvi.01473-22
[5] QIAN J T, LI M J, FENG Y F, et al. Genetic epidemiology of porcine transmissible gastroenteritis virus based on whole genome and S gene sequences[C]//2021 IEEE 9th International Conference on Bioinformatics and Computational Biology (ICBCB). May 25-27, 2021, Taiyuan, China. IEEE, 2021: 148-151.
[6] 郝振业. 猪传染性胃肠炎病毒反向遗传操作系统的构建及附属蛋白3(ORF3)的定位及功能研究[D]. 哈尔滨: 东北农业大学, 2023. HAO Z Y. Construction of reverse genetic operating system of porcine transmissible gastroenteritis virus and study on location and function of accessory protein 3(ORF3)[D]. Harbin: Northeast Agricultural University, 2023. (in Chinese)
[7] 王艳春. 猪传染性胃肠炎病毒S基因A位点杆状病毒表达及初步应用[D]. 扬州: 扬州大学, 2020. WANG Y C. Expression of baculovirus at site A of S gene of porcine infectious gastroenteritis virus and its preliminary application[D]. Yangzhou: Yangzhou University, 2020. (in Chinese)
[8] 张海燕. PEDV和TGEV的S蛋白融合抗原表位核酸疫苗的研究[D]. 武汉: 华中农业大学, 2019. ZHANG H Y. Study on S protein fusion epitope nucleic acid vaccine of PEDV and TGEV[D]. Wuhan: Huazhong Agricultural University, 2019. (in Chinese)
[9] LI X L, LI P C, CAO L Y, et al. Porcine IL-12 plasmid as an adjuvant improves the cellular and humoral immune responses of DNA vaccine targeting transmissible gastroenteritis virus spike gene in a mouse model [J]. Journal of Veterinary Medical Science, 2019, 81(10): 1438−1444. DOI: 10.1292/jvms.18-0682
[10] 师一鸣. TGEV单克隆抗体的制备及检测方法的建立[D]. 哈尔滨: 东北农业大学, 2019. SHI Y M. Preparation of TGEV monoclonal antibody and establishment of detection method[D]. Harbin: Northeast Agricultural University, 2019. (in Chinese)
[11] 李雅静, 宫强. 铜绿假单胞菌oprH基因DNA疫苗的构建与检测 [J]. 现代畜牧兽医, 2022(11):19−23. DOI: 10.3969/j.issn.1672-9692.2022.11.lnxmsy202211006 LI Y J, GONG Q. Construction and detection of Pseudomonas aeruginosa oprH gene DNA vaccine [J]. Modern Journal of Animal Husbandry and Veterinary Medicine, 2022(11): 19−23. (in Chinese) DOI: 10.3969/j.issn.1672-9692.2022.11.lnxmsy202211006
[12] 杨鹏, 吴燕, 岳筠, 等. 绵羊肺炎支原体P113蛋白C末端基因真核表达载体的构建及其小鼠免疫应答 [J]. 中国兽医学报, 2022, 42(3):496−501, 521. YANG P, WU Y, YUE J, et al. Construction of eukaryotic expression vector for C terminal gene of Mycoplasma ovipneumoniae P113 protein and its immune response in mice [J]. Chinese Journal of Veterinary Science, 2022, 42(3): 496−501, 521. (in Chinese)
[13] 姚思, 杨洁琼, 杨雨欣, 等. 结核分枝杆菌ESAT6-Fc DNA疫苗的免疫效应评价 [J]. 中国生物制品学杂志, 2023, 36(8):897−901. YAO S, YANG J Q, YANG Y X, et al. Evaluation of immune effect of ESAT6-Fc DNA vaccine against Mycobacterium tuberculosis [J]. Chinese Journal of Biologicals, 2023, 36(8): 897−901. (in Chinese)
[14] 黄小波, 杨恒, 曹三杰, 等. 猪传染性胃肠炎病毒S-N融合双基因疫苗的构建及其免疫原性分析 [J]. 中国兽医科学, 2012, 42(8):848−853. HUANG X B, YANG H, CAO S J, et al. Construction and immunogenicity analysis of the S-N fusion gene vaccine against porcine transmissible gastroenteritis virus [J]. Chinese Veterinary Science, 2012, 42(8): 848−853. (in Chinese)
[15] WANG G, LIANG R, LIU Z W, et al. The N-terminal domain of spike protein is not the enteric tropism determinant for transmissible gastroenteritis virus in piglets [J]. Viruses, 2019, 11(4): 313. DOI: 10.3390/v11040313
[16] 韩郁茹, 石达, 张记宇, 等. 猪急性腹泻综合征冠状病毒RT-LAMP快速检测方法的建立与应用 [J]. 中国预防兽医学报, 2021, 43(1):35−39. HAN Y R, SHI D, ZHANG J Y, et al. Development and application of RT-LAMP method for rapid detection of SADS-CoV [J]. Chinese Journal of Preventive Veterinary Medicine, 2021, 43(1): 35−39. (in Chinese)
[17] 樊杰. 猪传染性胃肠炎病毒N蛋白纳米抗体的制备和基于纳米抗体竞争ELISA的建立[D]. 杨凌: 西北农林科技大学, 2021. FAN J. Preparation of nano-antibody against N protein of transmissible gastroenteritis virus and establishment of competitive ELISA based on nano-antibody[D]. Yangling: Northwest A & F University, 2021. (in Chinese)
[18] ZHANG Y D, ZHANG X H, LIAO X D, et al. Construction of a bivalent DNA vaccine co-expressing S genes of transmissible gastroenteritis virus and porcine epidemic diarrhea virus delivered by attenuated Salmonella typhimurium [J]. Virus Genes, 2016, 52(3): 354−364. DOI: 10.1007/s11262-016-1316-z
[19] 何雷, 董玲娟, 张彦明, 等. 猪传染性胃肠炎病毒ORF7蛋白在ST细胞中定位及其对病毒复制影响的研究 [J]. 中国预防兽医学报, 2020, 42(6):543−548. HE L, DONG L J, ZHANG Y M, et al. The subcellular location of transmissible gastroenteritis virus protein ORF7 and its effect on viral replication [J]. Chinese Journal of Preventive Veterinary Medicine, 2020, 42(6): 543−548. (in Chinese)
[20] 何雷, 贾艳艳, 郁川, 等. 稳定表达猪传染性胃肠炎病毒N蛋白的ST细胞株的构建及其亚细胞定位 [J]. 中国预防兽医学报, 2016, 38(2):101−104. DOI: 10.3969/j.issn.1008-0589.2016.02.04 HE L, JIA Y Y, YU C, et al. Establishment of stably-expressed transmissible gastroenteritis virus N protein cell line and its subcellular location [J]. Chinese Journal of Preventive Veterinary Medicine, 2016, 38(2): 101−104. (in Chinese) DOI: 10.3969/j.issn.1008-0589.2016.02.04
[21] 韩涛涛, 黎露, 唐青海, 等. 不同佐剂对猪传染性胃肠炎病毒S蛋白和猪流行性腹泻病毒S蛋白免疫原性的影响 [J]. 中国农学通报, 2020, 36(30):143−150. DOI: 10.11924/j.issn.1000-6850.casb20191000740 HAN T T, LI L, TANG Q H, et al. Different adjuvants: Effects on S protein immunogenicity of porcine transmissible gastroenteritis virus and porcine epidemic diarrhea virus [J]. Chinese Agricultural Science Bulletin, 2020, 36(30): 143−150. (in Chinese) DOI: 10.11924/j.issn.1000-6850.casb20191000740
[22] 伊立超. PEDV和TGEV受体结合区基因在昆虫杆状病毒系统的表达与免疫原性分析[D]. 延吉: 延边大学, 2022. YI L C. Expression and immunogenicity analysis of PEDV and TGEV receptor binding region genes in insect baculovirus system[D]. Yanji: Yanbian University, 2022. (in Chinese)
-
期刊类型引用(3)
1. 卯明娟,刘迪,周会明,白玉英,洪鹏,王佳琳,杨流波,陈真敏. 基于高产胞外粗多糖的白灵芝发酵茶培养基配方优化. 食品工业科技. 2024(04): 93-100 .  百度学术
百度学术
2. 闫静,王伟科,陆娜,宋吉玲,周祖法. 利用孢子紫外诱变选育秀珍菇新菌株. 中国食用菌. 2024(02): 38-45 .  百度学术
百度学术
3. 胡佳,陈鑫. 高温天气因素对杏鲍菇不同生长阶段的影响. 中国农学通报. 2024(36): 57-62 .  百度学术
百度学术
其他类型引用(0)




 下载:
下载: