Identification and Sequences of Key Polyphenol-synthesis Genes in Mango Pulp
-
摘要:目的
鉴定探究杧果(Mangifera indica L.)苯丙氨酸解氨酶(phenylalanineammonialyase, PAL)与杧果多酚氧化酶(polyphenol oxidase, PPO)基因(MiPAL、MiPPO)家族成员及其在杧果果肉生长发育中的表达调控模式。
方法基于杧果基因组,利用生物信息学方法,从蛋白质特性、系统进化关系、基因结构、启动子顺式作用元件等多个方面鉴定MiPAL与MiPPO基因家族成员,并通过转录组分析鉴定MiPAL、MiPPO基因家族成员的表达模式。
结果杧果中共鉴定出10个MiPAL基因家族成员,1个MiPPO基因家族成员;MiPAL、MiPPO蛋白均属于稳定蛋白与亲水性蛋白,多定位于叶绿体。多物种PAL、PPO基因系统发育分析表明,PAL、PPO基因均被分为4个亚组,木本植物基因家族成员多分布在同一亚组。序列分析显示,25%MiPAL基因缺少内含子,MiPPO基因无内含子序列。10个MiPAL基因分布于5条染色体与1条碎片片段上,11对片段复制基因和7个串联重复基因;MiPPO基因定位于染色体Chr12,与毛果杨(Populus trichocarpa Torr. & Gray)PPO13为同源基因。顺式作用元件分析确定了MiPAL、MiPPO成员启动子中包含生长发育元件、激素响应元件和逆境响应相关元件,表明其呈诱导型表达。MiPAL基因分别在幼果期与成熟期高表达;MiPPO基因在成熟期高表达。
结论杧果基因组中共鉴定得到10个MiPAL基因家族成员与1个MiPPO基因家族成员,同家族成员基因结构较为保守;MiPAL与MiPPO表达调控因素较多,其中MiPAL7的表达模式与杧果果肉多酚含量变化趋势相符,表明其参与了杧果果肉中多酚积累。
Abstract:ObjectiveGenes of phenylalanine ammonia lyase (MiPAL) and polyphenol oxidase (MiPPO) syntheses inMangifera indica L. were identified with their regulation functions on fruit growth and development determined.
MethodsUsing bioinformatics methods and mango genomes, the PAL and PPO family members in the fruits were identified based on analyses of protein characteristics, phylogenetic relationship, gene structure, and promoter cis acting elements. Expressions of the identified genes during fruit development were determined by transcriptome analysis.
ResultsTen PALs and onePPO in the mangos were identified. They were stable, hydrophilic proteins located mainly in the chloroplasts. Being a woody plant, these genes are phylogenetically in a same subgroup of 4 PAL and PPO categories. The sequences of 25% of MiPALs lacked introns, and that of MiPPO without intron at all. The 10 MiPALs were distributed in 5 chromosomes and one fragment with 11 pairs of segmental duplication genes and 7 tandem duplication genes. Located in chromosome Chr12, MiPPO was homologous to PPO13 in Populus tomentosa. The cis acting elements in the promoters of MiPAL and MiPPO contained those of growth, development, and hormone and stress responses expressed in an inducible pattern. MiPALs were highly expressed in the fruits at both young and mature stages, whereas MiPPO only at maturation.
ConclusionWith the information obtained, further exploration on the molecular regulatory mechanisms and biological functions of polyphenol accumulation in mango pulp was in order.
-
Keywords:
- Mangifera indica L. /
- polyphenol synthesis /
- gene family /
- gene expression
-
0. 引言
【研究意义】苯丙氨酸解氨酶(phenylalanineammonialyase, PAL)与多酚氧化酶(polyphenol oxidase, PPO)作为苯丙烷合成的限速酶与多酚氧化末端酶,参与了多酚的合成与降解。杧果(Mangifera indica L.)作为世界上热带生态地区最重要的水果之一,是仅次于香蕉(Musa nana Lour.)、葡萄(Vitis vinifera L.)、苹果(Malus pumila Mill.)和橙子(Citrus reticulata Blanco)的全球第五大水果作物[1],在果业生产上具有重要地位,而探究杧果苯丙氨酸解氨酶基因(MiPAL)与杧果多酚氧化酶基因(MiPPO)家族成员及其在杧果果肉生长发育中的表达调控模式,对深入研究杧果果肉多酚积累的分子调控机理和生物学功能具有重要意义。【前人研究进展】多酚类化合物是植物中含有一个或多个羟基苯环结构的一类重要次生代谢物质[2],其参与植物的生长发育过程,类黄酮类物质在决定果实颜色[3−5]、花瓣颜色[6]的同时,也参与了植物响应逆境胁迫的过程,如重金属胁迫[7]、UV-B辐射胁迫[8]、盐胁迫[9]、干旱胁迫[10]等多种胁迫,当植株在经受非生物胁迫与生物胁迫时,会提高细胞中单宁的含量,从而提高抗氧化与抗虫能力 [11−13]。苯丙烷代谢途径是植物多酚合成最为重要的途径[14−15],PAL 作为控制植物初生代谢到次生代谢碳通量的关键酶,同时也是苯丙烷代谢途径的起始酶与限速酶 [16−17]。研究发现逆境胁迫诱导了苯丙烷代谢,以增强植物对胁迫的抗性,例如UV-B辐射胁迫下,胡萝卜PAL基因的表达量明显上升,同时多酚类物质积累增加,以抵抗氧化应激损伤[18];对丹参(Salvia miltiorrhiza Bunge)的干旱胁迫研究中,也发现了这一现象 [19];小麦(Triticum aestivum L.)与红树(Rhizophora apiculata Bl.)在重金属胁迫下,都表现出酚类化合物含量明显上升,且PAL酶活性与基因表达量明显上升[20−21];PPO是一种含铜的氧化还原酶,可将酚氧化为醌,根据底物类型与作用机制,可以将PPO分为:酪氨酸酶(EC 1.14.18.1)、儿茶酚氧化酶(EC 1.10.3.1)和漆酶(EC 1.10.3.2)[22]。多数的研究中,PPO参与了酶促褐变反应 ,但PPO也参与了酚类物质的合成,甜菜素的生物合成中,酪氨酸酶是负责氧化催化的酶之一[23];金鱼草的花瓣色素,是由金鱼草素合酶(多酚氧化酶),特异性催化查耳酮氧化形成橙酮积累的,这使花瓣呈现黄色[24]。目前对杧果苯丙氨酸解氨酶(PAL)与多酚氧化酶(PPO)的研究主要集中于果实采后贮藏品质变化、抗病响应与果皮颜色转化,PAL与PPO基因在杧果中的功能研究也主要集中于采后的果皮转色与抗病相关。【本研究切入点】而关于杧果果肉多酚积累的分子机理及其相关基因家族研究则鲜见报道。【拟解决的关键问题】本研究以杧果基因组与试验组获得的杧果不同生长周期的杧果果肉转录组数据为基础,对杧果PAL与PPO基因家族进行生物信息学分析,并探究不同生长周期的表达情况,以期为杧果抗逆生理机制与抗逆品种选育等研究奠定基础。
1. 材料与方法
1.1 试验材料
杧果基因族及注释数据下载于NCBI基因组数据库(https://www. ncbi.nlm.nih.gov/datasets/taxonomy/29 780/),毛果杨(Populus trichocarpa Torr. & Gray)基因组与注释下载于JGI数据库(https://data.jgi.doe.gov/refine-download/phytozome?organism=PtrichocarpaStettler14&expanded=532&_gl=1*1w085ne*_ga*MTI5Mzg3NDQwNS4xNzE0NzE5MDg5*_ga_YBLMHYR3C2*MTcxNTMzNzgwOC4xMS4wLjE3MTUzMzc4MDguMC4wLjA.),拟南芥[Arabidopsis thaliana (L.) Heynh.]PAL(AtPAL)基因家族序列下载于tair数据库(https://www.arabidopsis.org/),马铃薯(Solanum tuberosum L.)、毛果杨(Populus trichocarpa Torr. & Gray)、玉米(Zea mays L.)、水稻(Oryza sativa L.)的PAL(StPAL、PtrPAL、ZmPAL、OsPAL)与PPO(StPPO、PtrPPO、ZmPPO、OsPPO)基因序列下载于phytozome数据库(https://phytozome-next.jgi.doe.gov/)。
杧果果肉取样地为海南省三亚市海棠区升昌村的三亚游龙农业发展有限公司基地(18°24′47.99″N、109°46′43.65″E)。该地属热带海洋性季风气候,高温多湿,长夏无冬,5~10月为雨季,11月至翌年4月为旱季,年晴日300 d以上且太阳紫外辐射较强,日均辐射剂量约83.47 kJ·m−2·d−1。园区山地,砖红壤砂壤土,选择16年生且生长健壮、树势较均匀、无病虫害的台农一号杧果植株5株进行试验。
采样开始于花后30 d(2023年11月6日),之后每隔10 d进行实验设备的维护与样品采集,2023—2024年进行7次取样,取样时在每棵树的中部外围四周选择5个大小一致且适中的果实作为取样果,当场去皮,取完样后及时放入液氮中速冻带回实验室于超低温冰箱(−80 ℃)中保存备用。
1.2 试验方法
1.2.1 杧果果肉多酚含量测定
杧果果肉总酚含量利用植物总酚(total phenols, TP)试剂盒(苏州科铭生物技术有限公司)测定,过程按照试剂盒说明书进行。
1.2.2 杧果MiPAL、MiPPO基因家族成员鉴定
以AtPAL与PtrPPO基因家族成员蛋白序列作为参考,利用TBtools软件,运行本地Blast,与杧果蛋白序列进行比对,得到1条杧果多酚氧化酶基因(MiPPO)家族候选序列与10条杧果苯丙氨酸解氨酶基因(MiPAL)家族候选序列。
登录NCBI,利用在线工具NCBI blastp (https://blast.ncbi.nlm.nih.gov/Blast.cgi),以Swiss-Prot数据库(http://www.gpmaw.com/html/swissprot.html)作为参考,对MiPPO、MiPAL候选序列进行比对,去除多余基因。
利用InterPro (https://www.ebi.ac.uk/interpro/)与已知PPO保守结构域(PF00246、PF12143、PF12143)、PAL保守结构域(PF00221、PLN02457),结合在线分析工具NCBI batch wab CD-search tool(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi),剔除保守结构域不相符的基因,从而获得确定的MiPAL、MiPPO基因家族成员,完成后对已筛选出的基因进行重命名。
1.2.3 蛋白质特性分析
利用在线工具ProtParam(http://web.expasy.org/protparam/)预测MiPPO、MiPAL蛋白特性,包括氨基酸数、分子量(molecular weight, MW)、等电点(isoelectric point, pI)等,利用WoLF PSORT(https://wolfpsort.hgc.jp/)进行亚细胞定位预测。
1.2.4 杧果MiPAL、MiPPO家族基因结构、保守基序和保守结构域分析
将筛选后MiPAL、MiPPO蛋白序列提交到NCBI batch wab CD-search tool(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)进行保守结构域分析,再将蛋白文件提交到MEME(https://meme-suite.org/meme/tools/meme),设置分析motif数值为8,分析MiPAL、MiPPO蛋白的保守基序,最后利用基因组注释文件分析MiPAL、MiPPO基因结构;利用tbtools软件进行可视化处理。
1.2.5 MiPAL蛋白系统进化的构建
使用MEGA 11,以neighbor-joining(NJ)方法创建杧果、马铃薯、毛果杨、玉米、水稻的PAL与PPO蛋白序列的系统进化树,并将Bootstrap值设为
1000 ,另外构建10个MiPAL基因家族成员的系统进化树。1.2.6 MiPAL、MiPPO基因家族启动子顺式作用元件分析及功能预测
根据杧果基因族序列信息,利用TBtools 提取MiPPO、MiPAL基因家族序列上游长度为2 000 bp的启动子序列,并将其上传到PlantCARE(http:// bioinformatics.psb.ugent.be/webtools/ plantcare/html/)进行预测分析,通过TBtools进行可视化。
1.2.7 染色体分布和物种内共线性、选择压分析
使用TBtools进行杧果物种内共线性分析、杧果与毛果杨的中间共线性分析。利用杧果全基因族注释信息获取所有基因密度信息和MiPAL、MiPPO成员在染色上的位置,使用基因组全序列文件,计算染色体GC值与gap位置,使用自然梯度(natural gradient descent, NG)法计算同源基因对的Ka和Ks值,进行选择压分析。
1.2.8 MiPAL、MiPPO家族成员的表达模式分析
根据实验组委托武汉迈维代谢生物科技股份有限公司所测的杧果转录组数据,该转录组提取杧果花后30、50、91 d果肉的RNA进行转录组测序,每个时期各3个生物学重复。RNA经纯化并检验合格后建立cDNA文库,库检合格后,不同文库按照目标下机数据量进行pooling,用Illumina平台进行测序。将测序后的原始数据raw data过滤后得到clean reads,与参考基因组对比后,进行基因表达定量分析,从中提取MiPAL、MiPPO基因的FPKM值,利用TB-tools绘制基因在不同组织中的表达热图。
选取MiPAL1、3、6、7,用primer3 (https://bioinfo.ut.ee/primer3-0.4.0)设计qRT-PCR特异性引物(表1),由铂尚生物技术(上海)有限公司合成引物。提取的果肉RNA用SteadyPure 植物 RNA 提取试剂盒(杭州艾科瑞生物科技有限公司),使用Evo M-MLV 反转录预混型试剂盒(杭州艾科瑞生物科技有限公司)完成逆转录,操作按照试剂盒说明书进行。用2× Q3 SYBR qPCR Master mix (Universal) (TOLOBIO)和德国耶拿的qTOWER3仪器进行qRT-PCR验证。使用2−ΔΔCt法计算基因的相对表达量。
表 1 杧果MiPAL及内参基因引物序列Table 1. Sequences of primers of MiPALs and reference genes基因名
Gene name上游引物序列 (5′-3′)
Primer sequence (5′-3′)下游引物序列 (5′-3′)
Primer sequence (5′-3′)MiPAL1 AATGGCCAACGGAGAGAGAG TGGGGACTTTACTTTCTCACCA MiPAL3 ACCAATCATGTCCAGAGCGC CCTCCAAGTGCCTCAAGTCA MiPAL6 GACCCCTGCAGTGCTACTTA ATCGCCGCATTTCCATTCTC MiPAL7 CCAATCCAGTCACCAGCCAT TGCCTCAGGTCAATCGCTTG Actin7 ATCTGCTGGAAGGTGCTGAG CCAAGCAGCATGAAGATCAA 2. 结果与分析
2.1 杧果果实生长过程果肉总酚含量变化情况
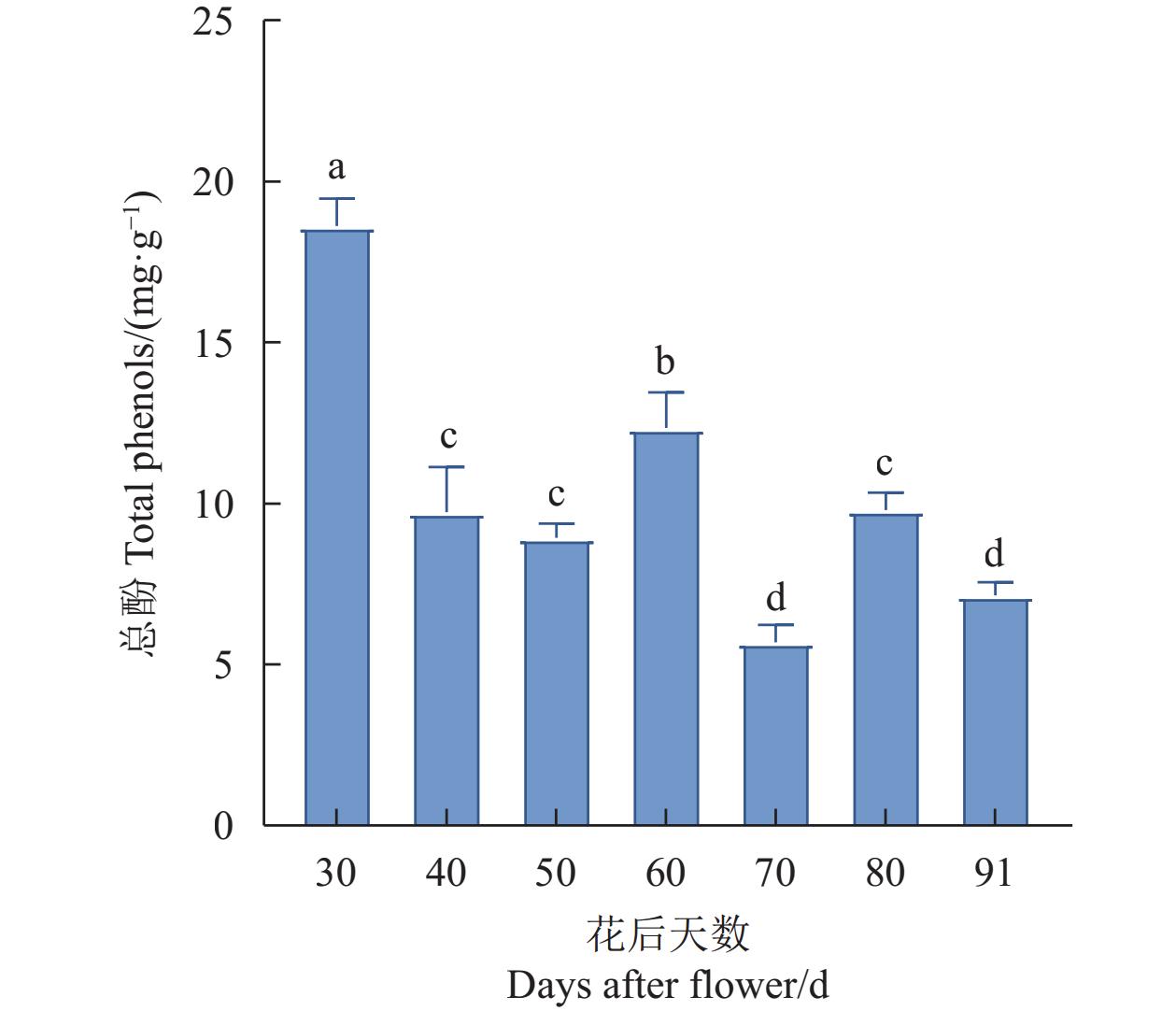
杧果果肉中总酚含量动态变化差异如图1所示。随果实的生长与发育,呈现“降—升—降—升”的趋势。在花后30 d时,果肉中总酚含量远远高于其他时期;随果实生长与发育,果肉总酚含量总体上呈下降趋势,在花后60 d和80 d两次出现升降波动,但花后80 d低于花后60 d。说明果肉总酚的积累趋势不稳定,受生长发育进程和环境变化影响。
2.2 MiPAL与MiPPO基因家族成员鉴定及其系统发育分析
利用AtPAL与PtrPPO氨基酸序列在杧果蛋白全序列中初步筛选出10个MiPAL与1个MiPPO候选基因,通过保守结构域分析,结合NCBI的Blastp,筛除不含PF00221、PLN02457的PAL候选序列与不含PF00246、PF12143、PF12143结构域的PPO候选序列,得到10个MiPAL基因家族成员与1个MiPPO基因家族成员,重命名为MiPAL1~10、MiPPO1(表2)。结果显示MiPAL基因家族成员蛋白序列所含氨基酸个数较为均匀,所含个数为700~719个,蛋白分子质量为
76578.56 ~78902.02 Da,理论等电点为5.76~6.57;不稳定系数30.41~36.46,所有成员不稳定系数小于40,属于稳定蛋白;脂肪系数为90.11~92.99;所有家族成员的平均亲水系数均小于 0,属于亲水性蛋白;MiPAL2、MiPAL5预测亚细胞定位于细胞质,MiPAL8定位于细胞核,MiPAL10定位于内质网,其余6个成员均定位于叶绿体,MiPAL蛋白理化性质与亚细胞定位差异并不明显。MiPPO1蛋白含氨基酸593个,蛋白分子质量为66775.87 Da,理论等电点6.95;不稳定系数37.57,属于稳定蛋白;脂肪系数为79.12;平均亲水系数均小于 0,属于亲水性蛋白;预测亚细胞定位于叶绿体。表 2 MiPAL、MiPPO蛋白基本信息Table 2. Basic information on MiPAL and MiPPO proteins基因
Gene基因编号
Gene ID氨基酸数量
Number of amino acids分子量
MW/Da等电点
pI不稳定系数
Instability index脂肪系数
Aliphatic index平均亲水系数
GRAVY亚细胞定位
Subcellular localizationMiPAL1 LOC123193566.1 707 77066.14 6.47 35.46 90.11 −0.151 叶绿体 MiPAL2 LOC123201898.1 704 76781.74 6.26 32.8 91.58 −0.184 细胞质 MiPAL3 LOC123206701.1 707 76933.96 6.46 34.55 90.11 −0.144 叶绿体 MiPAL4 LOC123206728.1 707 77051.13 6.57 35.45 90.11 −0.151 叶绿体 MiPAL5 LOC123209119.1 719 78902.02 6.16 33.16 90.21 −0.229 细胞质 MiPAL6 LOC123224091.1 712 77440.26 5.76 30.42 90.7 −0.179 叶绿体 MiPAL7 LOC123225153.1 708 76938.82 6.15 30.41 92.99 −0.162 叶绿体 MiPAL8 LOC123228411.1 700 76578.56 6.06 36.46 90.6 −0.16 细胞核 MiPAL9 LOC123192215.1 707 76933.96 6.46 34.55 90.11 −0.144 叶绿体 MiPAL10 LOC123224083.1 712 77333.39 6.1 27.11 92.49 −0.163 内质网 MiPPO1 LOC123193265.1 593 66775.87 6.95 37.57 76.12 −0.501 叶绿体 为分别研究MiPAL、MiPPO基因家族的进化关系,以MiPAL、StPAL、PtrPAL、ZmPAL、OsPAL与MiPPO、StPPO、PtrPPO、ZmPPO、OsPPO分别构建蛋白序列系统进化树;结果如图2、3所示。PAL进化树由4个亚组组成;PAL进化树中亚组B的基因数量最多(17个),亚组C(13个)与亚组D(15个)的基因数量相近,A亚组最少(2个)。PPO进化树由3个亚组组成,亚组A(15个)基因数量最多,其次为亚组B(13个),最少为亚组C(5个);值得注意的是,在PAL进化树中,单子叶植物中,ZmPAL、OsPAL都汇集在一个亚组中,双子叶植物中StPAL分布在亚组A、C,MiPAL、PtrPAL混合分布在亚组D。另外,在PPO进化树中,MiPPO1、ZmPPO4、PtrPPO10、PtrPPO12、PtrPPO13分布于亚组A中,PtrPPO11与ZmPPO、OsPPO分布于亚组C,其余PtrPPO分布于亚组B,StPPO独立分布在亚组D。总之,PAL基因在双子叶植物与单子叶植物间有较远的亲缘关系,在木本植物与草本植物之间也有明显的区分,说明PAL基因功能的形成,早于双子叶植物与单子叶植物间的分化,也可能早于木本植物与草本植物间的分化;PPO基因主要在木本植物与草本植物之间有较为远的亲缘关系,但部分基因没有遵循这一规律,MiPPO1所在的亚组中就存在水稻的PPO基因,说明PPO基因在物种分化后仍进行了进化。
2.3 MiPPO、MiPAL基因结构、保守结构域及保守基序分析
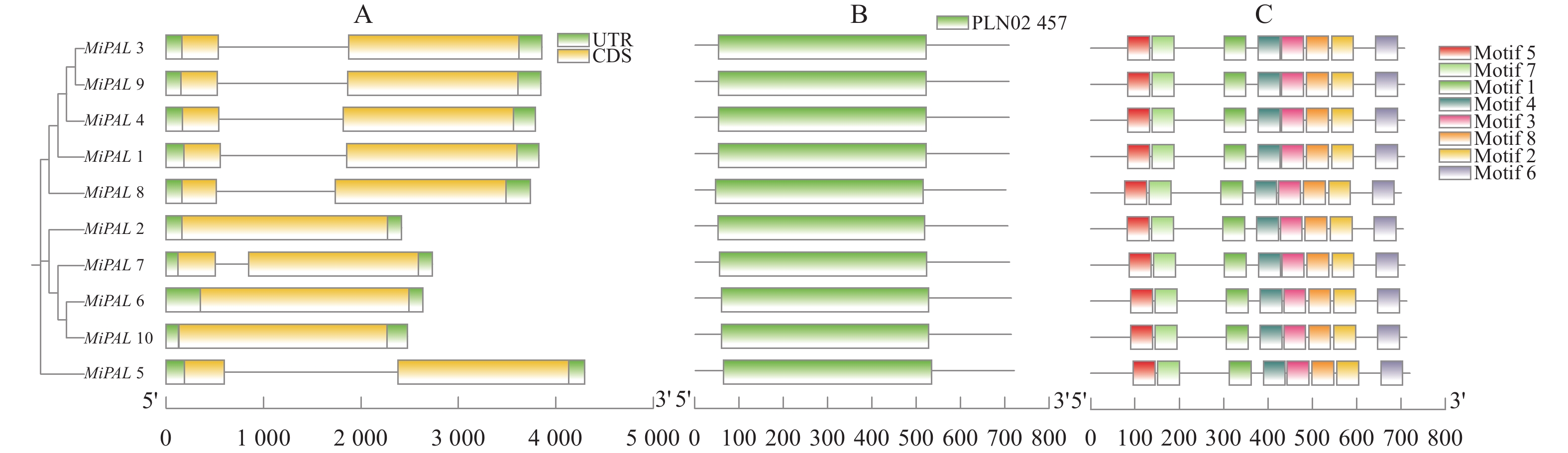
为了进一步探究MiPAL基因家族不同成员之间的进化关系,使用MiPAL蛋白构建了一个系统发育树,并结合保守基序、保守结构域和基因结构共同分析。由图4A所示,MiPAL基因家族成员拥有两种内含子-外显子模式,MiPAL2、6、10没有内含子,其他家族成员均有1个内含子与2个外显子。由图4B可知,所有的MiPAL基因家族成员都包含保守结构域PLN02457,保守结构域作为蛋白质三级结构的基本结构单位,作为蛋白功能的重要单元,由此表明结构域PLN02457决定了PAL蛋白的功能。保守基序分析结果如图4C所示。亲缘关系越近的成员其基序结构越相似,MiPAL基因家族成员都包含motif 1~8,且都以N→C端按序排列,motif 1~8可能与结构域PLN02457有关。
![]() 图 4 杧果PAL基因结构分析A:基因结构分析; B:保守结构域分析; C:保守基序分析;图5同。Figure 4. Structure of PAL in M. indicaA: Gene structure analysis; B: conservative structural domain analysis; C: conservative motif analysis. Same for Fig. 5.
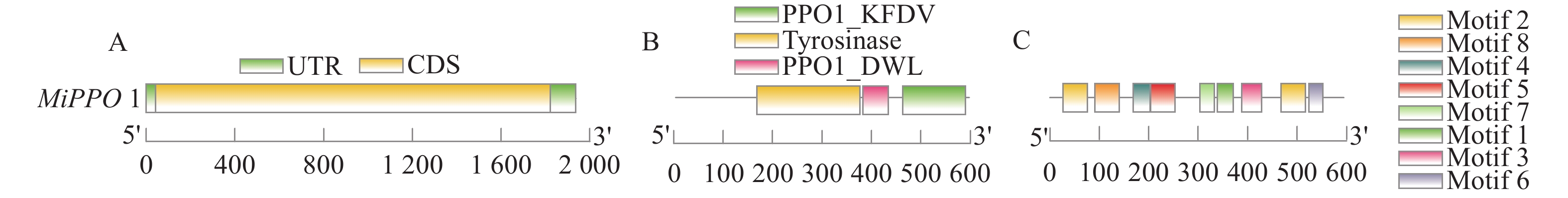
图 4 杧果PAL基因结构分析A:基因结构分析; B:保守结构域分析; C:保守基序分析;图5同。Figure 4. Structure of PAL in M. indicaA: Gene structure analysis; B: conservative structural domain analysis; C: conservative motif analysis. Same for Fig. 5.由图5可见,MiPPO1基因含有一个外显子,无内含子;MiPPO1蛋白序列中包含了3个保守结构域,包括PPO1-KFDV、tyrosinase和 PPO1_DWL;MiPPO中鉴定得到了8个保守基序,其中motif 4、5、7、1与C端tyrosinase结构域相关,motif 3与中间PPO_DWL结构域相关,motif 2、6与N端PPO-KFDV结构域相关。
2.4 MiPAL家族染色体分布、物种内共线性和选择压分析
MiPAL基因染色体定位分析结果表明,MiPAL分布在5条不同染色体与1条碎片片段上;MiPAL家族成员在染色体上的分布并不均匀,Chr1、NW_1138上各分布两个家族成员,而在Chr2、10、12、18的基因密度较高的区域中仅包含1个家族成员;共线性分析结果表明,7个家族成员(70%)参与了串联复制,共产生了4个串联复制基因簇,分布于Chr1、NM_1138上;6个基因(60%)间共发现了11对同源关系(图6),这些基因分布于5条染色体与1条碎片片段上,说明串联复制是MiPAL进化扩张的主要驱动力。其中2个基因(MiPAL3、MiPAL7)既参与了片段复制,也参与了串联复制;表3结果表明同源基因间的Ka/Ks值均远小于1,说明进化过程中主要受到纯化选择压力的影响。
表 3 杧果PAL复制基因对的Ka、Ks分析Table 3. Ka and Ks analysis on PAL replication pairs of M. indica同源基因
HomologsKa Ks Ka/Ks MiPAL7-MiPAL2 0.029459743 0.421135092 0.06995319 MiPAL5-MiPAL2 0.118273297 2.076835221 0.056948811 MiPAL8-MiPAL2 0.09078887 1.857097035 0.048887521 MiPAL1-MiPAL2 0.087241983 2.130415251 0.040950694 MiPAL7-MiPAL1 0.084734441 2.184662425 0.038786057 MiPAL5-MiPAL1 0.111913996 — — MiPAL8-MiPAL1 0.02388762 0.400106007 0.059703227 MiPAL1-MiPAL3 0.004306158 0.029084482 0.148056903 MiPAL7-MiPAL8 0.086248659 1.970777229 0.043763779 MiPAL5-MiPAL8 0.1112944 2.773517674 0.040127525 MiPAL7-MiPAL5 0.118588566 2.625444505 0.045168948 Ka:异义替换;Ks:用义替换。
Ka: Ambiguous substitution; Ks: meaning substitution.2.5 MiPPO染色体分布、物种间共线性和选择压分析
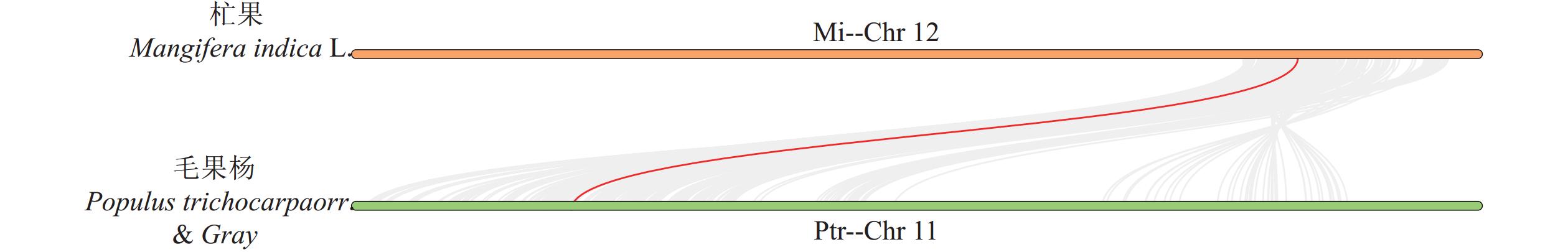
为了探究MiPPO1的进化过程,绘制MiPPO与PtrPPO基因的共线性图(图7)。MiPPO1定位于染色体Chr12,与定位于毛果杨染色体Chr11的PtrPPO13为同源基因,表3结果表明这对同源基因的Ka/Ks值小于1(
0.169943619 ),说明进化过程中主要受到纯化选择压力的影响。2.6 顺式作用元件分析
对MiPPO、MiPAL基因家族成员启动子中顺式作用元件进行可视化分析,结果如图8所示,每一个MiPAL家族成员启动子区域除了包含大量的上游元件(TATA-box、CAAT-box),还包含大量的光响应元件(G-box、GT1-motif、P-box、Box 4、Gap-box、TCT-motif、GA-motif、I-box、chs-CMA1a、MRE、ACE、3-AF1 binding site、ATC-motif、LS7、ATCT-motif、AT1-motif、GATA-motif)、脱落酸响应元件(EBRE)、抗氧化反应元件(ARE),茉莉酸甲酯响应元件(CGTCA-motif、TGACG-motif)。MiPAL1、2、4、8的启动子序列上存在AT-rich DNA结合蛋白(ATBP-1)的结合位点(AT-rich element),MiPAL1、2、3、4、5的启动子序列上存在细胞周期调节元件(MSA-like),MiPAL1、5、6的启动子序列上存在昼夜节律调节元件(circadian),MiPAL2的启动子序列上存在栅栏叶肉细胞分化相关元件(HD-Zip 1),MiPAL 7的启动子序列上存在胚乳表达的顺式调控元件(GCN4_motif),MiPAL1、3、4、6的启动子序列上存在分生组织表达调节原件(CAT-box),MiPAL1、2、3、4、7的启动子序列上存在MYBHv1结合位点(CCAAT-box),MiPAL1、2、4、5的启动子序列上存在种子特异性调控元件(RY-element)。
MiPAL1、3、4、8的启动子序列上存在生长素响应元件(AuxRR-core、TGA-element),MiPAL1、2、3、4、7、8的启动子序列上存在赤霉素响应元件(P-box、TATC-box),MiPAL1、4、8的启动子序列上存在水杨酸响应元件(TCA-element)。MiPAL2、3、8的启动子序列上存在防御与逆境应答元件(TC-rich repeats),MiPAL3、5、6、8的启动子序列上存在干旱响应元件(MBS),MiPAL1、2、3、4、6、8的启动子序列上存在低温响应元件(LTR),MiPAL8的启动子序列上存在MYB结合位点与类黄酮合成调节元件(MBSI),MiPAL1、2、3、4、5的启动子序列上存在存在机械伤害反应元件(WUN-motif)。可见不同MiPAL基因家族成员启动子区域所含的顺式作用元件类型差异较小,其中MiPAL1存在26种不同类型的顺式启动子元件,包括生长发育、胁迫响应、光响应、激素响应等四大方面,说明其功能较多,可能在杧果生长发育过程中具备重要的作用;MiPAL8除械伤害反应元件外,其余激素与胁迫响应元件都存在其启动子序列中,且特有存在MYB结合位点与类黄酮合成调节元件,说明MiPAL8在杧果响应逆境胁迫与类黄酮合成中起重要作用。
MiPPO1的启动子序列上同样包含大量的上游元件(TATA-box、CAAT-box)与大量的光响应元件(3-AF1 binding site、G-box、Box 4、chs-CMA2a、TCT-motif、GA-motif、I-box);MiPPO1包含的激素响应元件包括脱落酸响应元件(ABRE),37个启动子序列上存在生长素响应元件(TGA-element),茉莉酸甲酯响应元件(CGTCA-motif、TGACG-motif),水杨酸响应元件(TCA-element);胁迫响应元件包括抗氧化响应元件(ARE),防御与逆境应答元件(TC-rich repeats),MYB结合位点与类黄酮合成调节元件(MBSI),机械伤害反应元件(WUN-motif);生长调节元件包括昼夜节律调节元件(circadian),胚乳表达的顺式调控元件(GCN4_motif)。MiPPO1的启动子中所含元件种类丰富,且同样存在MYB结合位点与类黄酮合成调节元件,说明其在杧果生长发育过程中具备重要的作用。
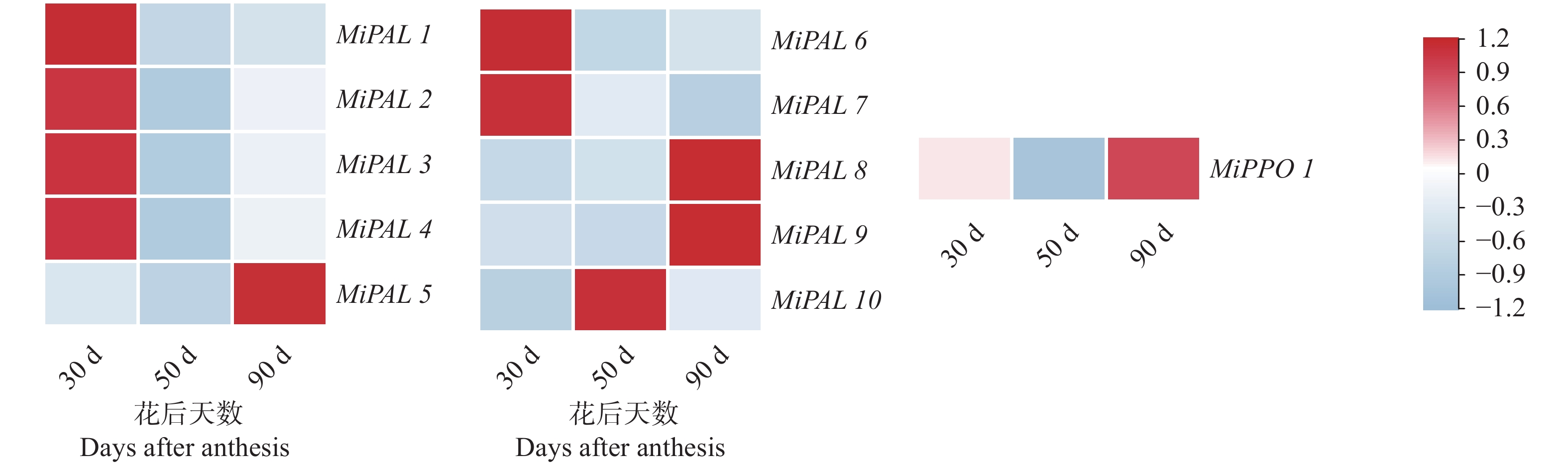
2.7 表达模式分析
根据转录组FPKM值绘制的热图如图9所示,MiPAL5、8、9在果实成熟的后期呈现上调表达,MiPAL10在果实成熟的中期呈现上调表达,其余MiPAL成员在幼果期呈现上调表达;MiPPO1在幼果期与成熟期呈现上调表达,中期呈现下调表达。由此可见,MiPAL与MiPPO家族成员在果实生长发育不同阶段受不同生长发育信号调控,为诱导型表达。为了进一步验证MiPAL、MiPPO在杧果多酚,选择MiPAL1、3、6、7进行qPCR试验,结果如图10所示。随果实的成熟,4个基因呈现两种不同的表达模式,MiPAL1、3、6在花后50 d与花后30 d相比,呈现下调表达,在花后91 d,表达量上调,无显著差异;MiPOD7随果实成熟呈现下调表达,表达量无显著差异。4个基因的相对表达量变化模式与转录组分析结果一致。
3. 讨论与结论
MiPAL基因家族成员蛋白理化性质和序列组成特征与巴西橡胶树(Ficus elastica Roxb. ex Hornem.)PAL(HbPAL)基因家族相似[25],但是HbPAL均定位于细胞质中,除MiPAL2、MiPAL5以外的MiPAL则大部分定位于叶绿体中。MiPAL成员数量与野生番茄[Solanum pimpinellifolium (Jusl) Mill][26]、黄瓜(Cucumis sativus L.)[27]、银杏(Ginkgo biloba L.)[28]等均为11个,少于栽培番茄(Solanum lycopersicum L.)(14个)与油菜(Brassica napus L)(17个)[26,29],说明MiPAL基因家族并没有较大的扩张。MiPAL基因家族中7个成员(70%)参与了串联复制,6个成员(60%)间存在共片段重复,MiPAL3、MiPAL7同时存在片段重复与串联重复,鉴于基因家族扩展主要通过多个基因的片段复制、单个基因的串联复制和全基因组复制等3种模式发生,说明MiPAL基因家族扩张由片段复制与串联复制共同促进,且串联重复占主导地位。多物种间的PAL基因进化关系分析结果表明,单子叶植物与双子叶植物的PAL基因可以进行良好的区分,双子叶植物中还可以按木本与草本进行更加细致的分组,与前人研究结果相同,说明PAL基因高度保守,基因功能的分化早于单/双子叶植物的分化[30]。
杧果与木薯(Manihot esculenta Crantz)和蓖麻豆(Ricinus communis L.)PPO基因家族成员数量均为1个,远少于大豆(Glycine max L. Merrill)(11个)和杨树(Populus L.)(11个),表明MiPPO基因家族可能因功能冗余或其他同工酶增多等而出现成员的减少[31]。多物种间的PPO进化关系分析结果表明,MiPPO与PtrPPO10、PtrPPO12、PtrPPO13亲缘关系较近,且与PtrPPO13具有相同的保守结构和成为一对同源基因。基于PtrPPO10、PtrPPO12保守结构域缺失,仅含有PPO1-KFDV[32],推测其他MiPPO家族成员发生了保守结构域完全缺失变异而消失,仅剩MiPPO1保存完整结构域。MiPAL与MiPPO的同源基因对之间Ka/Ks值小于1,说明进化过程中主要受到纯化选择压力的影响。
MiPAL、MiPPO启动子序列中包含大量的光信号诱导、激素、生长发育和胁迫相关的元件,推测MiPAL、MiPPO在杧果的生长发育、响应外界刺激等过程中发挥主要作用;MiPAL8与MiPPO启动子序列上都存在类黄酮合成调控元件,说明两者还在杧果类黄酮及其相关代谢过程中起重要作用。因此,MiPAL与MiPPO在杧果生长发育期呈现诱导型表达的特征,存在多因素的调控模式。qPCR验证结果表明MiPAL7的差异表达与杧果果肉多酚积累变化均呈随杧果成熟而下降的趋势,说明MiPAL7参与了杧果果肉中多酚积累。
综上所述,MiPAL基因家族成员基因结构较为保守,扩张由串联重复主导;MiPPO可能因基因功能冗余或保守结构域缺失而使其成为单拷贝基因。不同MiPAL基因家族成员和MiPPO因表达调控因素复杂而具有不同的表达模式。MiPAL7在杧果果肉多酚积累代谢中发挥关键作用。今后需要进一步将上述诱导表达条件对MiPAL基因家族成员和MiPPO表达调控机理进行深入研究。
-
图 4 杧果PAL基因结构分析
A:基因结构分析; B:保守结构域分析; C:保守基序分析;图5同。
Figure 4. Structure of PAL in M. indica
A: Gene structure analysis; B: conservative structural domain analysis; C: conservative motif analysis. Same for Fig. 5.
表 1 杧果MiPAL及内参基因引物序列
Table 1 Sequences of primers of MiPALs and reference genes
基因名
Gene name上游引物序列 (5′-3′)
Primer sequence (5′-3′)下游引物序列 (5′-3′)
Primer sequence (5′-3′)MiPAL1 AATGGCCAACGGAGAGAGAG TGGGGACTTTACTTTCTCACCA MiPAL3 ACCAATCATGTCCAGAGCGC CCTCCAAGTGCCTCAAGTCA MiPAL6 GACCCCTGCAGTGCTACTTA ATCGCCGCATTTCCATTCTC MiPAL7 CCAATCCAGTCACCAGCCAT TGCCTCAGGTCAATCGCTTG Actin7 ATCTGCTGGAAGGTGCTGAG CCAAGCAGCATGAAGATCAA 表 2 MiPAL、MiPPO蛋白基本信息
Table 2 Basic information on MiPAL and MiPPO proteins
基因
Gene基因编号
Gene ID氨基酸数量
Number of amino acids分子量
MW/Da等电点
pI不稳定系数
Instability index脂肪系数
Aliphatic index平均亲水系数
GRAVY亚细胞定位
Subcellular localizationMiPAL1 LOC123193566.1 707 77066.14 6.47 35.46 90.11 −0.151 叶绿体 MiPAL2 LOC123201898.1 704 76781.74 6.26 32.8 91.58 −0.184 细胞质 MiPAL3 LOC123206701.1 707 76933.96 6.46 34.55 90.11 −0.144 叶绿体 MiPAL4 LOC123206728.1 707 77051.13 6.57 35.45 90.11 −0.151 叶绿体 MiPAL5 LOC123209119.1 719 78902.02 6.16 33.16 90.21 −0.229 细胞质 MiPAL6 LOC123224091.1 712 77440.26 5.76 30.42 90.7 −0.179 叶绿体 MiPAL7 LOC123225153.1 708 76938.82 6.15 30.41 92.99 −0.162 叶绿体 MiPAL8 LOC123228411.1 700 76578.56 6.06 36.46 90.6 −0.16 细胞核 MiPAL9 LOC123192215.1 707 76933.96 6.46 34.55 90.11 −0.144 叶绿体 MiPAL10 LOC123224083.1 712 77333.39 6.1 27.11 92.49 −0.163 内质网 MiPPO1 LOC123193265.1 593 66775.87 6.95 37.57 76.12 −0.501 叶绿体 表 3 杧果PAL复制基因对的Ka、Ks分析
Table 3 Ka and Ks analysis on PAL replication pairs of M. indica
同源基因
HomologsKa Ks Ka/Ks MiPAL7-MiPAL2 0.029459743 0.421135092 0.06995319 MiPAL5-MiPAL2 0.118273297 2.076835221 0.056948811 MiPAL8-MiPAL2 0.09078887 1.857097035 0.048887521 MiPAL1-MiPAL2 0.087241983 2.130415251 0.040950694 MiPAL7-MiPAL1 0.084734441 2.184662425 0.038786057 MiPAL5-MiPAL1 0.111913996 — — MiPAL8-MiPAL1 0.02388762 0.400106007 0.059703227 MiPAL1-MiPAL3 0.004306158 0.029084482 0.148056903 MiPAL7-MiPAL8 0.086248659 1.970777229 0.043763779 MiPAL5-MiPAL8 0.1112944 2.773517674 0.040127525 MiPAL7-MiPAL5 0.118588566 2.625444505 0.045168948 Ka:异义替换;Ks:用义替换。
Ka: Ambiguous substitution; Ks: meaning substitution. -
[1] IQUEBAL M A, JAISWAL S, MAHATO A K, et al. MiSNPDb: A web-based genomic resources of tropical ecology fruit mango (Mangifera indica L. ) for phylogeography and varietal differentiation [J]. Scientific Reports, 2017, 7(1): 14968. DOI: 10.1038/s41598-017-14998-2
[2] 何强, 董静雯. 果蔬中的多酚及其功能特性 [J]. 西华大学学报(自然科学版), 2019, 38(4):37−44. DOI: 10.3969/j.issn.1673-159X.2019.04.006 HE Q, DONG J W. Polyphenols in fruits and vegetables and its function [J]. Journal of Xihua University (Natural Science Edition), 2019, 38(4): 37−44. (in Chinese) DOI: 10.3969/j.issn.1673-159X.2019.04.006
[3] MUHAMMAD N, LUO Z, ZHAO X, et al. Transcriptome-wide expression analysis of MYB gene family leads to functional characterization of flavonoid biosynthesis in fruit coloration of Ziziphus Mill [J]. Frontiers in Plant Science, 2023, 14: 1171288. DOI: 10.3389/fpls.2023.1171288
[4] LI G, QIN B B, LI S D, et al. LbNR-derived nitric oxide delays Lycium fruit coloration by transcriptionally modifying flavonoid biosynthetic pathway [J]. Frontiers in Plant Science, 2020, 11: 1215. DOI: 10.3389/fpls.2020.01215
[5] ZHANG A A, ZHENG J, CHEN X M, et al. Comprehensive analysis of transcriptome and metabolome reveals the flavonoid metabolic pathway is associated with fruit peel coloration of melon [J]. Molecules, 2021, 26(9): 2830. DOI: 10.3390/molecules26092830
[6] LI S J, LI X, WANG X D, et al. Flavonoid synthesis-related genes determine the color of flower petals in Brassica napus L [J]. International Journal of Molecular Sciences, 2023, 24(7): 6472. DOI: 10.3390/ijms24076472
[7] ALVAREZ-RIVERA G, SANZ A, CIFUENTES A, et al. Flavonoid accumulation varies in Medicago truncatula in response to mercury stress [J]. Frontiers in Plant Science, 2022, 13: 933209. DOI: 10.3389/fpls.2022.933209
[8] MMBANDO G S. The recent relationship between ultraviolet-B radiation and biotic resistance in plants: A novel non-chemical strategy for managing biotic stresses [J]. Plant Signaling & Behavior, 2023, 18(1): 2191463.
[9] ZHANG Q, ZHENG G Y, WANG Q, et al. Molecular mechanisms of flavonoid accumulation in germinating common bean (Phaseolus vulgaris) under salt stress [J]. Frontiers in Nutrition, 2022, 9: 928805. DOI: 10.3389/fnut.2022.928805
[10] WANG J, GAO X Q, WANG X, et al. Exogenous melatonin ameliorates drought stress in Agropyron mongolicum by regulating flavonoid biosynthesis and carbohydrate metabolism [J]. Frontiers in Plant Science, 2022, 13: 1051165. DOI: 10.3389/fpls.2022.1051165
[11] GOURLAY G, MA D W, SCHMIDT A, et al. MYB134-RNAi poplar plants show reduced tannin synthesis in leaves but not roots, and increased susceptibility to oxidative stress [J]. Journal of Experimental Botany, 2020, 71(20): 6601−6611. DOI: 10.1093/jxb/eraa371
[12] DIXIT G, SRIVASTAVA A, RAI K M, et al. Distinct defensive activity of phenolics and phenylpropanoid pathway genes in different cotton varieties toward chewing pests [J]. Plant Signaling & Behavior, 2020, 15(5): 1747689.
[13] CHEN Q, LIANG X, WU C L, et al. Overexpression of leucoanthocyanidin reductase or anthocyanidin reductase elevates tannins content and confers cassava resistance to two-spotted spider mite [J]. Frontiers in Plant Science, 2022, 13: 994866. DOI: 10.3389/fpls.2022.994866
[14] 朱三明, 郑敏敏, 田恬, 等. 植物次生代谢途径与调控研究进展 [J]. 植物生理学报, 2023(12):2188−2216. ZHU S M, ZHEN M M, TIAN T, et al. Research progress on plant secondary metabolism and regulation [J]. Plant Physiology Journal, 2023(12): 2188−2216.
[15] 高媛, 马帅, 代敏, 等. 果蔬酚酸生物合成及代谢调控研究进展 [J]. 食品科学, 2018, 39(9):286−293. DOI: 10.7506/spkx1002-6630-201809043 GAO Y, MA S, DAI M, et al. Progress in research on the biosynthesis pathway and metabolic regulation of phenolic acids [J]. Food Science, 2018, 39(9): 286−293. (in Chinese) DOI: 10.7506/spkx1002-6630-201809043
[16] BARROS J, DIXON R A. Plant phenylalanine/tyrosine ammonia-lyases [J]. Trends in Plant Science, 2020, 25(1): 66−79. DOI: 10.1016/j.tplants.2019.09.011
[17] LIU W X, FENG Y, YU S H, et al. The flavonoid biosynthesis network in plants [J]. International Journal of Molecular Sciences, 2021, 22(23): 12824. DOI: 10.3390/ijms222312824
[18] 甄文娜. UV-B处理促进鲜切胡萝卜酚类物质富集的调控机理研究[D]. 山东: 齐鲁工业大学, 2024. ZHEN W N. Study on the regulation mechanism of UV-B treatment promoting the enrichment of phenols in fresh-cut carrots[D]. Shandong: Qilu University of Technology, 2024.
[19] ZHANG S W, QI X L, ZHU R Y, et al. Transcriptome analysis of Salvia miltiorrhiza under drought stress [J]. Plants, 2024, 13(2): 161. DOI: 10.3390/plants13020161
[20] JIANG S, WENG B S, LIU T, et al. Response of phenolic metabolism to cadmium and phenanthrene and its influence on pollutant translocations in the mangrove plant Aegiceras corniculatum (L. ) Blanco (Ac) [J]. Ecotoxicology and Environmental Safety, 2017, 141: 290−297. DOI: 10.1016/j.ecoenv.2017.03.041
[21] JAŃCZAK-PIENIĄŻEK M, CICHOŃSKI J, MICHALIK P, et al. Effect of heavy metal stress on phenolic compounds accumulation in winter wheat plants [J]. Molecules, 2022, 28(1): 241. DOI: 10.3390/molecules28010241
[22] Bibhuti Bhusan Mishra Bibhuti x. Polyphonel oxidases: Biochemical and molecular characterization, distribution, role and its control [J]. Enzyme Engineering, 2016, 5(1): 141−149.
[23] GANDÍA-HERRERO F, GARCÍA-CARMONA F. Biosynthesis of betalains: Yellow and violet plant pigments [J]. Trends in Plant Science, 2013, 18(6): 334−343. DOI: 10.1016/j.tplants.2013.01.003
[24] ONO E, HATAYAMA M, ISONO Y, et al. Localization of a flavonoid biosynthetic polyphenol oxidase in vacuoles [J]. Plant Journal, 2006, 45(2): 133−143. DOI: 10.1111/j.1365-313X.2005.02625.x
[25] LIU H, HE Q G, HU Y Y, et al. Genome-wide identification and expression profile analysis of the phenylalanine ammonia-lyase gene family in Hevea brasiliensis [J]. International Journal of Molecular Sciences, 2024, 25(9): 5052. DOI: 10.3390/ijms25095052
[26] ZHANG F L, WANG J, LI X G, et al. Genome-wide identification and expression analyses of phenylalanine ammonia-lyase gene family members from tomato (Solanum lycopersicum) reveal their role in root-knot nematode infection [J]. Frontiers in Plant Science, 2023, 14: 1204990. DOI: 10.3389/fpls.2023.1204990
[27] AMJAD M, WANG Y X, HAN S M, et al. Genome wide identification of phenylalanine ammonia-lyase (PAL) gene family in Cucumis sativus (cucumber) against abiotic stress [J]. BMC Genomic Data, 2024, 25(1): 76. DOI: 10.1186/s12863-024-01259-1
[28] GAO X G, HU Y P, XU Z B, et al. Expression profiling of the phenylalanine ammonia-lyase (PAL) gene family in ginkgo biloba L [J]. Plant Signaling & Behavior, 2023, 18(1): 2271807.
[29] ZHANG H Y, ZHANG X H, ZHAO H X, et al. Genome-wide identification and expression analysis of phenylalanine ammonia-lyase (PAL) family in rapeseed (Brassica napus L. ) [J]. BMC Plant Biology, 2023, 23(1): 481. DOI: 10.1186/s12870-023-04472-9
[30] RAWAL H C, SINGH N K, SHARMA T R. Conservation, divergence, and genome-wide distribution of PAL and POX A gene families in plants[J]. International Journal of Genomics, 2013, 2013: 678969.
[31] TRAN L T, TAYLOR J S, CONSTABEL C P. The polyphenol oxidase gene family in land plants: Lineage-specific duplication and expansion [J]. BMC Genomics, 2012, 13: 395. DOI: 10.1186/1471-2164-13-395
[32] HE F, SHI Y J, ZHAO Q, et al. Genome-wide investigation and expression profiling of polyphenol oxidase (PPO) family genes uncover likely functions in organ development and stress responses in Populus trichocarpa [J]. BMC Genomics, 2021, 22(1): 731. DOI: 10.1186/s12864-021-08028-9




 下载:
下载: