Cloning and Prokaryotic Expression of Cassava MePOD10
-
摘要:目的
克隆木薯(Manihot esculenta Crantz)过氧化物酶基因MePOD10并进行生物信息学分析、原核表达分析和蛋白酶动力学分析,为进一步研究MePOD10基因的功能提供参考。
方法从木薯‘华南124’('South China 124', 'SC124')中扩增MePOD10基因编码区序列(coding sequence, CDS),对其进行生物信息学分析,并构建MePOD10-pET28a融合表达载体,再转化至BL21感受态细胞中进行蛋白诱导,通过SDS-PAGE以及Western blotting确定MePOD10蛋白的表达情况,纯化MePOD10蛋白后进行酶活性和动力学分析。
结果MePOD10基因CDS序列长度为981 bp,编码326个氨基酸,蛋白分子量为
35069.60 Da,理论等电点为6.59,属于不稳定疏水性蛋白。MePOD10含有植物过氧化物酶保守结构域,其氨基酸序列与橡胶树POD蛋白的相似性最高,为93.25%。在37 ℃、1 mmol·L−1异丙基-β-D-硫代半乳糖苷(Isopropyl β-D-Thiogalactoside, IPTG)诱导6 h条件下,上清和沉淀均有蛋白表达,纯化后的上清蛋白酶活性高于对照蛋白,以愈创木酚为底物时,随着底物浓度增加,MePOD10蛋白催化活性迅速增加,之后趋于平稳,说明MePOD10蛋白具有催化活性。结论MePOD10蛋白具有过氧化物酶(Peroxidase, POD)保守结构域,在37 ℃、1 mmol·L−1IPTG诱导6 h条件下,MePOD10-pET28a融合蛋白能正确表达,纯化后的融合蛋白具有催化活性。
Abstract:ObjectiveThe key gene associated with Class III peroxidase (POD) in stress responses of animals and plants, MePOD10 from cassava was cloned for bioinformatics, prokaryotic expression, and protease kinetics analyses.
MethodsThe coding sequence (CDS) of MePOD10 of South China 124 cassava cultivar (SC124) was amplified and subjected to bioinformatics analysis. A MePOD10-pET28a fusion expression vector was constructed and transformed into BL21 competent cells for protein induction.The protein was confirmed on its expression by SDS-PAGE and western blot and purified for enzymatic activity determination and kinetic analysis.
ResultsThe length of MePOD10 CDS was 981 bp encoded 326 amino acids with a molecular weight of 35 069.60 Da and a theoretical isoelectric point of 6.59. The unstable and hydrophobic MePOD10 contained a conserved domain of plant POD sharing the highest amino acid sequence similarity of 93.25% with that of rubber trees. Induced by 1 mmol·L−1 isopropyl-β-D-thiogalactoside (IPTG) at 37 ℃ for 6 h, the protein expressions in the supernatant and precipitate were examined. The purified supernatant showed greater enzymatic activity than control. When guaiacol was used as a substrate, the catalytic activity of MePOD10 increased rapidly with increasing substrate concentration to reach a peak.
ConclusionMePOD10 was determined to contain conserved POD domain. MePOD10-pET28a could be correctly expressed under the induction of 1 mmol·L−1 IPTG at 37 ℃ for 6 h, and the purified fusion protein exhibited a significant catalytic activity.
-
Keywords:
- Cassava /
- peroxidase /
- prokaryotic expression /
- enzyme activity
-
0. 引言
【研究意义】木薯(Manihot esculenta Crantz)为大戟科木薯属直立灌木状多年生热带作物,起源于拉丁美洲[1],被广泛种植于热带和亚热带地区,是全球第六大粮食作物[2],为全球10亿多人提供了主要的食物来源[3]。木薯在种植过程中,面临着多种逆境胁迫,其中木薯花叶病(Cassava mosaic disease, CMD)是木薯最严重的病毒性疾病之一,可导致木薯高达95%的产量损失,是木薯生产的主要制约因素[4]。鉴于此,运用现代生物技术来发掘与逆境胁迫相关的基因,增强木薯的抗逆性和抗病性,显得尤其重要。【前人研究进展】研究人员通过全基因组关联研究(Genome-wide association studies, GWAS)和双亲连锁图谱发现CMD的抗性主要是由12号染色体上的一个主要基因座赋予的,该基因被称为CMD2基因座[5,6]。同时,Rabbi等研究发现CMD2基因座中的两个候选基因MePOD10(Manes.12G076200)和MePOD58(Manes.12G076300)与CMD的抗性紧密相关[7]。过氧化物酶是广泛存在于各种动物、植物和微生物体内的一类氧化酶[8]。其按照来源可分为3类,分别是Class Ⅰ、Class Ⅱ和Class Ⅲ[9]。第一类POD(Class Ⅰ POD),作为广泛存在于植物、真菌以及原核生物中的一种过氧化物酶,其主要功能是清除体内过多的过氧化氢,并在此过程中调节细胞的氧化还原稳态[10]。第二类POD(Class Ⅱ POD)主要存在于真菌中,并在木质素的降解过程中发挥着至关重要的作用[11]。第三类POD(Class Ⅲ POD)是植物中特有的分泌型过氧化物酶,在抵抗逆境胁迫的过程中发挥着重要作用[12]。在植物的生长及发育过程中,Class Ⅲ POD涉及了很多的生理生化反应,如参与生长素的分解代谢、细胞壁的合成代谢[13]、活性氮的代谢[14]、防御病原体[15]、木质素化和木栓形成[16]、活性氧(Reactive oxygen species, ROS)清除、伤口愈合和植物抗毒素合成等对生物和非生物胁迫的防御反应[17]。【本研究切入点】前人通过GWAS在CMD2基因座找到的MePOD10和MePOD58尚未在木薯中探究其功能,且有关木薯POD基因克隆和功能分析的研究相对较少。【拟解决的关键问题】本研究拟克隆MePOD10基因,利用生物信息学工具对其编码蛋白序列进行分析,并构建原核表达载体确定其蛋白表达条件,为深入探究MePOD10蛋白的功能奠定理论基础。
1. 材料与方法
1.1 试验材料
选用木薯品种SC124作为试验材料,其种植于海南大学儋州校区种植实验苗圃基地(19°50'N, 109°49'E),原核表达载体pET28a由实验室自行保存。植物总RNA提取试剂盒、异丙基-β-D-硫代吡喃半乳糖苷(IPTG)、DNA回收试剂盒购自天根生化科技有限公司、His标签蛋白纯化试剂盒购自上海碧云天生物技术股份有限公司、POD酶活测定试剂盒购自南京建成生物工程研究所、愈创木酚购自上海麦克林生化科技股份有限公司等。
1.2 木薯总RNA的提取和反转录
从SC124健康植株上剪取适量新鲜叶片,将其剪碎置于研钵内,加入少许液氮研磨成细腻粉状,经RNA提取试剂盒提取木薯总RNA后,再反转为cDNA,−80 ℃保存备用。
1.3 基因的克隆
在Phytozome网站(https://phytozome.jgi.doe.gov)查询MePOD10基因(Manes.12G076300)的CDS序列,用Primer 5.0软件设计特异性引物(表1),用cDNA作为模板扩增MePOD10基因CDS序列。经电泳跑胶验证PCR产物有目的条带后,切胶回收目的条带并将其构建于pEASY-Blunt3载体上,转化后挑选边缘清晰、大小适中且明亮的单菌落,摇菌并提取质粒送测序,将测序正确的单克隆菌液于−40 ℃保存备用。
表 1 供试引物序列Table 1. Primers applied引物名称
Primer name引物序列
Primer sequence引物用途
Primer usageMePOD10-F
MePOD10-R5'-GGAATTCATGGGAGGTTTTGGTTCTTT-3'
5'-GGATCCTCAACTATTGATCACTGCAC-3'基因克隆
Gene cloningMePOD10-pET28a-F
MePOD10-pET28a-R5'-ATGGGTCGCGGATCCATGGGAGGTTTTGGTTCTTT-3'
5'-GTCGACGGAGCTCGAATTCCAGGTCCTCCTCTGAGA
TCAGCTTCTGCTCCTCACTATTGATCACTGCAC-3'原核表达
Prokaryotic expression1.4 生物信息学分析
MePOD10基因编码的蛋白理化特性由ExPASy(http://web.expasy.org/protparam/)分析,MePOD10蛋白保守结构域利用NCBI(https://www.ncbi.nlm.nih.gov/)的CDD数据库分析。MePOD10蛋白的二级和三级结构利用Prabi(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)和SWISS-MODEL(https://swissmodel.expasy.org/interactive)预测。通过NCBI数据库BLAST搜索MePOD10基因同源序列,并确定其开放阅读框(Open reading frame, ORF)。由MEGA7.0翻译MePOD10基因CDS序列,与其他物种进行同源比对分析后构建系统发育进化树。
1.5 原核表达载体的构建
以MePOD10-Blunt3质粒为模板,扩增MePOD10-pET28a片段,将扩增好的片段以同源重组方法构建于pET28a载体上,转化后挑选边缘清晰、大小适中且明亮的单菌落,摇菌并提取质粒送测序,将测序正确的单克隆菌液−40 ℃保存备用。
1.6 重组蛋白的诱导表达
将构建成功的MePOD10-pET28a质粒转入BL21(DE3)感受态细胞中,选取PCR检测无误的单克隆作为表达菌,将表达菌接种于LB培养基中,利用紫外分光光度计将表达菌液初始D600调至0.2~0.3,置于37 ℃摇床,表达菌液摇至D600为0.6~0.8,加入IPTG至终浓度为1 mmol·L−1并置于37 ℃摇床,200 r·min−1条件下诱导。取诱导6 h的重组蛋白为样品,经离心后取上清和沉淀加入上样缓冲液后充分涡旋,SDS-PAGE检测。
1.7 重组蛋白的Western blotting验证
经SDS-PAGE电泳后,进行转膜,转完的膜经1×TBS 漂洗后置于15 mL的脱脂牛奶中,室温下摇床轻晃1 h。每隔10 min用适量的1×TBS洗膜3次,加入Myc抗体室温孵育2 h,每隔10 min用适量的1×TBS洗膜3次,加入二抗室温孵育1 h,最后用A、B显影液进行显影成像。
1.8 重组蛋白的纯化和酶活测定
选用上述诱导条件对MePOD10重组蛋白进行大量诱导,将诱导后的菌液离心,用磷酸缓冲溶液重悬菌体,功率25%、冰上超声破碎10 min后离心得到的上清用试剂盒纯化。用POD酶活试剂盒测定MePOD10蛋白酶活性,pET28a空载蛋白作对照,重复3次。以不同浓度愈创木酚(0、10、20、30、40、50 mmol·L−1)为底物设置5组试验,以磷酸缓冲溶液作对照调零,在反应温度37 ℃、pH5.8条件下,取5 mL反应混合液(磷酸缓冲溶液3 mL、不同浓度的愈创木酚1 mL和1 mL 2%过氧化氢,摇匀),每组各加入1 mL纯化后的MePOD10蛋白,立即摇匀,并迅速倒入比色皿中,于470 nm波长下比色,1 min后记录各组吸光度值∆A,重复3次。按下式计算酶反应初始速度V0:
V0=ΔA⋅Vtε⋅Δt⋅Vs⋅c ∆A为反应初速度阶段内吸光度的变化;Vt为提取酶液总体积(mL);ε为愈创木酚的摩尔吸光系数(mol·L−1·cm−1);∆t为反应时间;Vs为测定时取用蛋白体积(mL);c为蛋白浓度(mg·mL−1)。使用GraphPad Prism 8.0拟合酶动力学曲线并进行单因素方差分析。
2. 结果与分析
2.1 MePOD10基因克隆及鉴定
提取SC124木薯新鲜叶片总RNA并反转录成cDNA,以cDNA为模板进行PCR扩增,得到目的基因片段(图1A)。回收的目的片段构建于pEASY-Blunt3载体上,转入DH5α菌株中,菌落PCR选取阳性克隆(图1B),摇菌并提取质粒送测序。经序列比对显示,该阳性克隆长度为981 bp,编码326个氨基酸,鉴定为MePOD10基因。
2.2 生物信息学分析
2.2.1 MePOD10编码蛋白理化性质和保守结构域分析
使用在线工具Expasy对MePOD10蛋白进行预测分析,结果显示MePOD10的分子式为C
1534 H2434 N434O481S13,分子量为35069.60 Da,接近中性的理论等电点为6.59。Prabi预测结果显示,MePOD10蛋白由α-螺旋、伸展链和不规则卷曲组成蛋白二级结构。MePOD10蛋白不稳定性指数为42.22,GRAVY指数(总平均疏水性)为−0.067,表明其是一种不稳定疏水性蛋白。WoLF PSORT II预测结果显示,MePOD10基因主要定位于细胞核膜上。NCBI-CDD数据库对MePOD10蛋白保守结构域预测,结果显示在24~324 bp区间存在一个POD保守结构域。2.2.2 多序列比对与进化分析
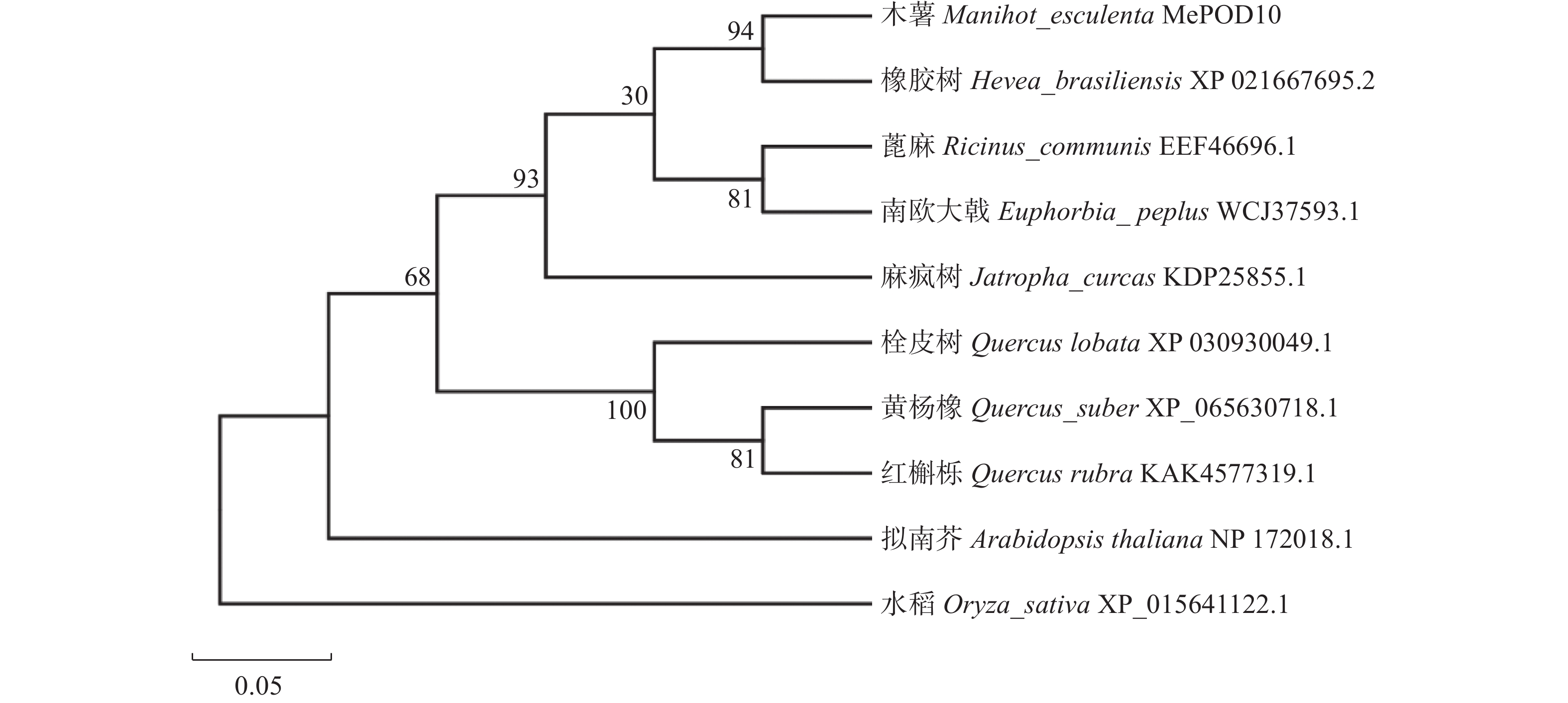
通过NCBI数据库中的BLAST工具对MePOD10的氨基酸序列进行比对分析,筛选与木薯MePOD10蛋白具有较高同源性的氨基酸序列,结果(图2)显示MePOD10与橡胶树(Hevea brasiliensis; XP_021667695.2)的POD蛋白氨基酸序列同源性最高,为93.25%。此外,与麻疯树(Jatropha curcas; KDP25855.1)和蓖麻(Ricinus communis; EEF46696.1)的同源性也较高,分别为88.04%和87.93%。系统发育进化树显示,MePOD10与橡胶树的POD蛋白亲缘关系最近(图3),表明两者在进化过程中保持了较高的序列相似性和功能保守性。
2.3 MePOD10原核表达载体的构建及鉴定
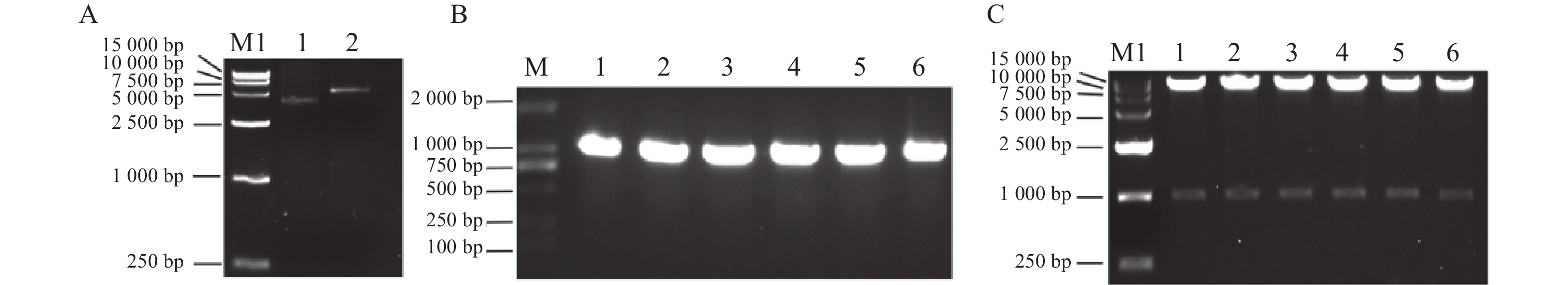
利用同源重组方法将MePOD10基因构建于EcoRⅠ和BamHⅠ双酶切好的pET28a载体(图4A)上,再转化至DH5α感受态细胞中,涂板后挑取6个单克隆进行PCR验证,得到条带大小与目的片段一致的阳性克隆(图4B),将阳性单克隆摇菌后碱裂解法提取质粒,再双酶切质粒验证(图4C)。
![]() 图 4 MePOD10-pET28a载体构建M:DL2000 DNA标记,M1: 15000 DNA标记;A:1为pET28a空载,2为酶切pET28a;B:同源重组后菌落PCR;1~6为PCR检验;C:1~6为酶切6个MePOD10-pET28载体。Figure 4. Construction of MePOD10-pET28a vectorM: DL2000 DNA marker; M1: 15,000 DNA marker; A: 1: empty vector of pET28a; 2: enzyme-digested pET28a; B: PCR of homologous recombinant colonies; 1-6: PCR tests; C: 6 digested MePOD10-pET28a vectors.
图 4 MePOD10-pET28a载体构建M:DL2000 DNA标记,M1: 15000 DNA标记;A:1为pET28a空载,2为酶切pET28a;B:同源重组后菌落PCR;1~6为PCR检验;C:1~6为酶切6个MePOD10-pET28载体。Figure 4. Construction of MePOD10-pET28a vectorM: DL2000 DNA marker; M1: 15,000 DNA marker; A: 1: empty vector of pET28a; 2: enzyme-digested pET28a; B: PCR of homologous recombinant colonies; 1-6: PCR tests; C: 6 digested MePOD10-pET28a vectors.2.4 MePOD10重组蛋白的原核表达
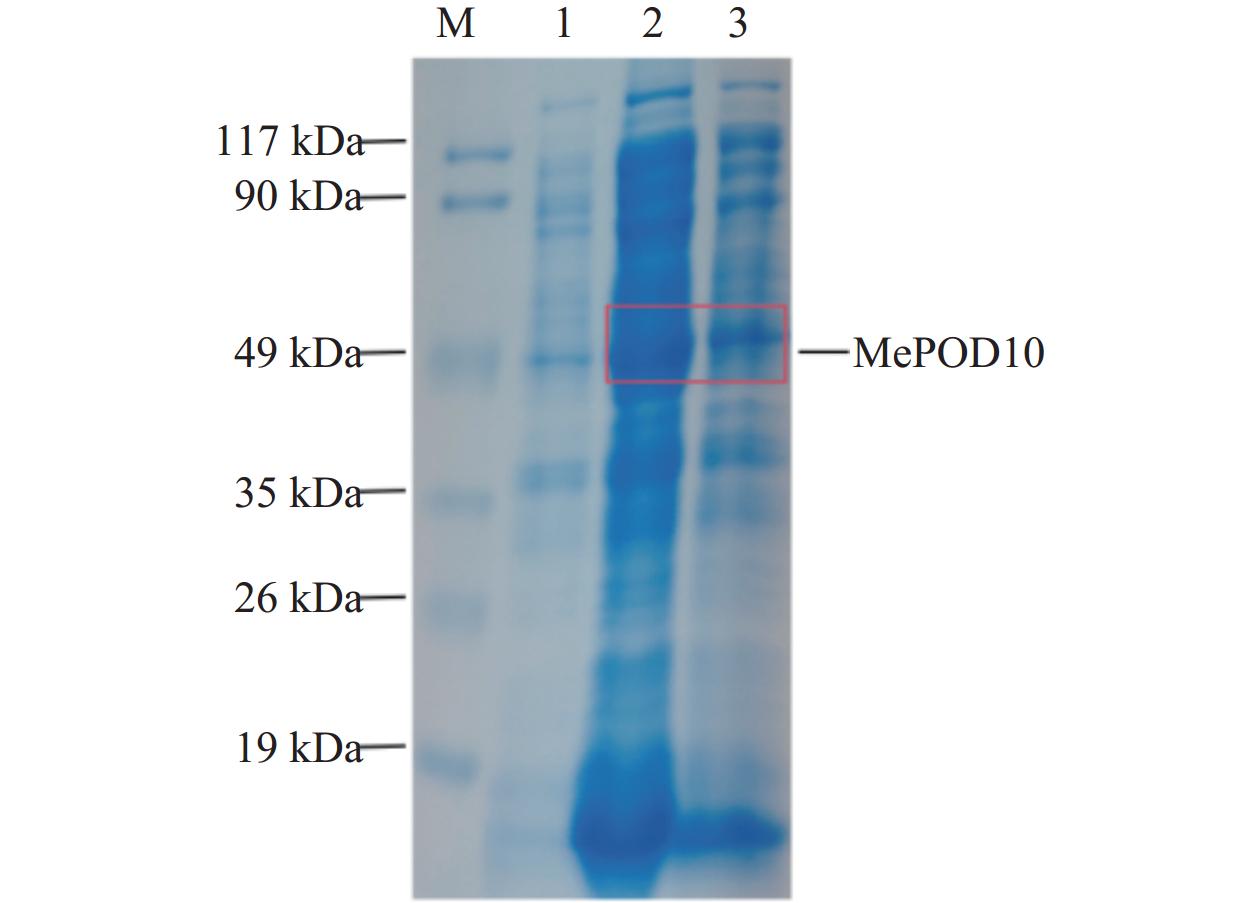
将验证正确的重组质粒转入BL21(DE3)感受态细胞中,在诱导温度为37 ℃,D600值为0.6~0.8条件下,加入IPTG至终浓度为1 mmol·L−1,并在200 r·min−1条件下培养。诱导蛋白表达6 h后,进行SDS-PAGE凝胶电泳检测。结果发现,在49 kDa之间有特异性条带,与预期的MePOD10重组蛋白大小一致(图5)。
2.5 MePOD10重组蛋白的Western blotting验证
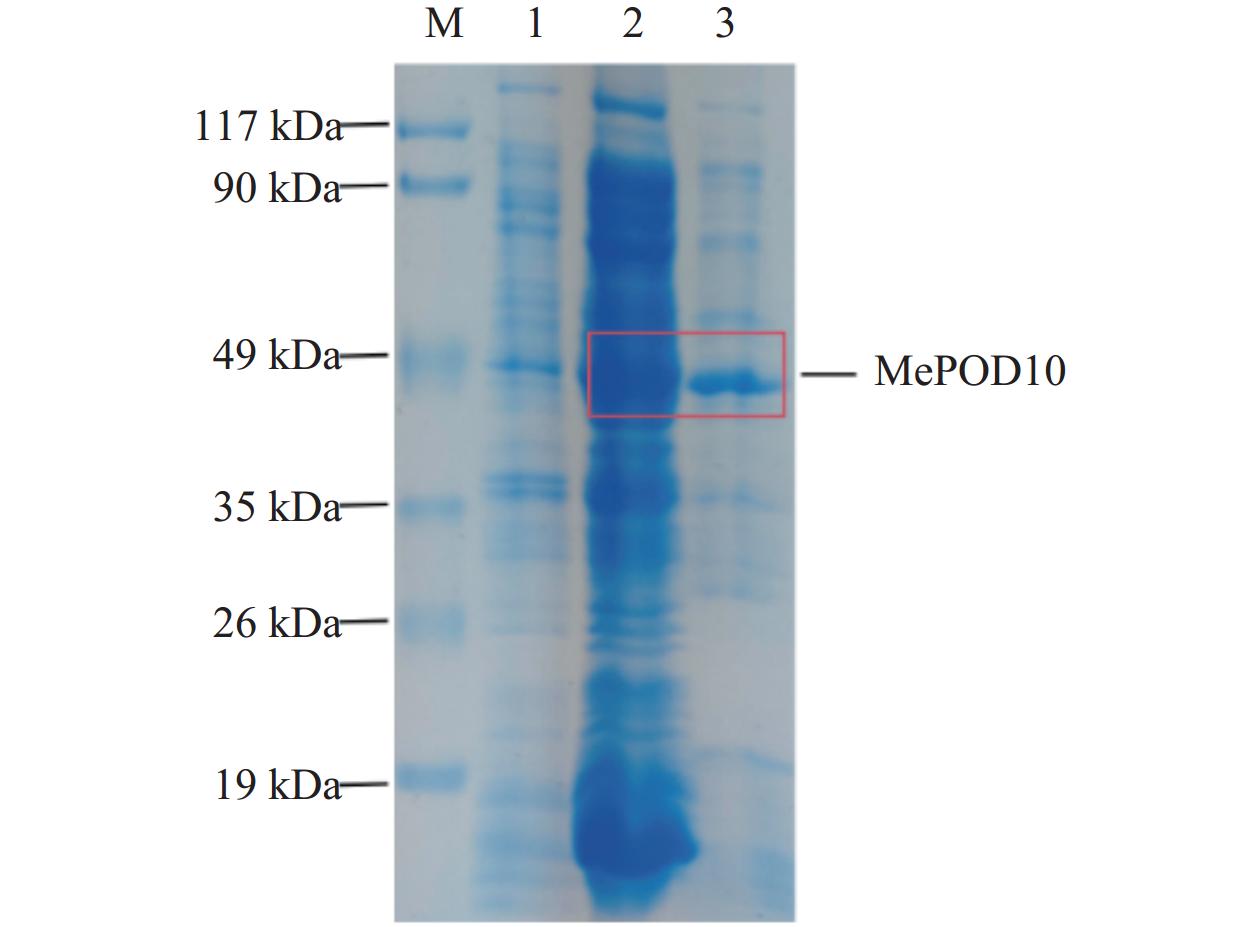
为进一步明确MePOD10重组蛋白表达的准确性,本研究利用Myc抗体,对收集的重组蛋白上清和沉淀进行Western blotting检测。结果表明:重组蛋白在49 kDa处出现特异性条带,说明重组蛋白表达成功(图6)。
2.6 MePOD10重组蛋白的纯化及酶活性测定
根据上述诱导条件对MePOD10重组蛋白大量诱导及纯化,获得纯化的蛋白(图7),大小与MePOD10重组蛋白大小一致,约49 kDa。由MePOD10重组蛋白的酶活性检测结果(图8A)可知,MePOD10蛋白酶活性显著高于对照蛋白(P<0.0001),说明MePOD10重组蛋白具有催化活性。以愈创木酚为底物的MePOD10蛋白酶动力学特性结果(图8B)表明,随着愈创木酚浓度增加,蛋白催化活性迅速增加,后趋于平稳,说明MePOD10蛋白具有POD酶活性。MePOD10蛋白Km值为3.113 mmol·L−1,表明MePOD10蛋白对愈创木酚亲和力大,酶促反应容易进行。
![]() 图 8 MePOD10蛋白酶活性和动力学分析A:对照蛋白(pET28a)和MePOD10蛋白酶活测定;B:以愈创木酚为底物时,MePOD10蛋白酶学动力曲线。****表示差异显著(P<0.0001)。Figure 8. Enzymatic activity and kinetic analysis of MePOD10A: enzymatic activities of pET28a (control) and MePOD10; B: kinetics of MePOD10 protein observed with guaiacol as substrate. ****: significant difference at P<0.0001.
图 8 MePOD10蛋白酶活性和动力学分析A:对照蛋白(pET28a)和MePOD10蛋白酶活测定;B:以愈创木酚为底物时,MePOD10蛋白酶学动力曲线。****表示差异显著(P<0.0001)。Figure 8. Enzymatic activity and kinetic analysis of MePOD10A: enzymatic activities of pET28a (control) and MePOD10; B: kinetics of MePOD10 protein observed with guaiacol as substrate. ****: significant difference at P<0.0001.3. 讨论
随着全球气候的变化,木薯在生长发育的过程中受到各种胁迫与病害的制约,其中木薯花叶病是木薯最严重的病害之一。从经济、环保、稳定性和持久性等长远目光来看,常规分子育种是应对花叶病的有效长期策略[18]。
目前的木薯分子育种研究不仅挖掘到了针对CMD的单基因显性抗性CMD2基因座,还发现了另外两种抗性来源:多基因隐性抗性基因座CMD1以及与CMD2不关联的CMD3基因座。携带CMD2抗性位点的木薯几乎都表现出对木薯花叶病的高度抗性[19],随后有相关研究在CMD2基因座中找到了两个候选基因MePOD10和MePOD58,发现其与木薯花叶病的抗性紧密相关,但其具体的抗病功能还未得到验证。为进一步研究MePOD10在木薯中抗花叶病的功能,本研究构建了原核表达载体MePOD10-pET28a。原核表达载体有多种系列,其中pET是目前使用最为广泛的载体之一[20]。BL21(DE3)也是一种高效且成本较低的表达系统[21]。本研究所选用的pET28a载体和大肠杆菌 BL21(DE3)可以正确、大量地诱导MePOD10蛋白表达,该融合蛋白大小约为49 kDa,符合预测大小。此外,本研究发现在1 mmol·L−1 IPTG、37 ℃、200 r·min−1条件下诱导6 h可以得到较多的目的蛋白,且上清和沉淀均能表达出蛋白。相关研究表明影响大肠杆菌表达的因素有时间、温度等多方面,且37 ℃为大肠杆菌诱导的适宜温度[22]。
POD是植物体内一种非常重要且普遍存在的酶类。POD在植物体内扮演着多重角色,特别是在氧化还原反应和细胞保护方面发挥着关键作用,是植物体内的主要保护酶之一[23]。POD活性随植物生长发育进程以及环境条件的改变而变化,在逆境条件下(如干旱、盐胁迫、病虫害等),植物体内会产生大量的活性氧,导致氧化胁迫加剧。此时,POD等抗氧化酶的活性会显著增强,以清除植物体内过剩的活性氧,减轻氧化损伤的程度。因此,测定POD活性的高低可以作为衡量植物抗逆性强弱的一个重要指标。POD酶活性测定方法有多种,使用广泛且操作简便的方法是愈创木酚法[24]。在酶动力学研究中,Km值是酶特征性常数,可以代表酶与底物的亲和能力,且其大小与酶的性质有关,Km越小,表明酶与底物的亲和力越高,催化作用越大[25]。本研究以不同浓度的愈创木酚为底物时测定MePOD10活性,拟合酶动力学曲线发现,随着底物浓度增加,酶促反应逐渐上升,后趋于平稳,且Km值为3.113 mmol·L−1,表明MePOD10具有POD活性且对愈创木酚的亲和力和催化作用大。本研究初步测定MePOD10蛋白酶活性并拟合酶动力学曲线计算Km值,为进一步探究其蛋白功能奠定基础。
4. 结论
本研究对MePOD10基因进行克隆、原核表达分析以及蛋白酶活性和动力学分析研究,并且对其进行了生物信息学和原核表达情况的初步分析,探索了一些目的蛋白的诱导方法和条件,具有一定的前瞻性和创新性。由于目的蛋白在上清表达量相对沉淀较少,在进一步纯化蛋白试验中目的蛋白纯度较低,因此诱导目的蛋白的更优条件仍有待深入发掘。
-
图 4 MePOD10-pET28a载体构建
M:DL2000 DNA标记,M1: 15000 DNA标记;A:1为pET28a空载,2为酶切pET28a;B:同源重组后菌落PCR;1~6为PCR检验;C:1~6为酶切6个MePOD10-pET28载体。
Figure 4. Construction of MePOD10-pET28a vector
M: DL2000 DNA marker; M1: 15,000 DNA marker; A: 1: empty vector of pET28a; 2: enzyme-digested pET28a; B: PCR of homologous recombinant colonies; 1-6: PCR tests; C: 6 digested MePOD10-pET28a vectors.
图 8 MePOD10蛋白酶活性和动力学分析
A:对照蛋白(pET28a)和MePOD10蛋白酶活测定;B:以愈创木酚为底物时,MePOD10蛋白酶学动力曲线。****表示差异显著(P<0.0001)。
Figure 8. Enzymatic activity and kinetic analysis of MePOD10
A: enzymatic activities of pET28a (control) and MePOD10; B: kinetics of MePOD10 protein observed with guaiacol as substrate. ****: significant difference at P<0.0001.
表 1 供试引物序列
Table 1 Primers applied
引物名称
Primer name引物序列
Primer sequence引物用途
Primer usageMePOD10-F
MePOD10-R5'-GGAATTCATGGGAGGTTTTGGTTCTTT-3'
5'-GGATCCTCAACTATTGATCACTGCAC-3'基因克隆
Gene cloningMePOD10-pET28a-F
MePOD10-pET28a-R5'-ATGGGTCGCGGATCCATGGGAGGTTTTGGTTCTTT-3'
5'-GTCGACGGAGCTCGAATTCCAGGTCCTCCTCTGAGA
TCAGCTTCTGCTCCTCACTATTGATCACTGCAC-3'原核表达
Prokaryotic expression -
[1] OTÁLORA A,GARCES VILLEGAS V,CHAMORRO A,et al. ‘Cassava,manioc or yuca’ (Manihot esculenta) :An overview about its crop,economic aspects and nutritional relevance[J]. Journal of Science with Technological Applications,2024,16:1–10.
[2] 时涛,李超萍,王国芬,等. 中国木薯病害研究进展与展望[J]. 热带作物学报,2023,44(12) :2355−2368. SHI T,LI C P,WANG G F,et al. Research review,current progress and future outlook on cassava diseases in China[J]. Chinese Journal of Tropical Crops,2023,44(12) :2355–2368.(in Chinese)
[3] MOHIDIN S R N S P,MOSHAWIH S,HERMANSYAH A,et al. Cassava(Manihot esculentaCrantz) :A systematic review for the pharmacological activities,traditional uses,nutritional values,and phytochemistry[J]. Journal of Evidence-Based Integrative Medicine,2023,28:2515690X231206227.
[4] CHIKOTI P C,TEMBO M. Expansion and impact of cassava brown streak and cassava mosaic diseases in Africa:A review[J]. Frontiers in Sustainable Food Systems ,2022,6:1076364.
[5] AKANO A,DIXON A,MBA C,et al. Genetic mapping of a dominant gene conferring resistance to cassava mosaic disease[J]. Theoretical and Applied Genetics,2002,105(4) :521–525.
[6] WOLFE M D,RABBI I Y,EGESI C,et al. Genome-wide association and prediction reveals genetic architecture of cassava mosaic disease resistance and prospects for rapid genetic improvement[J]. The Plant Genome,2016,9(2) . doi: 10.3835/plantgenome2015.11.0118
[7] RABBI I Y,KAYONDO S I,BAUCHET G,et al. Genome-wide association analysis reveals new insights into the genetic architecture of defensive,agro-morphological and quality-related traits in cassava[J]. Plant Molecular Biology,2022,109(3) :195–213.
[8] KIDWAI M,AHMAD I Z,CHAKRABARTY D. Class III peroxidase:An indispensable enzyme for biotic/abiotic stress tolerance and a potent candidate for crop improvement[J]. Plant Cell Reports,2020,39(11) :1381–1393.
[9] SU P S,YAN J,LI W,et al. A member of wheat class III peroxidase gene family,TaPRX-2A,enhanced the tolerance of salt stress[J]. BMC Plant Biology,2020,20(1) :392.
[10] LI S C. Novel insight into functions of ascorbate peroxidase in higher plants:More than a simple antioxidant enzyme[J]. Redox Biology,2023,64:102789.
[11] HENDEL B,SINSABAUGH R L,MARXSEN J. Lignin-degrading enzymes:phenoloxidase and peroxidase[M]// Methods to study litter decomposition:a practical guide,2020:425–431.
[12] FERNÁNDEZ-PÉREZ F,POMAR F,PEDREÑO M A,et al. Suppression of Arabidopsis peroxidase 72 alters cell wall and phenylpropanoid metabolism[J]. Plant Science,2015,239:192–199.
[13] XIONG T T,ZHANG S S,KANG Z Z,et al. Dose-dependent physiological and transcriptomic responses of lettuce (Lactuca sativa L.) to copper oxide nanoparticles-insights into the phytotoxicity mechanisms[J]. International Journal of Molecular Sciences,2021,22(7) :3688.
[14] PASSARDI F,PENEL C,DUNAND C. Performing the paradoxical:How plant peroxidases modify the cell wall[J]. Trends in Plant Science,2004,9(11) :534–540.
[15] LISZKAY A,KENK B,SCHOPFER P. Evidence for the involvement of cell wall peroxidase in the generation of hydroxyl radicals mediating extension growth[J]. Planta,2003,217(4) :658–667.
[16] BINDSCHEDLER L V,DEWDNEY J,BLEE K A,et al. Peroxidase-dependent apoplastic oxidative burst in Arabidopsis required for pathogen resistance[J]. The Plant Journal,2006,47(6) :851–863.
[17] ALMAGRO L,GÓMEZ ROS L V,BELCHI-NAVARRO S,et al. Class III peroxidases in plant defence reactions[J]. Journal of Experimental Botany,2009,60(2) :377–390.
[18] 王琪琦,潘冉冉,王亚杰,等. 斯里兰卡木薯花叶病毒在亚洲的暴发、传播与防控[J]. 生物安全学报,2025, 34(1) :20–26. WANG Q Q,PAN R R,WANG Y J,et al. Outbreak,transmission and control of cassava Mosaic virus from Sri Lanka in Asia[J]. Journal of BioSecurity,2025,34(1) :20–26.(in Chinese)
[19] REY C,VANDERSCHUREN H. Cassava mosaic and brown streak diseases:Current perspectives and beyond[J]. Annual Review of Virology,2017,4(1) :429–452.
[20] ARYA R,SABIR J S M,BORA R S,et al. Optimization of culture parameters and novel strategies to improve protein solubility[J]. Methods in Molecular Biology,2015,1258:45–63.
[21] CARDOSO V M,CAMPANI G,SANTOS M P,et al. Cost analysis based on bioreactor cultivation conditions:Production of a soluble recombinant protein using Escherichia coli BL21(DE3) [J]. Biotechnology Reports,2020,26:e00441.
[22] 程潇,宋锦佳,安仕芹,等. 木薯MeRsp基因克隆与原核表达[J]. 分子植物育种,2022,20(11) :3517–3523. CHENG X,SONG J J,AN S Q,et al. Cloning and prokaryotic expression of MeRsp gene in cassava[J]. Molecular Plant Breeding,2022,20(11) :3517–3523.(in Chinese)
[23] RAJPUT V D,HARISH,SINGH R K,et al. Recent developments in enzymatic antioxidant defence mechanism in plants with special reference to abiotic stress[J]. Biology,2021,10(4) :267.
[24] 吕春芳,李海霞,贾然然,等. 植物过氧化物酶活性测定实验方法的改良[J]. 轻工科技,2024,40(2) :15–18. LV C F,LI H X,JIA R R,et al. Improvement of experimental method for determination of plant peroxidase activity[J]. Light Industry Science and Technology,2024,40(2) :15–18.(in Chinese)
[25] 吴艳玲,谭竹钧,韩雅莉,等. 黄粉虫纤溶活性蛋白酶(TFP) 酶学性质以及红外光谱分析[J]. 时珍国医国药,2011,22(9) :2061–2064. WU Y L,TAN Z J,HAN Y L,et al. Infrared spectral analysis and enzymological characteristic of Tenebrio fibrinolyric proteins (TFP) [J]. Lishizhen Medicine and Materia Medica Research,2011,22(9) :2061–2064.(in Chinese)




 下载:
下载: