A Concise Phenotype-based Collection of Phalaenopsis Germplasms
-
摘要:目的
基于表型性状构建蝴蝶兰初级核心种质,为蝴蝶兰资源的保存利用和品种选育提供理论依据。
方法以210份蝴蝶兰种质资源为材料,对32个表型性状进行调查,其中包括13个质量性状和19个数量性状。采用3种取样方法(随机取样法、优先取样法、偏离度取样法)、2种遗传距离(欧氏距离、马氏距离)、3种取样比例(20%、25%、30%)和8种聚类方法(最短距离法、最长距离法、中间距离法、重心法、不加权类平均法、可变类平均法、可变法、离差平方和法),探讨最佳取样策略,构建核心种质。利用均值差异百分率(Mean difference percentage, MD)、方差差异百分率(Variance difference percentage, VD)、极差符合率(Range coincidence rate, CR)和变异系数变化率(Variation coefficient change rate, VR)对备选核心种质进行评价,利用均值、极值比较分析和主成分分析对核心种质进行确认。
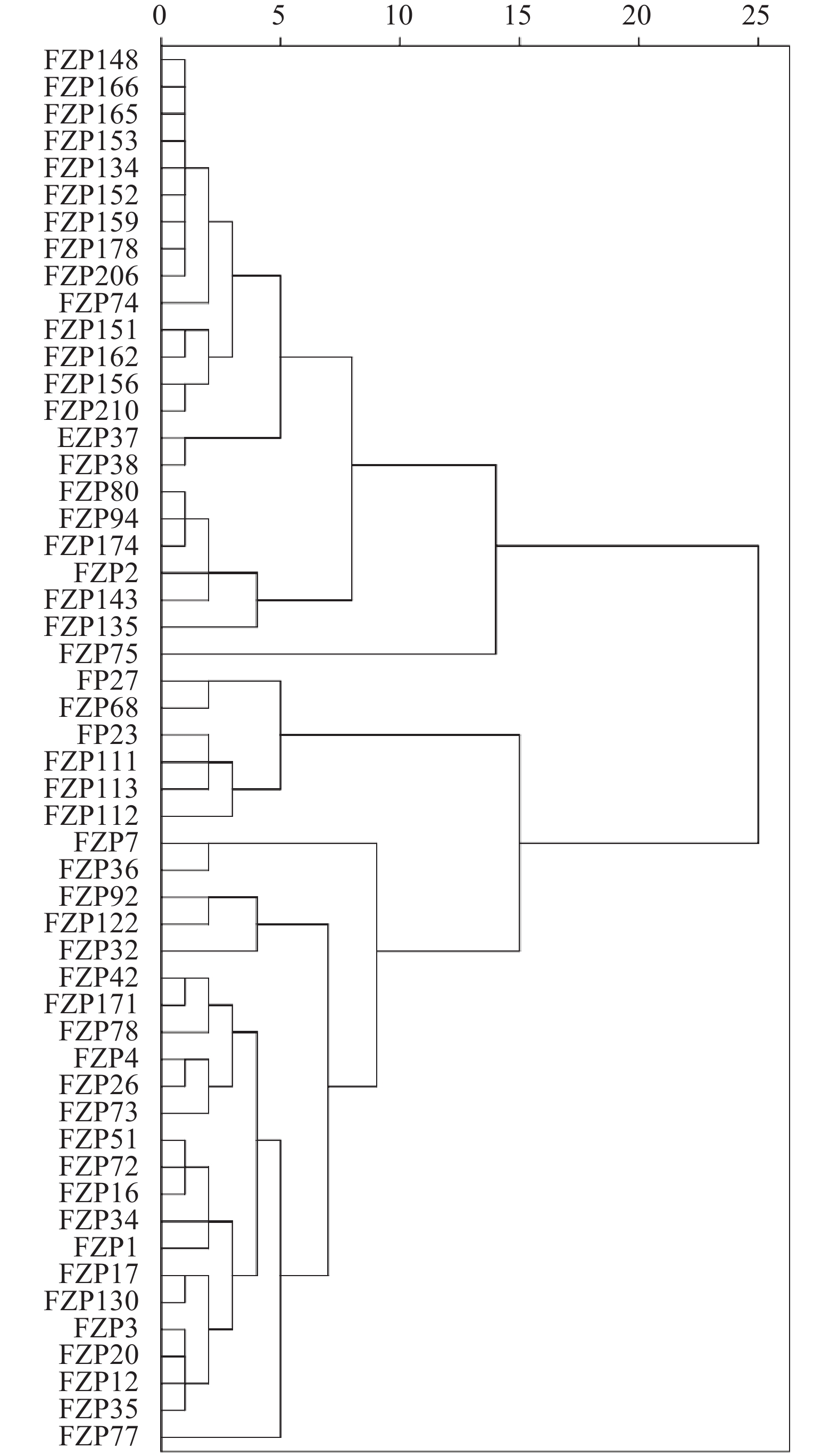
结果优先取样法能够使核心种质更具代表性,欧式距离优于马氏距离,25%为最佳取样比例,最优聚类方法为最长距离法,最终抽提出52份材料的初级核心种质。构建的核心种质各项评价参数优秀,能够代表原始种质的遗传多样性,主成分信息得到保留,并且很好地去除了原种质的遗传冗余。
结论构建的蝴蝶兰初级核心种质可以优先作为后续蝴蝶兰种质资源研究的材料,为种质的保存和利用提供理论基础。
Abstract:ObjectiveA concise collection of Phalaenopsis germplasms based on their phenotypic traits was preliminarily assembled to facilitate conservation and utilization of the natural resource.
MethodsTwo hundred and ten Phalaenopsis germplasms encompassing 32 phenotypes, including 13 qualitative and 19 quantitative traits, were included in this study. The random, preferred, and deviation sampling methods, the Euclidean and Mahalanobis genetic distances, the 20%, 25%, and 30% sampling ratios as well as the single, complete, median, centroid, unweighted, weighted, flexible, and ward clustering methods were employed in comparison to optimize the selection process. The information obtained was scrutinized with evaluations on the mean difference percentage (MD), variance difference percentage (VD), range coincidence rate (CR), and variation coefficient change rate (VR). It was further verified by the mean and extreme comparative analyses and principal component analysis to arrive at a concise and representative collection of the germplasms.
ResultsThe germplasms for the collection were eventually gathered by means of the preferred sampling at the Euclidean distance and 25% sampling ratio with the complete clustering. Out of the 210 germplasms, 52 selected cultivars were deemed to statistically represent the genetic diversity of the current collection with all the principal components on phenotypic traits preserved without redundancy.
ConclusionThe collection of Phalaenopsis germplasms assembled by this study was concise and representative of the resource presently in stock. It covered all key phenotypes needed to be preserved for future studies and applications.
-
Keywords:
- Phalaenopsis /
- germplasm collection /
- phenotypic traits /
- sampling methods
-
0. 引言
【研究意义】水禽圆环病毒(waterfowl circovirus, WFCV)主要包括鹅圆环病毒(goose circovirus, GoCV)和鸭圆环病毒(duck circovirus, DuCV)。根据国际病毒分类委员会(International Committee on Taxonomy of Viruses, ICTV)分类建议,WFCV为圆环病毒科圆环病毒属的成员,是一种小型无囊膜 DNA 病毒,基因组为共价闭合环状单链基因组,大小为
1821 ~1996 nt,含有6个主要开放阅读框(open reading frame, ORF)V1~V3和ORF C1~C3,其中ORF V1 编码病毒基因组复制相关的蛋白(Rep),ORF C1编码病毒抗原相关的衣壳蛋白(Cap)[1]。GoCV和DuCV分别于1999年和2003年首次在德国的鹅群[2]和鸭群[3]中被发现,随后这两种病毒感染在世界各地陆续出现了报道[4−13],且临床感染率呈上升趋势[14−17],近来还出现了WFCV交叉跨种感染病例[18−22]。深入开展水禽WFCV的感染情况调查,明确病毒基因组的分子特征和变异规律,将为今后开展该病分子致病机制研究及制定行之有效的疫病防控策略提供科学依据。【前人研究进展】GoCV主要引起鹅生长发育迟缓、羽毛絮乱、腹泻,法氏囊、胸腺和脾脏等组织淋巴细胞减少和组织细胞增生,以及法氏囊中单核细胞可见嗜碱性包涵体的存在[23]。我国大陆于2005年首次报道了鹅群中存在GoCV的感染[8],随后开始逐渐发展流行。尽管调查发现鹅感染的圆环病毒主要为GoCV[24],但赵光伟等[18]2009年首次报道了重庆一例患病鹅中检测到DuCV,Shehata等[19]在2013年波兰境内收集的一批发生鹅细小病毒感染的观赏鸭样品中检测到了GoCV的共感染,Xu等[20,21]在鸭群中发现鹅圆环病毒,随后又在鹅群中检测出到DuCV基因1型和2型,可见鹅群中的圆环病毒感染十分严峻,需及时对鹅群开展圆环病毒流行病学调查。【本研究切入点】鉴于当前鹅群较为复杂的WFCV病毒生态流行状况,本研究应用三引物检测方法对近年来收集的临床鹅样品开展WFCV感染调查,进而明确鹅群中GoCV和DuCV两种病毒的流行情况。【拟解决的关键问题】明确当前鹅群GoCV与DuCV流行特点,解析发现的自然共感染病例中的GoCV与DuCV基因组分子特征及变异情况,为全面了解鹅群中WFCV流行规律及科学防治提供依据。1. 材料与方法
1.1 试验材料
1.1.1 病料收集
2023—2024年,自福建省、广西省和安徽等6个省共收集到153份鹅肝脏、脾脏、胸腺、法氏囊等组织样品,这些组织来源于临床上生长发育不良/体况消瘦、羽毛脱落及死亡的鹅,初步怀疑存在水禽圆环病毒感染,样品收集后于-80 ℃保存备用,相关信息见表1。
表 1 收集的鹅组织样品信息Table 1. Information of clinical tissue samples collected from geese收集时间
Collection time组织样品来源与数量 Sample sources and number/份 合计 Total/份 福建 广西 安徽 广东 江西 浙江 2023年 28 21 25 10 8 7 99 2024年 19 13 5 8 5 4 54 合计 Total 47 34 30 18 13 11 153 1.1.2 主要试剂与仪器
Simply P Virus DNA/RNA Extraction Kit购自杭州博日科技股份有限公司,2 × Phanta Max Master Mix购自南京诺唯赞生物科技股份有限公司,DL2000 DNA Marker购自北京宝日医生物技术有限公司,高速冷冻离心机(武汉基因科技有限公司),梯度PCR仪(Bio-Rad公司),高速低温组织研磨仪(武汉赛维尔科技有限公司)。
1.2 试验方法
1.2.1 病料处理与核酸提取
取适量采集的脏器置于研磨管中,加入3倍体积的1×PBS与钢制研磨珠,经高速低温组织研磨仪匀浆后,反复冻融3次,于高速冷冻离心机4 ℃ 12 000 ×g 离心10 min;取200 μL 上清液使用Simply P Virus DNA/RNA Extraction Kit提取纯化核酸,按照试剂盒说明书进行,提取的病毒核酸储存于−80 ℃。
1.2.2 GoCV与DuCV检测
样品GoCV与DuCV的检测参照王梦鸽等[25]的方法,由生工生物工程(上海)股份有限公司合成可同时检测GoCV与DuCV引物对:CVF 5′-AAY CWC GCG GGA AGT GGT GGG A -3 ′;DuCV-R1:5′- TTC TAR GCA TAA ACG AGA TC -3′;GoCV-R1:5′- ATA MGA TTC GGA CAA TGG ACT G -3′。以提取的核酸为模板,进行GoCV与DuCV的PCR检测。反应体系为20 μL:2 × Phanta Max Master Mix 10 μL,CVF(10 μmol·L-1) 0.6 μL,DuCVR (10 μmol·L-1)0.45 μL,GoCVR (10 μmol·L-1)0.3 μL,模板1 μL,RNase Free H2O补足至20 μL。反应条件:95 ℃预变性5 min;95 ℃ 变性30 s,54.6 ℃退火30 s,72 ℃ 延伸30 s,36个循环;72 °C彻底延伸10 min。反应结束后经1%琼脂糖凝胶电泳检测。阳性样品送至生工生物工程(上海)股份有限公司进行测序。
1.2.3 全基因组扩增与序列测序
根据GenBank上公布的GoCV和DuCV全基因序列,应用primer 3 plus 在线引物设计程序,在王梦鸽等[25]检测引物的基础上,设计反向扩增引物,扩增余下的基因片段。引物对序列如下:GoCV-F1:5′-CGT TGT CTT GAA TGG AGT TTA GCC A-3′,GoCV-R2:5′- ATA GCC RTC CCA CCA CTT CCC -3′;DuCV-F1:5′-GGA TTA AAC ACT TGG GCA GC-3′,DuCV-R2:5’- GTA ACG GGT AAC GGT CGG TA -3’,其中GoCV-F1与GoCV-R2用于扩增GoCV基因片段,DuCV-F1与DuCV-R2用于扩增DuCV基因片段。反应体系为20 μL:2 × Phanta Max Master Mix 10 μL,上下游引物(10 μmol·L-1)各0.25 μL,模板1 μL,ddH2O补足至20 μL。反应条件:95 ℃预变性5 min;95 ℃ 变性30 s,55~58 ℃退火30 s,72 ℃ 延伸2 min,35个循环;72 °C彻底延伸10 min。反应结束后经1%琼脂糖凝胶电泳检测扩增结果。将阳性样品送至生工生物工程(上海)股份有限公司进行测序。
1.3 数据分析
将测序结果应用DNAStar软件SeqMan程序进行拼接整理,获得的全基因组序列与GenBank数据库中的GoCV和DuCV基因组序列进行比对分析。应用Lasergene 7.0软件包中的MegAlign程序分别进行GoCV和DuCV全基因组同源性分析,并应用TBtools-Ⅱ 软件Heatmap程序绘制同源性热图[26]。应用 MEGA 11 软件 phylogeny 程序进行建树。采用Neighbor Joining 方法,bootstrap method重复
1000 次,采用默认模型进行全基因组遗传进化分析。将获得共感染毒株的Rep及Cap蛋白氨基酸序列与NCBI数据库中的相应蛋白进行比较。应用MegAlign程序分别进行GoCV和DuCV主要功能蛋白同源性分析。应用在线程序BepiPred 3.0 Server 预测分析共感染毒株的主要功能Cap蛋白 B 细胞抗原表位。
为研究是否存在重组的事件,将采集的自然共感染病例样本与 GoCV 和 DuCV 参考序列进行比较。使用重组检测程序 RDP5 的默认设置对序列进行分析,并应用Simplot 3.5.1进行验证。
2. 结果与分析
2.1 GoCV与DuCV检测
对采集的鹅组织样品进行GoCV与DuCV的PCR检测,检出大小为407 bp和554 bp的两条目的条带,其中407 bp的扩增片段为GoCV,554 bp大小的扩增片段为DuCV,片段大小与预期相符。对153份样品的检测结果发现53份WFCV核酸阳性样品,阳性率达到34.6%,包括GoCV阳性样品47份,阳性率为30. 7%(47/153),DuCV阳性样品5份,阳性率为3.3%(5/153),而一份来自安徽的鹅组织中同时检出GoCV与DuCV,部分结果如图1所示。
2.2 共感染病例病毒序列分析
2.2.1 全基因组分子特征及同源性分析
全基因组测序结果显示,共感染病例中的GoCV Go-AH2367株基因组全长1821 nt,ORF V1长882 nt,编码293个氨基酸的Rep蛋白;ORF C1长753 nt,编码250个氨基酸的Cap蛋白。共感染病例中的DuCV Du-AH2367株基因组全长1992 nt,ORF V1长879 nt,编码292个氨基酸Rep蛋白;ORF C1长774 nt,编码257个氨基酸的Cap蛋白。
获得的Go-AH2367株与Du-AH2367株的全基因组序列之间的同源性仅为69%。两株WFCV基因序列与已发布的GoCV和DuCV毒株序列同源性分析表明(图2),GoCV Go-AH2367株与GoCV(KR869727.1~AF536933.1)的全基因组序列同源性在82.9%~99.0%,与DuCV-1(MN078102.1~PP056136.1)的同源性在78.5%~79.5%,与DuCV-2(MN928806.1~ON227557.1)的同源性在79.2%~81.0%,与DuCV-3病毒株(OP432310.1)的同源性仅为70.9%。DuCV病毒株Du-AH2367株全基因序列与DuCV-1(EU344803.1~OR134489.1)的同源性最高,在93.2%~99.7%,与DuCV-2(MK814585.1~MN928807.1)的序列同源性在83.2%~92.9%,而与DuCV-3株的序列同源性较低,仅为73.7%(OP432310.1),与GoCV(KR869727.1~AF536933.1)的同源性仅在66.1%~68.8%。
2.2.2 全基因组遗传进化分析
将本研究获得的GoCV Go-AH2367与DuCV Du-AH2367株与GenBank数据库中的30株GoCV、45株DuCV全基因组序列进行遗传进化分析(图3)。结果显示,Go-AH2367株处于GoCV病毒株进化分支中,与2015年山东的鹅源GoCV株 Shandong(KT387277.1)遗传距离最近,与2012年波兰的鹅病毒株2GK(KR869727.1)及2014年波兰的鹅病毒株DG5(KT808653.1)遗传距离较远。DuCV Du-AH2367株处于DuCV大进化分支中,与DuCV-1病毒株处于同一小进化分支,与广西2022年的鸭源DuCV株DuCV-GX51-2022(OR134489.1)和2020年鸭源DuCV株DuCV-GX28-2020(OR134475.1)亲缘关系最近。
2.2.3 共感染病毒基因组重组分析
使用RDP5分别对GoCV Go-AH2367和DuCV Du-AH2367以及GenBank数据库中76株WFCV基因组序列(DuCV 46株和GoCV 30株)参考序进行重组分析。结果显示GoCV Go-AH2367和DuCV Du-AH2367与参考序列之间均未发生重组事件。
2.2.4 GoCV Go-AH2367毒株主要功能蛋白变异分析
GoCV Go-AH2367毒株ORF V1基因编码氨基酸序列同源性分析结果表明,GoCV Go-AH2367 ORF V1与GenBank数据库中GoCV ORF V1 同源性为92.15%~100%,与DuCV ORF V1 的同源性在81.2%~84.2%,与2015年山东的鹅源GoCV株 Shandong(ALZ45069.1)氨基酸序列同源性达到100%。ORF C1基因编码氨基酸序列同源性分析结果显示,Go-AH2367株ORF C1与GenBank数据库中的GoCV ORF C1 同源性为79.9%~100%,与GenBank数据库中的DuCV-1 ORF C1 序列同源性为45.0%~48.8%,与2015年山东鹅源GoCV株 Shandong(ALZ45071.1)及2005年浙江鹅源GoCV病毒株yk3(ABA39142.1)ORF C1同源性高达100%。
GoCV Go-AH2367毒株ORF V1长882 nt,编码一个293个氨基酸Rep蛋白,该蛋白上与滚环复制相关的3处功能基序I(FTINNP)、II(TPHLQGF)、III(YCSKE),及病毒dNTP结合域超家族解旋酶基序Walker A(GRPGSGKSR)和B(DDFYGW)均高度保守;ORF C1长753 nt,编码一个250个氨基酸的Cap蛋白,与以发布的基因I型GoCV(GoCV-I)Cap蛋白 8个B细胞抗原表位(18–22、57–65、72–84、94–104、127–168、179–190、195–199、220–231 nt)高度保守,与GoCV-Ⅱ和GoCV-Ⅲ在这些表位分别存在3个和5个位点差异(图4)。
2.2.5 DuCV Du-AH2367毒株主要功能蛋白变异分析
DuCV Du-AH2367毒株ORF V1基因编码氨基酸序列同源性分析结果表明,DuCV Du-AH2367 ORF V1与GenBank数据库中的GoCV ORF V1 序列同源性为82.2%~85.1%,与DuCV-1 ORF V1 的同源性为96.2%~100.0%,与DuCV-2 ORF V1 的同源性为92.8%~99.7%,与DuCV-3 ORF V1 的同源性为89%,与2018年山东鸭源DuCV病毒株SDHZ1223(QIP76518.1)等多株DuCV-1的ORF V1核苷酸序列同源性达到100%。Du-AH2367株ORF C1与GenBank数据库中的GoCV ORF C1的同源性为46.9%~51.4%,与GenBank数据库中的DuCV-1 ORF C1的同源性为92.6%~99.6%,与DuCV-2 ORF C1的同源性为86%~94.6%,与DuCV-3 ORF C1的同源性为50.8%,差异较大,与2018年山东鸭源DuCV病毒株SDHZ1223(QIP76519.1)等多株DuCV-1的ORF C1序列同源性达到99.6%。
DuCV Du-AH2367毒株ORF V1长879 nt,编码292个氨基酸Rep蛋白,其中包含的滚环复制相关及dNTP结合域超家族解旋酶基序等多个功能基序与已报道的基因1型DuCV毒株高度一致;ORF C1长774 nt,编码一个257个氨基酸的Cap蛋白,与DuCV-1 Cap蛋白 6个B细胞抗原表位(5–32、56–88、101–113、130–139、147–195、229–235)高度保守,与DuCV-2和DuCV-3在这些表位分别存在9个和92个位点差异(图5)。
3. 讨论
水禽圆环病毒可引起禽得生长迟缓、腹泻和羽毛脱落等,损伤免疫器官,并引起淋巴细胞严重减少的免疫抑制性疾病[27−29]。已有的病例报道表明两种WFCV具有各自的易感动物,具有明显的种属特异性,GoCV仅感染不同品种和日龄的鹅,DuCV仅感染不同品种和日龄的鸭[30]。赵光伟等[18]于2009年首次报道了自重庆一例患病鹅中检测到DuCV,2024年本团队在评估建立的鸭圆环病毒新型检测技术特异性时,自早期收集的27份患病鹅样品中检测到3份DuCV-2阳性样品[22],Xu等[20]也报道自2021年和2022年收集的鹅组织样品中分别检测到DuCV-1和DuCV-2。与鹅群中出现了DuCV跨种感染类似,鸭群中同样检测到GoCV的跨种感染。2013年Shehata等[19]从观赏鸭中检测到1例GoCV阳性样品,2024年Xu等[21]开展鸭圆环病毒分子流行病学调查时发现,从河南和安徽的两家养鸭场采集的10份样品中均检测到GoCV,WFCV感染呈现出了极为复杂的流行态势。
本研究对收集的153份鹅样品进行GoCV和DuCV检测发现,鹅群水禽圆环阳性率高达34.6%(53/153),其中GoCV单纯感染的鹅阳性样品占88.7%(47/53),DuCV单纯感染的鹅阳性样品占9.4%(5/53),另外还发现1例GoCV和DuCV共感染阳性样品。可见,鹅群中WFCV感染呈现病毒生态多样性,以GoCV感染为主,同时还存在DuCV的跨种感染及GoCV和DuCV的共感染的情况。经病例回访跟踪调查了解到,该样品来源于安徽一家鸭、鹅混养的农户送检的鹅病例,实验室的病原检测发现该病例还存在小鹅瘟病毒感染。可见该病例存在较为严重的多病原共感染现象,这可能与水禽的饲养方式有关。尽管网床平养、笼养等设施化养殖模式已在我国水禽(尤其是肉鸭)的养殖中广泛应用,但东南沿海地区的水禽养殖户或农户以地面旱养或地面加水域圈养方式较多,同时存在鸡、鸭、鹅混养的现象,这些因素为GoCV和DuCV在水禽中的广泛流行及交叉跨种感染提供了十分便利的条件。
本研究在鹅中发现的共感染病例是迄今发现的全球首例GoCV和DuCV共感染病例。对共感染鹅病例样品中的GoCV与DuCV的全基因组分子特征、遗传进化、ORF V1和ORF C1编码的蛋白氨基酸变异分析表明,二者的基因组均未出现基因重组、插入或缺失等显著变异,两个主要的功能蛋白的重要功能基序高度保守。但临床上GoCV与DuCV交叉跨种感染及共感染病例的出现无疑为WFCV的防控造成更严峻的挑战,尤其是共感染的出现是否导致更复杂、更严重的临床发病还有待进一步研究。GoCV和DuCV相互间的交叉跨种感染已是一个不容忽视的问题,对WFCV病的诊断与防控造成了严峻考验。深入了解水禽中圆环病毒感染情况,对保障水禽健康养殖、制定科学防控策略具有重要意义。
4. 结论
本研究应用PCR技术对鹅群中水禽圆环病毒开展流行检测发现,当前鹅群中的GoCV和DuCV水禽圆环病毒流行较严重,呈现病原生态多样性,除检测到DuCV的跨种感染外,还发现首例GoCV和DuCV共感染病例。基因组序列分析表明该自然共感染病例中的GoCV和DuCV遗传稳定,未发生明显的遗传变异现象。研究结果将为今后深入开展WFCV跨种感染的分子机制研究及鹅WFCV病的防治策略制定提供科学依据。
-
表 1 蝴蝶兰性状及代码
Table 1 Codes of Phalaenopsis phenotypic traits
代码 Code 性状Traits 代码 Code 性状Traits C1 植株大小 Plant length/cm C17 花序梗数量 Peduncle number/个 C2 叶片长度 Leaf length/cm C18 合蕊柱长度 Gynostemium length/cm C3 叶片宽度 Leaf width/cm C19 合蕊柱粗度 Gynostemium thickness/cm C4 花序长度 Inflorescence length/cm C20 叶片形状 Leaf shape C5 花数量 Flower number/朵 C21 叶片上表面颜色 Color of the upper side of leaf C6 花序梗长度 Peduncle length/cm C22 叶片花青甙显色 Anthocyanin coloration of leaf C7 花序梗粗度 Peduncle thickness/cm C23 叶片厚度 Leaf thickness C8 花梗长度 Pedicel length/cm C24 叶片表面纹理 Leaf surface texture C9 花长度 Flower length/cm C25 花序类型 Inflorescence type C10 花宽度 Flower width/cm C26 花序梗表面颜色 Surface color of peduncle C11 萼片长度 Dorsal sepal length/cm C27 花瓣排列方式 Petal arrangement C12 萼片宽度 Dorsal sepal width/cm C28 花香味 Scent of flower C13 花瓣长度 Petal length/cm C29 中萼片颜色数量 Number of middle sepal colors C14 花瓣宽度 Petal width/cm C30 侧萼片颜色数量 Number of lateral sepal colors C15 中裂片长度 Apical lobe length/cm C31 花瓣颜色数量 Number of petal colors C16 中裂片宽度 Apical lobe width/cm C32 花蜡质 Waxiness of flower 表 2 蝴蝶兰质量性状分级与赋值
Table 2 Classification and codes of qualitative Phalaenopsis traits
性状 Traits 赋值 Assigning value 叶片形状
Leaf shape披针形=1,窄卵圆形=2,椭圆形=3,窄倒卵圆形=4,倒卵圆形=5
lanceolate=1,narrowly oval=2,elliptical=3,narrowly inverted ovoid=4,obovoid=5叶片上表面颜色
Color of the upper side of leaf浅绿色=1,中等绿色=2,深绿色=3,灰绿色=4
light green=1,medium green=2,dark green=3,greyish green=4叶片花青甙显色
Anthocyanin coloration of leaf无=1,有=2
no=1,yes=2叶片厚度
Leaf thickness薄=1,中=2,厚=3
thin=1,medium=2,thick=3叶片表面纹理
Leaf surface texture无=1,有=2
no=1,yes=2花序类型
Inflorescence type单生花=1,总状花序=2,圆锥花序=3
solitary=1,raceme=2,panicle=3花序梗表面颜色
Surface color of peduncle浅绿色=1,中等绿色=2,深绿色=3,灰绿色=4,红褐色=5,褐色=6
light green=1,medium green=2,dark green=3,greyish green=4,reddish brown=5,brown=6花瓣排列方式
Petal arrangement分开=1,相接=2,重叠=3
separate=1,adjacent=2,overlapping=3花香味
Scent of flower无=1,有=2
no=1,yes=2中萼片颜色数量
Number of middle sepal colors1种=1,2种=2,3种=3,3种以上=4
1 color=1,2 colors=2,3 colors=3,more than 3 colors=4侧萼片颜色数量
Number of lateral sepal colors1种=1,2种=2,3种=3,3种以上=4
1 color=1,2 colors=2,3 colors=3,more than 3 colors=4花瓣颜色数量
Number of petal colors1种=1,2种=2,3种=3,3种以上=4
1 color=1,2 colors=2,3 colors=3,more than 3 colors=4花蜡质
Waxiness of flower无=1,有=2
no=1,yes=2表 3 144个备选核心种质的评价参数
Table 3 Parameters for evaluating 144 candidate germplasms
取样比例
Sample ratio遗传距离
Genetic distance聚类方法
Cluster method多次聚类随机取样法
Multiple cluster random
sampling method多次聚类优先取样法
Multiple cluster preferred sampling method多次聚类偏离度取样法
Multiple cluster deviation
sampling methodMD VD CR VR MD VD CR VR MD VD CR VR 20% 欧氏距离
Euclidean distanceSingle 3.13 18.75 91.34 111.47 3.13 40.63 100.00 119.45 9.38 28.13 97.96 119.58 Complete 0.00 15.63 89.52 107.99 3.13 40.63 100.00 119.45 0.00 37.50 98.02 120.42 Median 0.00 9.38 90.43 108.20 3.13 40.63 100.00 119.45 3.13 15.63 95.28 114.72 Centroid 3.13 15.63 91.48 112.49 3.13 40.63 100.00 119.45 0.00 15.63 95.69 113.18 Unweighted 0.00 6.25 93.92 109.68 3.13 40.63 100.00 119.45 0.00 15.63 94.49 119.55 weighted 0.00 15.63 89.88 106.84 3.13 40.63 100.00 119.45 0.00 31.25 95.39 116.32 Flexible 3.13 12.50 86.44 105.95 3.13 40.63 100.00 119.45 0.00 28.13 94.05 116.58 Ward 0.00 15.63 90.43 110.29 3.13 40.63 100.00 119.45 0.00 43.75 95.43 117.14 马氏距离
Mahalanobis distanceSingle 6.25 15.63 90.77 110.74 3.13 40.63 100.00 119.45 21.88 53.13 96.81 116.87 Complete 0.00 6.25 90.36 107.11 3.13 40.63 100.00 119.45 3.13 46.88 96.18 121.91 Median 3.13 15.63 90.28 109.75 3.13 40.63 100.00 119.45 9.38 53.13 97.08 120.88 Centroid 21.88 25.00 93.94 115.19 3.13 40.63 100.00 119.45 40.63 50.00 97.34 114.20 Unweighted 0.00 9.38 89.96 104.78 3.13 40.63 100.00 119.45 3.13 53.13 95.31 122.61 weighted 0.00 9.38 90.15 105.21 3.13 40.63 100.00 119.45 0.00 53.13 94.04 117.15 Flexible 0.00 9.38 84.82 99.43 3.13 40.63 100.00 119.45 0.00 53.13 92.76 118.25 Ward 0.00 15.63 88.01 110.22 3.13 40.63 100.00 119.45 3.13 50.00 96.31 124.36 25% 欧氏距离
Euclidean distanceSingle 3.13 18.75 94.06 108.15 3.13 46.88 100.00 116.83 9.38 21.88 97.99 116.42 Complete 0.00 9.38 90.33 107.97 0.00 37.50 100.00 118.20 0.00 15.63 98.02 117.14 Median 0.00 6.25 91.10 104.70 18.75 37.50 100.00 113.46 0.00 12.50 96.03 111.96 Centroid 3.13 12.50 92.52 108.82 0.00 28.13 100.00 116.11 0.00 12.50 96.19 110.06 Unweighted 0.00 9.38 96.02 106.98 0.00 37.50 100.00 117.18 0.00 15.63 96.02 116.94 weighted 0.00 9.38 91.54 104.42 0.00 34.38 100.00 116.48 0.00 12.50 95.83 112.31 Flexible 3.13 12.50 89.51 104.44 0.00 34.38 100.00 116.41 0.00 15.63 95.61 113.33 Ward 0.00 12.50 92.22 107.95 0.00 34.38 100.00 117.65 0.00 21.88 95.49 113.83 马氏距离
Mahalanobis distanceSingle 3.13 15.63 93.96 108.25 3.13 43.75 100.00 116.84 25.00 43.75 97.80 114.64 Complete 0.00 6.25 92.49 104.49 18.75 21.88 100.00 113.25 3.13 43.75 96.18 117.74 Median 12.50 15.63 90.96 106.71 6.25 37.50 100.00 114.98 12.50 50.00 97.65 116.36 Centroid 25.00 40.63 94.40 111.51 6.25 34.38 100.00 114.99 12.50 50.00 97.80 115.74 Unweighted 0.00 9.38 91.23 105.90 3.13 21.88 100.00 115.20 0.00 43.75 95.72 119.67 weighted 0.00 6.25 90.32 102.42 0.00 31.25 100.00 115.65 0.00 34.38 94.04 114.45 Flexible 0.00 6.25 89.18 102.02 9.38 43.75 100.00 115.10 0.00 43.75 94.37 115.04 Ward 0.00 12.50 88.99 105.98 0.00 28.13 100.00 113.91 0.00 46.88 97.06 118.15 30% 欧氏距离
Euclidean distanceSingle 3.13 18.75 94.06 105.70 6.25 28.13 100.00 113.08 9.38 18.75 98.45 115.29 Complete 0.00 12.50 91.83 106.48 0.00 28.13 100.00 115.17 0.00 15.63 98.51 115.55 Median 0.00 6.25 92.13 104.42 3.13 21.88 100.00 110.75 0.00 12.50 97.82 109.50 Centroid 0.00 9.38 93.09 106.86 0.00 18.75 100.00 112.80 0.00 12.50 96.23 108.23 Unweighted 0.00 9.38 96.82 105.33 0.00 21.88 100.00 114.35 0.00 9.38 98.03 113.32 weighted 0.00 15.63 92.98 102.65 0.00 25.00 100.00 114.54 0.00 12.50 95.94 110.04 Flexible 0.00 15.63 90.92 104.92 0.00 21.88 100.00 113.34 0.00 12.50 95.69 111.23 Ward 0.00 12.50 93.17 107.20 0.00 25.00 100.00 114.83 0.00 18.75 96.00 111.83 30% 马氏距离
Mahalanobis distance最短距离法Single 9.38 15.63 94.73 107.77 0.00 31.25 100.00 113.80 15.63 40.63 97.80 113.30 最长距离法Complete 0.00 9.38 93.56 105.10 6.25 21.88 100.00 113.05 0.00 43.75 97.32 115.64 中间距离法Median 0.00 9.38 93.50 106.51 3.13 21.88 100.00 112.91 6.25 40.63 97.65 113.08 重心法Centroid 31.25 43.75 95.09 110.91 21.88 37.50 100.00 113.00 12.50 40.63 97.80 111.99 类平均法Unweighted 0.00 9.38 92.31 105.21 0.00 25.00 100.00 111.08 3.13 46.88 96.20 116.42 可变类平均法weighted 0.00 9.38 94.73 105.33 0.00 18.75 100.00 112.31 0.00 18.75 94.52 112.03 可变法Flexible 0.00 6.25 90.23 103.40 3.13 25.00 100.00 112.42 0.00 40.63 94.84 113.80 离差平方和法Ward 0.00 15.63 92.36 107.50 0.00 21.88 100.00 111.19 0.00 37.50 97.06 116.19 MD:均值差异百分率,VD:方差差异百分率, CR:极差符合率,VR:变异系数变化率;最短距离法,最长距离法,中间距离法,重心法,类平均法,可变类平均法,可变法,离差平方和法。
MD:mean difference percentage; VD:variance difference percentage; CR: range coincidence rate; VR:variation coefficient change rate;single,complete,median,centroid,unweighted,weighted,flexible,and ward clustering methods.表 4 原始种质与核心种质遗传差异比较
Table 4 Genetic differences between entire and representative collection of germplasms
性状
Traits类别
Categories均值
MeanP值
P value显著性
Significance方差
VarianceP值
P value显著性
Significance极差
Range变异系数
CV/%C1 原始种质 Original 29.98 0.98 NS 47.67 0.02 S* 42.5 23.03 核心种质 Core 29.94 73.40 42.5 28.62 C2 原始种质 Original 16.21 0.89 NS 12.59 0.01 S* 21.67 21.89 核心种质 Core 16.31 19.83 21.67 27.31 C3 原始种质 Original 6.70 0.63 NS 2.41 0.00 S** 11.17 23.17 核心种质 Core 6.54 5.00 11.17 34.20 C4 原始种质 Original 14.71 0.53 NS 94.34 0.07 NS 40.06 66.01 核心种质 Core 15.71 127.87 40.06 71.99 C5 原始种质 Original 11.31 0.90 NS 55.47 0.40 NS 33 65.87 核心种质 Core 11.16 58.05 33 68.27 C6 原始种质 Original 19.13 0.25 NS 126.63 0.10 NS 49.83 58.82 核心种质 Core 21.23 166.13 49.83 60.71 C7 原始种质 Original 0.47 0.34 NS 0.05 0.00 S** 2.93 46.54 核心种质 Core 0.52 0.15 2.93 75.11 C8 原始种质 Original 32.85 0.46 NS 383.95 0.06 NS 86.63 59.65 核心种质 Core 35.19 528.77 86.63 65.34 C9 原始种质 Original 5.43 0.14 NS 2.58 0.01 S** 8.2 29.58 核心种质 Core 5.90 4.30 8.2 35.13 C10 原始种质 Original 5.83 0.21 NS 3.58 0.01 S* 10.57 32.46 核心种质 Core 6.28 5.71 10.57 38.03 C11 原始种质 Original 2.98 0.14 NS 0.84 0.03 S* 4.73 30.75 核心种质 Core 3.20 1.22 4.73 34.61 C12 原始种质 Original 2.11 0.14 NS 0.67 0.01 S* 4.07 38.66 核心种质 Core 2.34 1.07 4.07 44.26 C13 原始种质 Original 2.70 0.18 NS 0.77 0.03 S* 4.83 32.46 核心种质 Core 2.89 1.14 4.83 36.94 C14 原始种质 Original 2.85 0.17 NS 2.20 0.03 S* 6.6 52.05 核心种质 Core 3.18 3.27 6.6 56.85 C15 原始种质 Original 1.73 0.41 NS 0.28 0.00 S** 3.27 30.66 核心种质 Core 1.64 0.56 3.27 45.41 C16 原始种质 Original 1.45 0.60 NS 0.39 0.01 S* 3.17 43.04 核心种质 Core 1.38 0.61 3.17 56.65 C17 原始种质 Original 1.71 0.33 NS 0.74 0.47 NS 4.66 50.24 核心种质 Core 1.58 0.74 4.66 54.58 C18 原始种质 Original 1.01 0.21 NS 0.02 0.00 S** 1.17 15.03 核心种质 Core 1.05 0.04 1.17 20.15 C19 原始种质 Original 0.45 0.35 NS 0.06 0.00 S** 3.54 56.28 核心种质 Core 0.51 0.22 3.54 91.93 C20 原始种质 Original 2.72 0.47 NS 1.19 0.11 NS 4 40.10 核心种质 Core 2.60 1.54 4 47.79 C21 原始种质 Original 1.85 0.75 NS 0.61 0.33 NS 3 42.33 核心种质 Core 1.81 0.67 3 45.22 C22 原始种质 Original 1.19 0.13 NS 0.16 0.08 NS 1 33.10 核心种质 Core 1.29 0.21 1 35.51 C23 原始种质 Original 1.90 0.58 NS 0.42 0.30 NS 2 34.17 核心种质 Core 1.96 0.47 2 34.92 C24 原始种质 Original 1.14 0.85 NS 0.12 0.36 NS 1 30.74 核心种质 Core 1.15 0.13 1 31.58 C25 原始种质 Original 2.41 0.19 NS 0.25 0.46 NS 2 20.86 核心种质 Core 2.31 0.26 2 21.94 C26 原始种质 Original 2.86 0.22 NS 2.24 0.13 NS 5 52.38 核心种质 Core 3.15 2.84 5 53.42 C27 原始种质 Original 1.07 0.27 NS 0.12 0.00 S** 2 32.53 核心种质 Core 1.15 0.29 2 46.64 C28 原始种质 Original 1.32 0.71 NS 0.22 0.45 NS 1 35.41 核心种质 Core 1.29 0.21 1 35.51 C29 原始种质 Original 2.00 0.88 NS 0.36 0.14 NS 3 29.95 核心种质 Core 2.02 0.45 3 33.24 C30 原始种质 Original 2.22 0.69 NS 0.49 0.40 NS 3 31.54 核心种质 Core 2.27 0.51 3 31.60 C31 原始种质 Original 2.04 0.47 NS 0.38 0.10 NS 3 30.09 核心种质 Core 2.12 0.50 3 33.30 C32 原始种质 Original 1.75 0.18 NS 0.19 0.17 NS 1 24.97 核心种质 Core 1.65 0.23 1 29.05 NS表示无显著差异;S*表示显著水平;S**表示极显著水平;P值表示判定假设检验结果的参数。
NS: No significant difference; S*: significant difference; S**: extremely significant difference; P: parameter for evaluating hypothesis test result.表 5 原始种质与核心种质主成分分析比较
Table 5 Principal components on phenotypic traits of entire and representative collection of germplasms
主成分
Component原始种质 Original collection 核心种质 Core collection 特征根
Characteristic-root贡献率/%
Contribution rate累积贡献率/%
Cumulative contribution rate特征根
Characteristic-root贡献率/%
Contribution rate累积贡献率/%
Cumulative contribution rate1 10.352 32.351 32.351 10.212 31.913 31.913 2 3.500 10.936 43.287 4.273 13.354 45.267 3 2.421 7.565 50.853 2.779 8.684 53.951 4 1.901 5.941 56.793 2.050 6.408 60.358 5 1.664 5.200 61.993 1.674 5.231 65.590 6 1.515 4.734 66.726 1.522 4.758 70.348 7 1.203 3.758 70.485 1.311 4.096 74.443 8 1.086 3.394 73.879 1.229 3.840 78.283 9 1.038 3.243 77.122 1.083 3.383 81.666 -
[1] 朱根发. 蝴蝶兰种质资源及杂交育种进展[J]. 广东农业科学,2015,42(5) :31–38. ZHU G F. Progress in germplasm resources and crossbreeding of Phalaenopsis[J]. Guangdong Agricultural Sciences,2015,42(5) :31–38. (in Chinese)
[2] 许申平,张腾飞,廖飞雄,等. 蝴蝶兰种质资源与育种研究[J]. 中国园艺文摘,2010,26(5) :27–30. XU S P,ZHANG T F,LIAO F X,et al. A review on studies of Phalaenopsis germplasm resources and breeding[J]. Chinese Horticulture Abstracts,2010,26(5) :27–30. (in Chinese)
[3] FRANKEL O. Genetic perspectives of germplasm conservation[M]. Cambridge:Cambridge University Press,1984:161–170.
[4] BROWN A H D. Core collections:A practical approach to genetic resources management[J]. Genome,1989,31(2) :818−824. DOI: 10.1139/g89-144
[5] TANAKA N,SHENTON M,KAWAHARA Y,et al. Investigation of the genetic diversity of a rice core collection of Japanese landraces using whole-genome sequencing[J]. Plant &; Cell Physiology,2021,61(12) :2087–2096.
[6] 朱业宝,王金英,江川. 水稻种质资源核心种质的研究进展[J]. 江西农业学报,2023,35(4) :27–32. ZHU Y B,WANG J Y,JIANG C. Research progress in core collection of rice germplasm resources[J]. Acta Agriculturae Jiangxi,2023,35(4) :27–32. (in Chinese)
[7] 李永祥,李会勇,扈光辉,等. 玉米应用核心种质的构建与应用[J]. 植物遗传资源学报,2023,24(4) :911–916. LI Y X,LI H Y,HU G H,et al. Construction and utilization of applied core collection in maize[J]. Journal of Plant Genetic Resources,2023,24(4) :911–916. (in Chinese)
[8] 赵欣蕊,陈啸天,薛薇,等. 基于表型性状分析构建冀北地区马铃薯核心种质[J]. 核农学报,2024,38(5) :805–818. ZHAO X R,CHEN X T,XUE W,et al. Construction of core potato germplasm resources in north Hebei based on phenotypic traits[J]. Journal of Nuclear Agricultural Sciences,2024,38(5) :805–818. (in Chinese)
[9] 郑福顺,王晓敏,李国花,等. 基于表型性状的宁夏番茄种质资源核心种质构建[J]. 浙江大学学报(农业与生命科学版) ,2021,47(2) :171–181. ZHENG F S,WANG X M,LI G H,et al. Core collection construction of Ningxia tomato germplasm resources based on phenotypic traits[J]. Journal of Zhejiang University (Agriculture and Life Sciences) ,2021,47(2) :171–181. (in Chinese)
[10] 李嘉伟,苏江硕,张飞,等. 基于表型性状构建传统菊花核心种质[J]. 中国农业科学,2021,54(16) :3514–3526. LI J W,SU J S,ZHANG F,et al. Construction of core collection of traditional Chrysanthemum morifolium based on phenotypic traits[J]. Scientia Agricultura Sinica,2021,54(16) :3514–3526. (in Chinese)
[11] 赵立民,李嘉伟,张飞,等. 基于表型数据构建切花小菊核心种质[J]. 园艺学报,2022,49(10) :2273–2284. ZHAO L M,LI J W,ZHANG F,et al. Construction of a core collection of spray cut Chrysanthemum based on phenotypic data[J]. Acta Horticulturae Sinica,2022,49(10) :2273–2284. (in Chinese)
[12] 秦子璐,徐正康,戴晓港,等. 望春玉兰种质资源遗传多样性分析与核心种质构建[J]. 园艺学报,2024,51(8) :1823–1832. QIN Z L,XU Z K,DAI X G,et al. Genetic diversity analysis and core collection construction of Magnolia biondii germplasm[J]. Acta Horticulturae Sinica,2024,51(8) :1823–1832. (in Chinese)
[13] 张林娟,李向茂,奉树成. 紫薇种质资源与应用研究进展[J]. 广东农业科学,2024,51(2) :81–91. ZHANG L J,LI X M,FENG S C.Research progress on germplasm resources and application of Lagerstroemia indica[J]. Guangdong Agricultural Sciences,2024,51(2) :81–91.(in Chinese)
[14] 陈明堃,陈璐,孙维红,等. 建兰种质资源遗传多样性分析及核心种质构建[J]. 园艺学报,2022,49(1) :175–186. CHEN M K,CHEN L,SUN W H,et al. Genetic diversity analysis and core collection of Cymbidium ensifolium germplasm resources[J]. Acta Horticulturae Sinica,2022,49(1) :175–186. (in Chinese)
[15] 张保青,黄玉新,周珊,等. 广西割手密核心种质构建与关联分析[J]. 华中农业大学学报 ,2024,43(5) :75–81. ZHANG B Q,HUANG Y X,ZHOU S,et al. Construction and association analysis of core germplasm for Saccharum spontaneum L. in Guangxi[J]. Journal of Huazhong Agricultural University,2024,43 (5) :75–81. (in Chinese)
[16] 陈妍,杨午,万坤,等. 基于表型多样性构建山西大豆地方品种核心种质[J]. 中国油料作物学报,2025,47(1) :105–114. CHEN Y,YANG W,WAN K,et al.Establishment of soybean core collection based on phenotypic characters in Shanxi Province[J]. Chinese Journal of Oil Crop Sciences,2025,47(1) :105–114.(in Chinese)
[17] 丁艺冰,辛旭霞,冯智尊,等. 东北春播区糜子核心种质及其DNA分子身份证构建[J]. 作物学报,2024,50(5) :1181–1192. DING Y B,XIN X X,FENG Z Z,et al. Core germplasm and DNA molecular identity card of proso millet in Northeast Spring sowing region in China[J]. Acta Agronomica Sinica,2024,50(5) :1181–1192. (in Chinese)
[18] 明军,张启翔,兰彦平. 梅花品种资源核心种质构建[J]. 北京林业大学学报,2005,27(2) :65–69. MING J,ZHANG Q X,LAN Y P. Core collection of Prunus mume sieb. et zucc[J]. Journal of Beijing Forestry University,2005,27(2) :65–69. (in Chinese)
[19] 陈剑锋,钟声远,陈宇华,等. 基于花表型性状的蝴蝶兰品种资源多样性研究[J]. 热带作物学报,2023,44(3) :494–505. CHEN J F,ZHONG S Y,CHEN Y H,et al. Research on diversity of Phalaenopsis germplasm resources based on flowers phenotype traits[J]. Chinese Journal of Tropical Crops,2023,44(3) :494–505. (in Chinese)
[20] 王钦,黄捷,涂松,等. 蝴蝶兰不同品种表型性状遗传多样性分析[J]. 西南林业大学学报(自然科学) ,2023,43(6) :8–18. WANG Q,HUANG J,TU S,et al. Analysis of phenotypic genetic diversity of various Phalaenopsis varieties[J]. Journal of Southwest Forestry University (Natural Sciences) ,2023,43(6) :8–18. (in Chinese)
[21] 李佐,肖文芳,尤毅,等. 蝴蝶兰核心种质构建初探[J]. 广东农业科学,2013,40(1) :46–49. LI Z,XIAO W F,YOU Y,et al. Preliminary establishment of core collection for Phalaenopsis[J]. Guangdong Agricultural Sciences,2013,40(1) :46–49. (in Chinese)
[22] 中华人民共和国农业部. 植物新品种特异性、一致性和稳定性测试指南 蝴蝶兰:NY/T 2230—2012[S]. 北京:中国农业出版社,2013. [23] 胡晋,徐海明,朱军. 保留特殊种质材料的核心库构建方法[J]. 生物数学学报,2001,16(3) :348–352. HU J,XU H M,ZHU J. A method of constructing core collection reserving special germplasm materials[J]. Journal of Biomathematics,2001,16(3) :348–352. (in Chinese)
[24] 徐海明,胡晋,朱军. 构建作物种质资源核心库的一种有效抽样方法[J]. 作物学报,2000,26(2) :157–162. XU H M,HU J,ZHU J. An efficient method of sampling core collection from crop germpl asm[J]. Acta Agronomica Sinica,2000,26(2) :157–162. (in Chinese)
[25] 刘遵春,张春雨,张艳敏,等. 利用数量性状构建新疆野苹果核心种质的方法[J]. 中国农业科学,2010,43(2) :358–370. LIU Z C,ZHANG C Y,ZHANG Y M,et al. Study on method of constructing core collection of Malus sieversii based on quantitative traits[J]. Scientia Agricultura Sinica,2010,43(2) :358–370. (in Chinese)
[26] 刘艳阳,梅鸿献,杜振伟,等. 基于表型和SSR分子标记构建芝麻核心种质[J]. 中国农业科学,2017,50(13) :2433–2441. LIU Y Y,MEI H X,DU Z W,et al. Construction of core collection of sesame based on phenotype and molecular markers[J]. Scientia Agricultura Sinica,2017,50(13) :2433–2441. (in Chinese)
[27] 刘娟,廖康,赵世荣,等. 利用ISSR分子标记构建新疆野杏核心种质资源[J]. 中国农业科学,2015,48(10) :2017–2028. LIU J,LIAO K,ZHAO S R,et al. The core collection construction of Xinjiang wild apricot based on ISSR molecular markers[J]. Scientia Agricultura Sinica,2015,48(10) :2017–2028. (in Chinese)
[28] 彭枫,李阳,戴雨柔,等. 基于表型性状的菠菜核心种质构建[J]. 上海师范大学学报(自然科学版) ,2022,51(1) :9–19. PENG F,LI Y,DAI Y R,et al. Construction of spinach’s core germplasms based on its phenotypic traits[J]. Journal of Shanghai Normal University (Natural Sciences) ,2022,51(1) :9–19. (in Chinese)
[29] 陈建华,曲凯伦,张云程,等. 基于表型性状的酸枣核心种质构建[J]. 沈阳农业大学学报,2024,55(2) :176–186. CHEN J H,QU K L,ZHANG Y C,et al. Construction of Ziziphus jujuba var. spinosa core collection based on phenotypic traits[J]. Journal of Shenyang Agricultural University,2024,55(2) :176–186. (in Chinese)
[30] 郎彬彬,黄春辉,朱博,等. 基于果实相关性状的江西野生毛花猕猴桃初级核心种质的构建方法研究[J]. 果树学报,2016,33(7) :794–803. LANG B B,HUANG C H,ZHU B,et al. Study on the method of constructing a primary core collection of Jiangxi wild Actinidia eriantha based on fruit traits[J]. Journal of Fruit Science,2016,33(7) :794–803. (in Chinese)
[31] PEETERS J P,MARTINELLI J A. Hierarchical cluster analysis as a tool to manage variation in germplasm collections[J]. Theoretical and Applied Genetics,1989,78(1) :42–48.
[32] 缪黎明,王神云,邹明华,等. 园艺作物核心种质构建的研究进展[J]. 植物遗传资源学报,2016,17(5) :791–800. MIAO L M,WANG S Y,ZOU M H,et al. Review of the studies on core collection for horticultural crops[J]. Journal of Plant Genetic Resources,2016,17(5) :791–800. (in Chinese)
[33] 侯志强,王丽慧,赵孟良,等. 基于表型数据的菊芋核心种质初步构建[J]. 分子植物育种,2021,19(10) :3463–3472. HOU Z Q,WANG L H,ZHAO M L,et al. Preliminary construction of core collection of Jerusalem artichoke based on phenotypic data[J]. Molecular Plant Breeding,2021,19(10) :3463–3472. (in Chinese)
[34] 崔艳华,邱丽娟,常汝镇,等. 黄淮夏大豆(G. max) 初选核心种质代表性检测[J]. 作物学报,2004,30(3) :284–288. CUI Y H,QIU L J,CHANG R Z,et al. Representative test for primary core collection of summer sowing soybeans in Huanghuai region of China[J]. Acta Agronomica Sinica,2004,30(3) :284–288. (in Chinese)
[35] HU J,ZHU J,XU H M. Methods of constructing core collections by stepwise clustering with three sampling strategies based on the genotypic values of crops[J]. Theoretical and Applied Genetics,2000,101(1) :264−268.
[36] 王建成,胡晋,张彩芳,等. 建立在基因型值和分子标记信息上的水稻核心种质评价参数[J]. 中国水稻科学,2007,21(1) :51–58. WANG J C,HU J,ZHANG C F,et al. Evaluating parameters of rice core collections based on genotypic values and molecular marker information[J]. Chinese Journal of Rice Science,2007,21(1) :51–58. (in Chinese)




 下载:
下载: