Genome Sequencing and Antimicrobial Metabolite Synthesis Genes of Bacillus mojavensis JK-18
-
摘要:目的
对核桃黑斑病病原拮抗菌莫海威芽孢杆菌JK-18 进行全基因组测序和基因功能注释,初筛多种活性代谢产物合成基因簇。
方法采用PacBio测序技术对从核桃根际土壤中筛选得到的莫海威芽孢杆菌JK-18进行全基因组测序,获得菌株基因组特征信息、基因功能注释及分类和次级代谢产物等关键信息。
结果JK-18的基因组为一条环状闭合DNA,大小为4 166 746 bp,由1个环状染色体组成,共有4 138 个蛋白质编码基因。基因组中共预测到4种类型的CRISPR。GO、KEGG、EGGNOG 、CAZY和CARD 数据库中分别注释到
5844 、2299 、3257 、183和7 个功能基因。应用在线软件AntiSMASH预测,该菌株基因组中包含Lichenysin、Macrobrevin和Aurantinin等聚酮类,Surfactin、 Bacillomycin 、Plipastatin、Fengycin等脂肽类,Dumulmycin及Dactylocycline A大环内酯类等多种活性代谢产物合成基因簇。结论通过全基因组测序及相关功能基因的挖掘分析,表明该菌株具有较好的生防潜能。相关研究结果对抗菌物质的后期深入研究具有重要的参考价值。
Abstract:ObjectiveSequence and annotation of Bacillus mojavensis JK-18 genome were obtained, and gene clusters related to the synthesis of antimicrobial secondary metabolites in the bacterium identified for the development of a biocontrol agent for walnut tree blight.
MethodThe whole genome of B. mojavensis JK-18 isolated from the rhizosphere soil of walnut trees was sequenced using the PacBio technology. The characteristics, functions, and classification of the gene and secondary metabolites secreted by the bacterium were analyzed.
ResultsThe genome was a closed circular DNA with a size of 4 166 746 bp containing 4 138 protein-coding genes and 4 kinds of CRISPR. It had 5 844, 2 299, 3 257, 183, and 7 functional genes annotated in GO, KEGG, EGGNOG, CAZY, and CARD databases, respectively. Predicted by antiSMASH, the genes related to the synthesis of secondary metabolites including the antimicrobial substances like polyketide compounds (e.g., Lichenysin, Macrobrevin and Aurantinin), peptidolipids (e.g., Surfactin, Bacillomycin, Plipastatin and Fengycin), and macrolides (e.g., Dumulmycin and Dactylocycline A).
ConclusionIdentification of the genes in B. mojavensis JK-18 that related to the syntheses of secondary metabolites with an antimicrobial property would aid further study leading to the development of bioagents for blight prevention and control on walnut trees.
-
Keywords:
- walnut /
- Bacillus mojavensis /
- whole-genome sequencing /
- synthetic gene cluster
-
0. 引言
【研究意义】核桃作为一种重要的经济林果,目前除了东北地区的严寒地带和南部沿海等地没有或极少种植外,其他27个省份均有栽培[1]。核桃黑斑病是一种危害较大的核桃细菌性病害,传播力强、危害程度高,经常造成落花落果,对核桃的果实品质也有一定的影响[2]。长期以来,该病以传统的化学方法为主要防治手段,这些手段通常会造成环境污染和果实农药残留,严重影响人们的生命健康。随着生物技术的不断发展,绿色防治技术应运而生。【前人研究进展】目前,利用细菌菌株之间相互拮抗作用来实施绿色综合防治越来越受到国内外研究者的关注[3−6]。芽孢杆菌是一种自然界广泛分布的具有生防作用的细菌,其便于培养、抑菌谱广等特点引起了广大研究者的关注。多种芽孢杆菌具有较好的促生、抗病及改良土壤的作用。张琦等[7]从番茄根部分离获得内生菌贝莱斯芽孢杆菌(Bacillus velezensis),经研究发现,该菌株对防治番茄灰霉病具有一定的作用。张丽芳等[8]发现解淀粉芽孢杆菌(Bacillus amyloliquefaciens)B235能显著提升花生的发芽率和出苗率,且能使花生根腐病病情指数显著降低。李莹等[9]从花生根际土壤中分离到1株对多种花生病原真菌具有较好的抑制作用的贝莱斯芽孢杆菌。研究结果显示,该菌株还具有较强的促生作用。宋波等[10]分离得到1株枯草芽孢杆菌(Bacillus subtilis),该菌株对芒果暹罗刺盘孢菌(Colletotrichum siamense)有较强的抑制作用。近年来的研究表明,抗生素、细菌素及细胞壁降解酶等物质是芽孢杆菌产生较好生防效果的关键物质[11]。全基因组测序的快速发展为抗菌相关基因及抗菌物质的预测提供了切实有效的方法[12]。【本研究切入点】莫海威芽孢杆菌(Bacillus mojavensis)JK-18是本课题组从核桃根际土壤分离的1株芽孢杆菌[6]。经研究发现,该菌株对核桃黑斑病病原具有较好的拮抗作用,有望用于核桃细菌性黑斑病的生物防治,但对拮抗物质及其机理还有待深入研究。【拟解决的关键问题】本研究在实验室前期研究的基础上,以莫海威芽孢杆菌JK-18为研究对象,对其进行全基因组测序与分析,采用在线软件antiSMASH挖掘其潜在的活性代谢产物合成基因簇,为后期通过生物化学方法深入分析抗菌物质、研究其拮抗机制奠定基础。
1. 材料与方法
1.1 试验材料
供试菌株为本课题组通过平板对峙法从核桃树根际土壤中筛选并鉴定的1株拮抗菌株[6]。研究表明,该菌株对陇南核桃黑斑病病原菌(Xanthomonas campestris )具有较好的拮抗作用。
供试培养基为细菌LB 液体培养基(1 L):胰蛋白胨10.0 g,酵母膏5.0 g,NaCl 10.0 g;LB平板培养基:胰蛋白胨10.0 g,酵母膏5.0 g,NaCl 10.0 g,15.0 g琼脂粉。
1.2 试验方法
1.2.1 测序文库构建
将菌株JK-18从冰箱取出后,在LB培养基上进行活化培养,然后挑取单菌落转接至适量250 mL LB 液体培养基中,37 ℃振荡培养12 h,于4 ℃、10 000 r·min−1 离心5 min 后弃上清,纯化后送百迈客生物技术有限公司进行高通量测序。利用细菌基因组提取试剂盒提取基因组DNA后,以g-TUBE打断DNA样品,然后对打断的DNA样品进行末端修复,连接哑铃型接头,核酸外切酶消化后使用BluePippin进行目的片段筛选,获得测序文库。
1.2.2 菌株JK-18全基因组测序
基因组测序后,所得原始数据利用Readfq 软件除去低质量reads,保留长度在2 kb以上的reads进行基因组组装,组装后经纠错处理后对基因组的重复序列、编码基因、非编码RNA、前噬菌体、基因岛、CRISPR等进行组分分析。
1.2.3 基因组功能预测及注释
利用相关数据库和在线工具对相关功能基因进行预测。利用Non-Redundant Protein Database进行序列比对(http://www.bioinformatics.org/cd-hit);利用GO数据库(http://geneontology.org)、KEGG数据库( https://www.kegg.jp )、eggNOG数据库( http://eggnogdb.embl.de )、CAZY 数据库( http://www.cazy.org )、CARD数据库(https://card.mcmaster.ca)和antisMASH(https://antismash.secondarymetabolites.org)等在线工具等进行基因功能注释。
1.2.4 基因组图谱分析
测序所得的基因组序列、编码基因预测及非编码RNA 等信息文件组装成GBK文件后,利用Circos v0.66[13]绘制单个样本基因组圈图,全面展示测序基因组特征。
2. 结果与分析
2.1 JK-18全基因组序列系统演化分析
JK-18菌株全基因组序列在GenBank数据库中进行BLAST,挑选同源序列分值较高的代表性菌株以邻接法(NJ)构建进化树。结果表明,JK-18与Bacillus mojavensis(NZ-CP051464.1:201-1640)的序列相似程度较高,结合试验过程中的形态学鉴定,确定该菌株为Bacillus mojavensis(图1)。
2.2 菌株JK-18全基因组基本特征
利用三代PacBio RS II 测序方法对JK-18进行测序后,所得细菌基因组序列全长为4 166 746 bp,由一条环状闭环的DNA组成。编码基因数量为
4138 ,其中,基因总长度为3 649 620 bp。最长16 560 bp,最短90 bp,平均881 bp,GC含量43.93%。经测序组装后,预测该基因组的重复序列总长度为4697 bp,占细菌基因组的0.11%。非编码RNA中,tRNA和rRNA数量分别是86和30个。2.3 CRISPR序列预测
使用CRT v1.2[14]对基因组进行规律间隔成簇短回文重复序列CRISPR(clustered regularly interspaced palindromic repeats)预测,发现该细菌共有4种类型的CRISPR,结果见表1。
表 1 CRISPR预测结果统计Table 1. Prediction by CRISPR规律间隔
成簇短回文
重复序列
CRISPR重复数量
Repeat number平均重复长度
Average repeat
length /bp间隔数量
Spacer number平均间隔
长度Average
Spacer length/
bp1 5 24 4 36 2 11 33 10 42 3 3 23 2 37 4 10 29 9 57 2.4 基因组圈图分析
根据测序所得信息tRNA、rRNA、重复序列、GC含量以及基因功能信息等,利用相关软件绘制基因组圈图(图2)。通过结构多样、位置关系清楚的圈图可以更为全面、直观地分析基因组组分在基因组上的位置关系。从图中可以发现,最外一圈为基因组的大小,每个刻度为5 kb;第二圈和第三圈分别表示基因组正链和负链上的基因,不同的颜色表示不同的直系同源物组(COG);第四圈表示重复序列;第五圈中主要为非编码的tRNA和rRNA,分别以蓝色和紫色标记;第六圈为GC含量,分别以浅黄色和蓝色表示此区域GC含量高于或低于基因组的平均含量,峰值的大小与平均GC含量成正相关。最内圈表示GC-SKEW,深灰色表示GC-SKEW大于0的区域,红色表示GC-SKEWGC小于0的区域。
2.5 基因功能注释
2.5.1 GO数据库注释
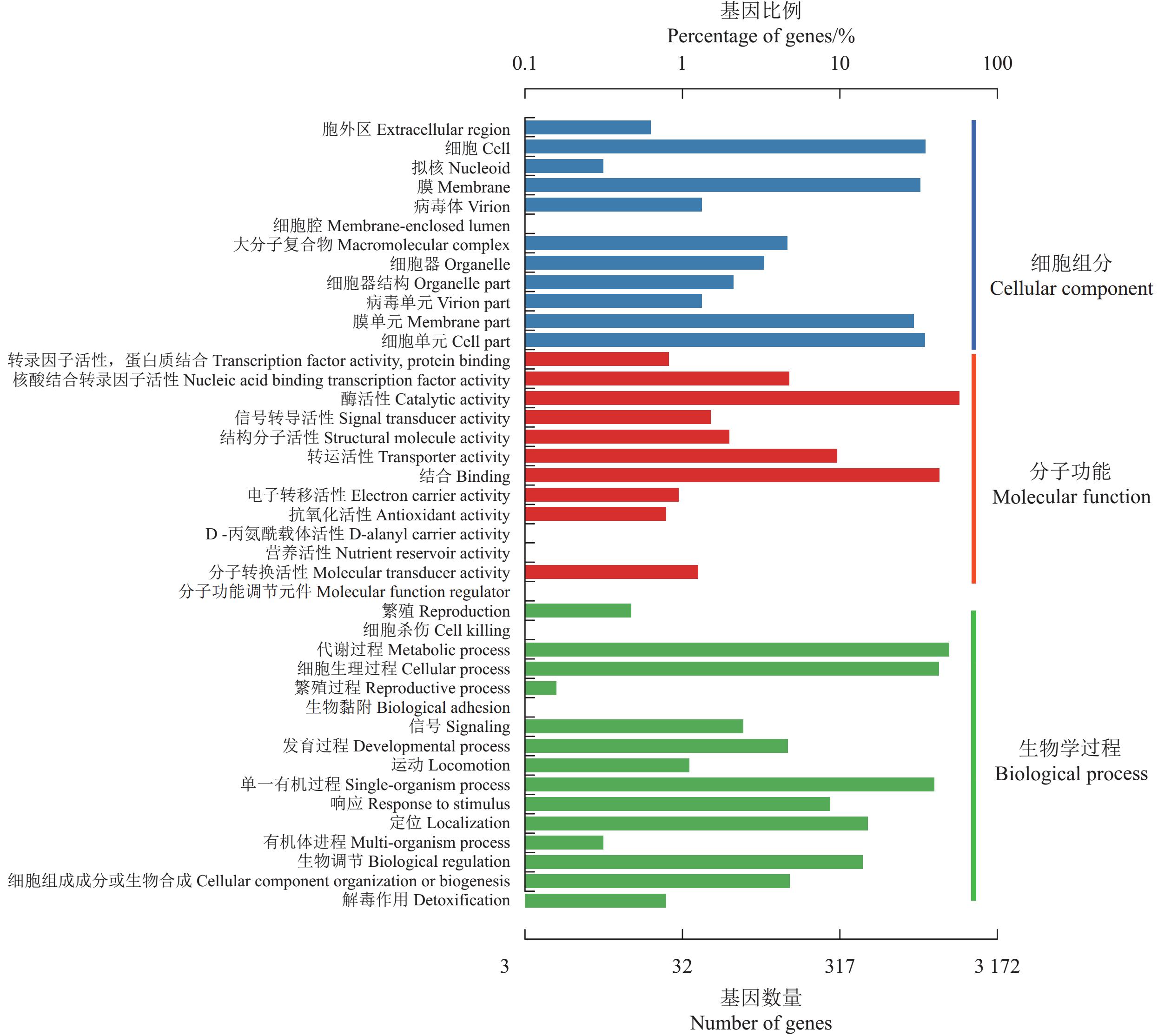
JK-18的编码基因的GO注释如图3所示。该基因组中,与细胞组分(cellular component)相关的基因数为
4606 个,主要与细菌的亚细胞结构有关,所编码物质组成细菌菌体细胞壁、细胞膜及细胞外区域及大分子复合物等结构成分;与分子功能(molecular function)相关的基因数为3879 个,主要与细胞内各类转录因子、信号转导相关因子及核内各活性因子的功能有关;与生物过程(biological process)相关的基因数为5844 个,这些基因主要与细胞内错综复杂的各类代谢活动有关。由图中可以看出,与细胞生物过程有关的代谢过程、细胞过程及单有机体过程是富集基因最多的生物学过程,与此有关的基因数占比最高,分别为27%、23%和22%。表明该菌株为一株代谢旺盛、增殖能力较强的微生物,尤其是细胞内与代谢相关的基因较多,次生代谢物合成能力强,具有拮抗菌株的较好潜能。2.5.2 KEGG功能注释
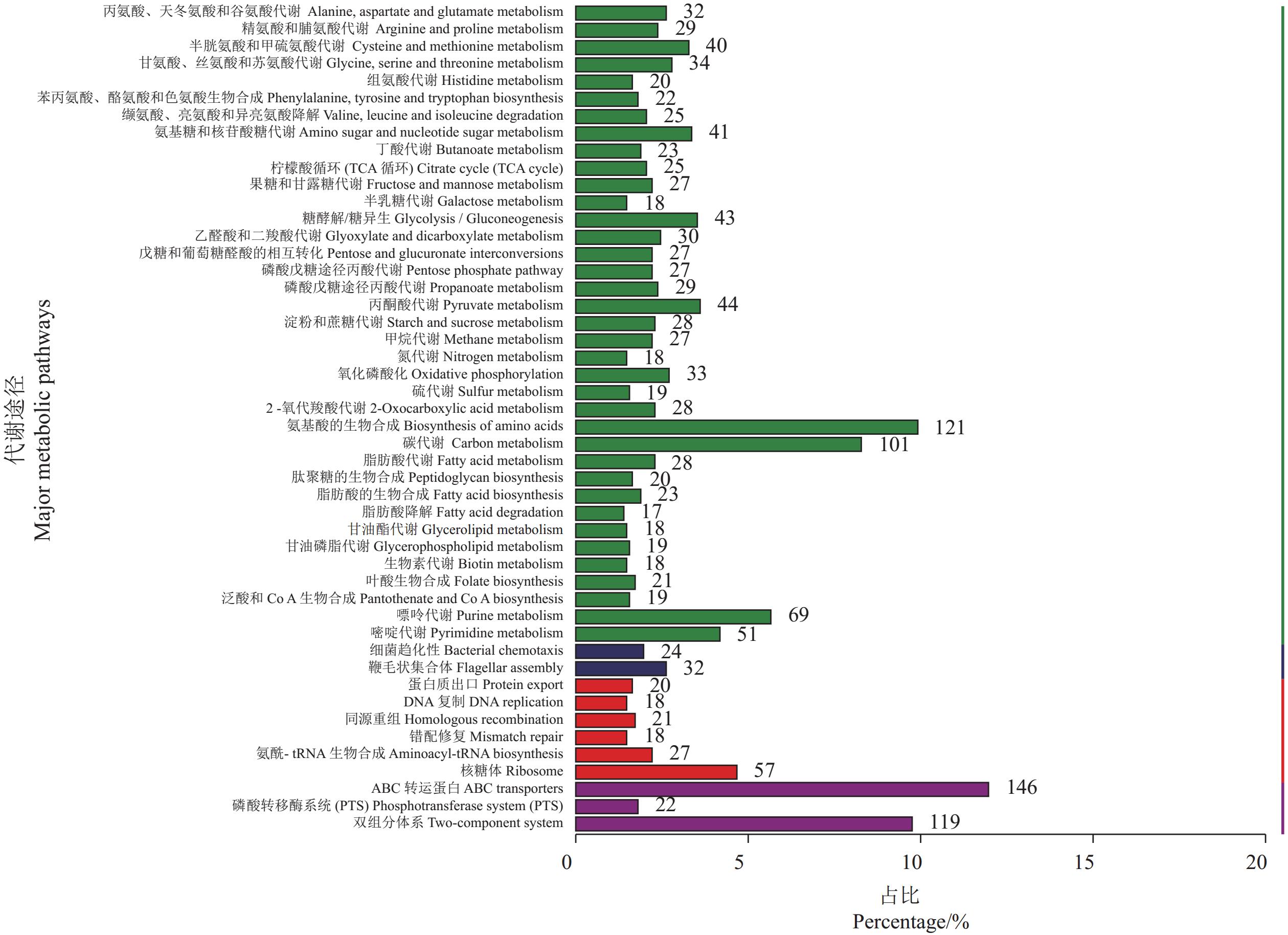
从图4可以看出,涉及到新陈代谢、细胞过程、遗传信息过程和环境信息过程这四大类KEGG一级功能类别中的编码基因共有
2300 个。从所占比例可以看出,与新陈代谢相关的基因所占比例最高,其次为环境信息过程有及环境适应相关的基因。因细胞分裂增殖的需要,与氨基酸生物合成通路相关的基因数量为121,与碳代谢、嘌呤代谢和嘧啶代谢相关的基因数量分别为101、69和51;与核蛋白体代谢相关的基因数目为57;环境信息处理有关基因中,与ABC质膜转运功能相关的基因数量146。上述结果表明,JK-18细胞分裂增殖及营养物质代谢能力较强,尤其是ABC质膜转运相关的基因较高表达。这些基因功能的展示有助于细菌发挥重要的拮抗作用。2.5.3 eggNOG数据库注释
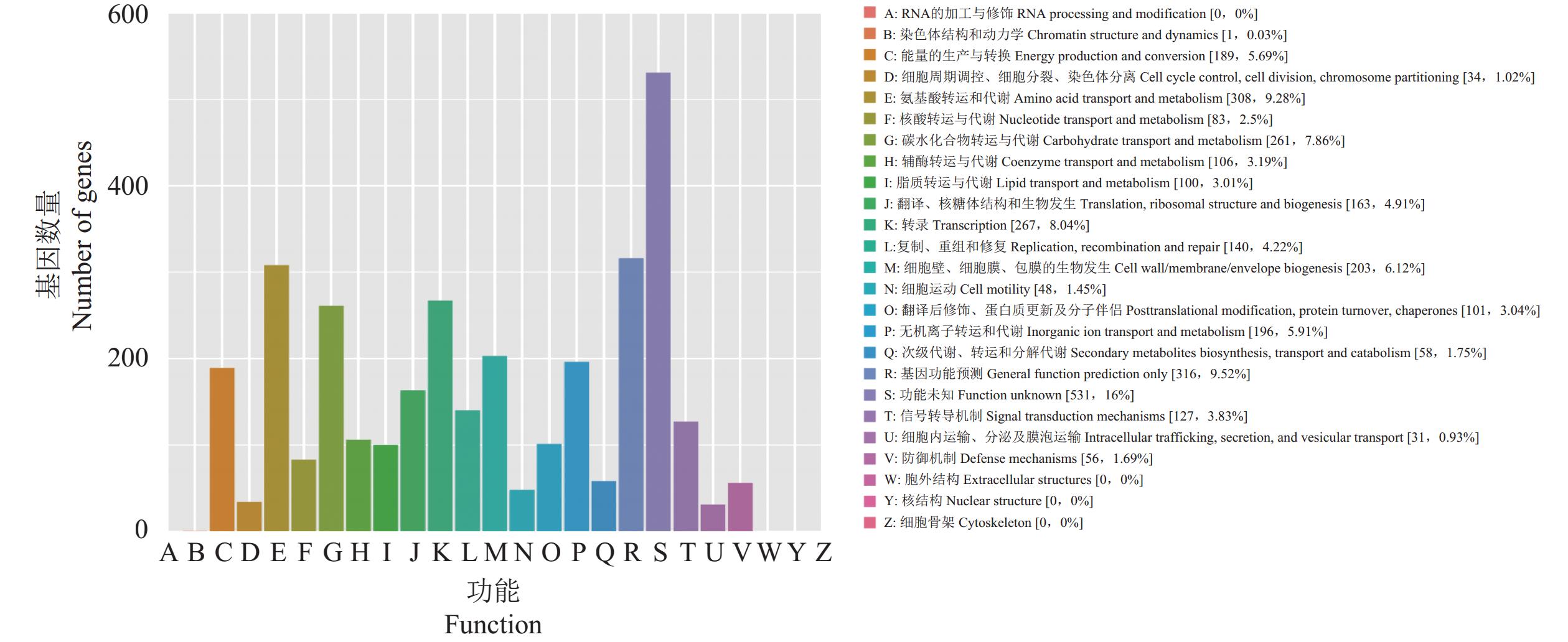
eggNOG[15]是收录生物直系同源基因簇的数据库。根据eggNOG数据库比对结果,莫海威芽孢杆菌功能基因根据其功能可分为22类(图5)。除了一些功能未知的基因之外,其基因组中一般性功能基因为316个,占比为9.52%;氨基酸转运及代谢相关的基因有308个,占比为9.28%;与转录有关的基因有267个,占比为8.04%;与碳水化合物代谢相关的基因有261个,占比为7.86%;与细胞壁(膜)形成有关的基因为203个,占比为6.12%。除了与细菌基本的代谢功能相关的基因外,与无机离子转运、次生代谢物的合成、辅酶的转运、脂类合成以及与核糖体相关的基因数量分别为196、58、106、100、163。由此表明,该菌株新陈代谢能力较为旺盛,产生次生代谢物的能力较强,这与其作为一种重要的生防细菌,功能是相一致的。
2.5.4 CAZY 数据库注释
CAZY[16](Carbohydrate-active enzymes database)是收录碳水化合物活性酶的数据库。按其功能将该数据库中蛋白分为六大类别,分别为:糖苷水解酶、糖基转移酶、多糖裂解酶、碳水化合物酯酶、辅助氧化还原酶和非催化的结合碳水化合物的功能域。经与CAZY 数据库进行比对分析,基因组中共有183个基因编码的蛋白质结构域属于CAZy 家族。由表2可以看出,该菌株中糖苷水解酶与糖基转移酶所占基因数量较多,分别占32.60%和25.54%,接下来依次为碳水化合物酯酶、碳水化合物结合域、多糖裂解酶及氧化还原酶,预示着该菌株在代谢物的合成、分解及细菌细胞壁的合成方面都具有较好的活性。
表 2 CAZY 数据库注释结果Table 2. CAZY annotation类别
Class definition基因数量
Number of genes占比
Percentage/%糖苷水解酶
Glycoside Hydrolases60 32.60 糖基转移酶
Glycosyl Transferases47 25.54 多糖裂解酶
Polysaccharide Lyases7 3.80 碳水化合物酯酶
Carbohydrate Esterases35 19.02 氧化还原酶
Auxiliary Activities6 3.26 碳水化合物结合域
Carbohydrate-Binding Modules28 15.76 2.5.5 CARD 数据库注释
CARD[17](Comprehensive Antibiotic Research Database)是目前使用最为广泛的收录抗生素抗性相关信息的数据库。CARD包含耐药基因、耐药受体及抗性机制信息。菌株JK-18的基因组中分布有7个耐药基因,分别为:GE002632、GE002054、GE000263、GE000248、GE000844、GE003561和GE001428。其中,GE002632、GE002054、GE000263和GE000248分别具有氨基糖苷类抗生素(aminoglycoside antibiotic)抗性、喹诺酮类抗生素 (fluoroquinolone antibiotics)抗性、林可霉素抗生素 (lincosamide antibiotic)抗性和大环内酯类化合物(macrolide antibiotics)抗性。GE000844和GE003561具肽类抗生素(peptide antibiotics)抗性 。GE001428基因对四环素抗生素(tetracycline antibiotics)、氨基糖苷类抗生素(aminoglycoside antibiotic)和苯尼考尔抗生素(phenicol antibiotic)具多重耐药性,推测JK-18可能对常见的一些抗生素具有一定的抗药性。
2.5.6 分泌蛋白信息预测
拮抗菌株能够分泌一些重要的分泌蛋白,对其他微生物具有一定的拮抗作用,有望作为重要的生防细菌。周京龙等[18]筛选出1株能够降解 β-1,4 糖苷键的棉花内生蜡状芽孢杆菌,通过Fosmid文库,初步证明该菌体内能够降解 β-1,4 糖苷键的物质为环糊精糖基转移酶(CGTase),为蜡状芽孢杆菌(Bacillus cereus)的分泌蛋白。舒霞[19]研究表明,贝莱斯芽孢杆菌SQR9产生的一种独特分泌蛋白Yuk E可有效提升其在植物根际的定殖能力,并发挥重要的促生作用。本研究中,经基因组结构预测分析,共筛选到分泌蛋白(secretory protein)121个(表3),但其具体生理功能还需后期研究进一步确认。
表 3 芽孢杆菌JK-18 分泌蛋白信息预测Table 3. Predicted secretory protein from JK-18描述
Description基因列表
Gene list糖苷水解酶
Glycoside hydrolasesGE000017;GE000173;GE000280;GE000303;GE000343;GE000433;GE000547;GE000564;GE000575;GE000579;GE000691;GE000700;
GE000705;GE000770;GE000822;GE000832;GE001060;GE001092;GE001166;GE001281;GE001307;GE001384;GE001502;GE001876;
GE001929;GE001932;GE001987;GE001990;GE001991;GE002042;GE002065;GE002103;GE002652;GE002734;GE002809;GE002833;
GE002842;GE002956;GE002972;GE002974;GE002991;GE003051;GE003067;GE003072;GE003086;GE003314;GE003374;GE003384;
GE003385;GE003420;GE003421;GE003551;GE003797;GE003852;GE003901;GE003920;GE003931;GE003967;GE004012;GE004114糖基转移酶
Glycosyl transferasesGE000436;GE000565;GE000720;GE000721;GE000722;GE000750;GE000751;GE000862;GE001017;GE001261;GE001334;GE001405;
GE001406;GE001435;GE001456;GE001460;GE001653;GE002129;GE002171;GE002201;GE002241;GE002255;GE002506;GE003047;GE003048;GE003108;GE003399;GE003401;GE003403;GE003404;GE003405;GE003519;GE003520;GE003524;GE003527;GE003533;GE003534;GE003540;GE003542;GE003545;GE003741;GE003777;GE003782;GE003783;GE003790;GE003839;GE004060多糖裂解酶
Polysaccharide lyasesGE000696;GE000697;GE000701;GE000743;GE002047;GE003463;GE003627 糖基转移酶
Carbohydrate esteraseGE000117;GE000160;GE000167;GE000318;GE000368;GE000693;GE000699;GE000787;GE000861;GE000974;GE001097;GE001122;
GE001197;GE001322;GE001681;GE001801;GE002134;GE002146;GE002183;GE002256;GE002373;GE002584;GE002629;GE002986;
GE003013;GE003020;GE003042;GE003158;GE003177;GE003328;GE003354;GE003410;GE003469;GE003876;GE003908辅助活性
Auxiliary activitiesGE000911;GE000941;GE001125;GE002069;GE002826;GE003428 碳水化合物结合模块
Carbohydrate-binding modulesGE000017;GE000303;GE000564;GE000746;GE000944;GE000949;GE001288;GE001359;GE001385;GE001398;GE001500;GE001501;
GE001531;GE001958;GE001987;GE001991;GE002045;GE002112;GE002128;GE002311;GE002652;GE002741;GE002769;GE002956;
GE003051;GE003234;GE003268;GE0034212.5.7 潜在抑菌物质的预测
目前,部分微生物所产生的次生代谢产物是抗菌剂的重要来源。软件antiSMASH v5.0.0[20]是一种能够较为快速鉴定和分析相关微生物基因组序列中生物合成基因簇(BGC)的在线工具。antiSMASH不仅能够准确识别编码所有已知广泛化学类别的次级代谢产物的基因簇,而且还可以提供详细的序列分析。antiSMASH 分析的结果表明(表4),菌株JK-18有多个具有潜在抗菌功能的基因簇。其 中 JK-18中细菌素基 因簇与Bacillus velezensis FZB42的Polyketide、Fengycin、Plipastatin、Paenibactin、Subtilosin A、Bacilysin生物合成基因簇的相似性为100%,与Bacillus velezensis FZB42的 Surfactin生 物 合 成 基 因 簇 相 似性为86%。与 Bacillus subtilis subsp. subtilis str. 168 Bacillibactin的生物合成基因簇的相似性为100%。说明 JK-18 具有合成聚酮化合物、环状脂肽和铁载体等抗菌物质的潜力。
表 4 莫海威芽孢杆菌JK-18基因组中预测的次生代谢物相关的基因簇及其合成的抑菌物质Table 4. Genes related to secondary metabolites and antimicrobial substances in B. mojavensis
3. 讨论与结论
目前,微生物全基因组测序已成为生物技术领域一项重要的高效检测技术,已广泛应用于多种微生物特殊功能基因的分析和预测。通过全基因组测序技术预测微生物次生代谢物种类并揭示其抗菌机理引起了人们的极大关注。Shen等[21]对一株采自长白山的芽孢杆菌TK-2进行全基因组测序,从624个特殊基因簇中发现了一些耐冷基因,对推动低温生物工程技术的应用具有重要意义。Li等[22]通过全基因组测序技术预测到链霉菌HP-A2021菌株33个次生代谢物合成基因簇,包括Thiotetroamide、 Alkylresorcinol、Coelichelin和Geosmin。揭示了HP-A2021具有较好的病原微生物抗性。Duan等[23]通过全基因组测序技术对分离自白冰洋中链霉菌Streptomyces sp. R527F菌株进行研究,共鉴定出至少30个次生代谢物合成基因簇,为后期开展抗菌物质的分析分离及鉴定提供理论基础。作为一种从核桃根际土壤中分离的一种重要拮抗菌株,莫海威芽孢杆菌对于核桃细菌性黑斑病的综合防治具有重要意义。但对于其拮抗机制的探索亟待开展后期研究工作。
绝大多数微生物可以产生广谱性抗菌物质,这些物质具有较好的抗真菌、抗病毒甚至具有抗肿瘤活性[24]。全基因组测序技术已成为各类菌株次生代谢物合成基因簇发掘的重要工具,对潜在的生防菌株及其次生代谢物的发现具有重要意义。研究表明,抗生素、细菌素、细胞壁降解酶及分泌蛋白等类抗菌物质是芽孢杆菌具有生防作用的一系列重要物质。以生防模式菌B. velezensis FZB42为对照,antiSMASH 在线预测分析结果表明菌株JK-18全基因组上包含活性代谢产物合成的同源基因簇,包括Lichenysin、Macrobrevin和Aurantinin等聚酮类化合物,Surfactin、 Bacillomycin 、Plipastatin、Fengycin等脂肽类化合物,Dumulmycin及Dactylocycline A大环内酯类抗菌化合物等。另外还有Calyculin A、Glucorhamnan及S-layer glycan等。其中,Polyketide、Fengycin、Plipastatin、Bacillibactin、Paenibactin、Bacillibactin、Subtilosin A、Bacilysin与相关对照菌株的相似性达100%,Bacillibactin与相关对照菌株的相似性为70%。这些研究表明,该菌株可能合成多种抗菌物质,对细菌和真菌可能具有广谱的抑制活性,有望作为生防菌株用于核桃黑斑病的综合防治。在预测过程中,莫海威芽孢杆菌基因组有4种类型的成簇的、有规律间隔的短回文重复序列结构家族。这种序列对于DNA的重组、修复及原核生物的免疫防御系统关系密切[25]。目前,随着CRISPR/Cas 技术的出现,人们有望通过这些重复序列设计具有特定杀菌作用的 CRISPR 抗菌剂[26]。另外,细菌的分泌蛋白中部分成分对靶细菌也具有较好的拮抗作用,这些也为人们开展抗菌化合物的分离鉴定提供了一条重要的研究思路,具有重要的研究价值。
综上所述,JK-18菌株的全基因组测序及功能预测结果,对相关功能基因的发掘及功能研究具有重要的理论价值。但是,JK-18菌株的抗菌机制有待进一步的研究。
-
表 1 CRISPR预测结果统计
Table 1 Prediction by CRISPR
规律间隔
成簇短回文
重复序列
CRISPR重复数量
Repeat number平均重复长度
Average repeat
length /bp间隔数量
Spacer number平均间隔
长度Average
Spacer length/
bp1 5 24 4 36 2 11 33 10 42 3 3 23 2 37 4 10 29 9 57 表 2 CAZY 数据库注释结果
Table 2 CAZY annotation
类别
Class definition基因数量
Number of genes占比
Percentage/%糖苷水解酶
Glycoside Hydrolases60 32.60 糖基转移酶
Glycosyl Transferases47 25.54 多糖裂解酶
Polysaccharide Lyases7 3.80 碳水化合物酯酶
Carbohydrate Esterases35 19.02 氧化还原酶
Auxiliary Activities6 3.26 碳水化合物结合域
Carbohydrate-Binding Modules28 15.76 表 3 芽孢杆菌JK-18 分泌蛋白信息预测
Table 3 Predicted secretory protein from JK-18
描述
Description基因列表
Gene list糖苷水解酶
Glycoside hydrolasesGE000017;GE000173;GE000280;GE000303;GE000343;GE000433;GE000547;GE000564;GE000575;GE000579;GE000691;GE000700;
GE000705;GE000770;GE000822;GE000832;GE001060;GE001092;GE001166;GE001281;GE001307;GE001384;GE001502;GE001876;
GE001929;GE001932;GE001987;GE001990;GE001991;GE002042;GE002065;GE002103;GE002652;GE002734;GE002809;GE002833;
GE002842;GE002956;GE002972;GE002974;GE002991;GE003051;GE003067;GE003072;GE003086;GE003314;GE003374;GE003384;
GE003385;GE003420;GE003421;GE003551;GE003797;GE003852;GE003901;GE003920;GE003931;GE003967;GE004012;GE004114糖基转移酶
Glycosyl transferasesGE000436;GE000565;GE000720;GE000721;GE000722;GE000750;GE000751;GE000862;GE001017;GE001261;GE001334;GE001405;
GE001406;GE001435;GE001456;GE001460;GE001653;GE002129;GE002171;GE002201;GE002241;GE002255;GE002506;GE003047;GE003048;GE003108;GE003399;GE003401;GE003403;GE003404;GE003405;GE003519;GE003520;GE003524;GE003527;GE003533;GE003534;GE003540;GE003542;GE003545;GE003741;GE003777;GE003782;GE003783;GE003790;GE003839;GE004060多糖裂解酶
Polysaccharide lyasesGE000696;GE000697;GE000701;GE000743;GE002047;GE003463;GE003627 糖基转移酶
Carbohydrate esteraseGE000117;GE000160;GE000167;GE000318;GE000368;GE000693;GE000699;GE000787;GE000861;GE000974;GE001097;GE001122;
GE001197;GE001322;GE001681;GE001801;GE002134;GE002146;GE002183;GE002256;GE002373;GE002584;GE002629;GE002986;
GE003013;GE003020;GE003042;GE003158;GE003177;GE003328;GE003354;GE003410;GE003469;GE003876;GE003908辅助活性
Auxiliary activitiesGE000911;GE000941;GE001125;GE002069;GE002826;GE003428 碳水化合物结合模块
Carbohydrate-binding modulesGE000017;GE000303;GE000564;GE000746;GE000944;GE000949;GE001288;GE001359;GE001385;GE001398;GE001500;GE001501;
GE001531;GE001958;GE001987;GE001991;GE002045;GE002112;GE002128;GE002311;GE002652;GE002741;GE002769;GE002956;
GE003051;GE003234;GE003268;GE003421表 4 莫海威芽孢杆菌JK-18基因组中预测的次生代谢物相关的基因簇及其合成的抑菌物质
Table 4 Genes related to secondary metabolites and antimicrobial substances in B. mojavensis

-
[1] 徐永杰,裴东,马庆国,等. 遗传标记在核桃属植物遗传多样性研究中的应用[J]. 中国农学通报,2014,30(13) :1−5. DOI: 10.11924/j.issn.1000-6850.2013-2533 XU Y J,PEI D,MA Q G,et al. The application of genetic markers in the study of walnut genetic diversity[J]. Chinese Agricultural Science Bulletin,2014,30(13) :1−5. (in Chinese) DOI: 10.11924/j.issn.1000-6850.2013-2533
[2] 王瀚,卓平清,王让军,等. 甘肃陇南核桃黑斑病病原菌的分离鉴定及其致病性研究[J]. 中国果树,2018(4) :69−71. WANG H,ZHUO P Q,WANG R J,et al. Studies on identification and pathogenicity of walnut black spot in Longnan,Gansu province[J]. China Fruits,2018(4) :69−71. (in Chinese)
[3] 王迪,高岩,吴小双,等. 两株贝莱斯芽胞杆菌鉴定及其对薄壳山核桃黑斑病病原菌的拮抗效果[J]. 中国生物防治学报,2022,38(6) :1572−1581. WANG D,GAO Y,WU X S,et al. Identification of two strains of Bacillus velezensis isolated from Carya illinoensis leaf and their antagonistic effects on pecan black spot pathogen[J]. Chinese Journal of Biological Control,2022,38(6) :1572−1581. (in Chinese)
[4] 瞿佳,孙晓宇,赵玲侠,等. 核桃黑斑病生防链霉菌YNF36的鉴定及其抑菌活性测定[J]. 微生物学通报,2022,49(11) :4727−4739. QU J,SUN X Y,ZHAO L X,et al. Identification and activity of Streptomyces YNF36 against walnut blight[J]. Microbiology China,2022,49(11) :4727−4739. (in Chinese)
[5] 瞿佳,门欣,陈锐,等. 核桃黑斑病拮抗放线菌WMF106的筛选、鉴定及防效[J]. 微生物学通报,2021,48(10) :3621−3631. QU J,MEN X,CHEN R,et al. Screening,identification and biocontrol effect of antagonistic actinomycete WMF106 against walnut blight[J]. Microbiology China,2021,48(10) :3621−3631. (in Chinese)
[6] 王瀚,卓平清,王让军,等. 陇南核桃黑斑病病原菌拮抗菌筛选及鉴定[J]. 经济林研究,2018,36(1) :64−68,74. WANG H,ZHUO P Q,WANG R J,et al. Screening and identification of antagonistic bacteria against Xanthomonas campestris pv. campestris isolated from walnut blight in Longnan region[J]. Nonwood Forest Research,2018,36(1) :64−68,74. (in Chinese)
[7] 张琦,刘应敏,杨东燕,等. 贝莱斯芽孢杆菌SM2对番茄灰霉病的生防效果[J]. 中国瓜菜,2024,37(2) :66−73. ZHANG Q,LIU Y M,YANG D Y,et al. Biological control effects of against itomatogray mold by Bacillus velezensis SM2 Botrytis cinerea causing tomato gray mold[J]. China Cucurbits and Vegetables,2024,37(2) :66−73. (in Chinese)
[8] 张丽芳,曹阳,张兰. 解淀粉芽孢杆菌B235对花生根腐病的防治效果研究[J]. 花生学报,2024,53(1) :72−78. ZHANG L F,CAO Y,ZHANG L. Study on the control effect of Bacillus amyloliquefaciens B235 on flower root rot[J]. Journal of Peanut Science,2024 ,53(1) :72−78.
[9] 李莹,宋新颖,何康,等. 贝莱斯芽孢杆菌ZHX-7的分离鉴定及抑菌促生效果[J]. 生物技术通报,2023,39(12) :229−236. LI Y,SONG X Y,HE K,et al. Isolation and identification of Bacillus velezensis ZHX-7 and its antibacterial and growth-promoting effects[J]. Biotechnology Bulletin,2023,39(12) :229−236. (in Chinese)
[10] 宋波,文国琴,何瑶. 芒果病原菌的分离鉴定及生防芽孢杆菌的筛选[J]. 西南农业学报,2024,37(12) :2757−2764. SONG B,WEN G Q,HE Y. Isolation and identification of mango pathogen and screening of its antagonistic Bacillus[J]. Southwest China Journal of Agricultural Sciences,2024,37(12) :2757−2764. (in Chinese)
[11] 邓云,田大刚,苏妍,等. 多粘类芽孢杆菌NPDY05-8对玉米茎基腐病的防治效果及对土壤微生物的影响[J]. 福建农业学报,2023,38(12) :1445−1452. DENG Y,SU Y,TIAN D G,et al. Control on Maize Stalk Rot and Effects on Soil Microbes of Paenibacillus polymyxa[J]. Fujian Journal of Agricultural Sciences,2023,38(12) :1445−1452.
[12] JOHNY L C,SURESH P V. sequencing and strain characterization of a novel marine Bacillus velezensis FTL7 with a potential broad inhibitory spectrum against food borne pathogens[J]. World Journal of Microbiology and Biotechnology,2022,38(9) :164.
[13] KRZYWINSKI M,SCHEIN J,BIROL İ,et al. Circos:An information aesthetic for comparative genomics[J]. Genome Research,2009,19(9) :1639−1645. DOI: 10.1101/gr.092759.109
[14] BLAND C,RAMSEY T L,SABREE F,et al. CRISPR recognition tool (CRT) :A tool for automatic detection of clustered regularly interspaced palindromic repeats[J]. BMC Bioinformatics,2007,8(1) :209. DOI: 10.1186/1471-2105-8-209
[15] POWELL S,FORSLUND K,SZKLARCZYK D,et al. eggNOG v4.0:Nested orthology inference across 3686 organisms[J]. Nucleic Acids Research,2014,42(D1) :D231−D239. DOI: 10.1093/nar/gkt1253
[16] CANTAREL B L,COUTINHO P M,RANCUREL C,et al. The carbohydrate-active EnZymes database (CAZy) :An expert resource for glycogenomics[J]. Nucleic Acids Research,2009,37(suppl_1) :D233−D238.
[17] JIA B F,RAPHENYA A R,ALCOCK B,et al. CARD 2017:Expansion and model-centric curation of the comprehensive antibiotic resistance database[J]. Nucleic Acids Research,2017,45(D1) :D566−D573. DOI: 10.1093/nar/gkw1004
[18] ZHOU J L,FENG Z L,LIU S C,et al. CGTase,a novel antimicrobial protein from Bacillus cereus YUPP-10,suppresses Verticillium dahliae and mediates plant defence responses[J]. Molecular Plant Pathology,2021,22(1) :130−144. DOI: 10.1111/mpp.13014
[19] 舒霞. 贝莱斯芽孢杆菌SQR9 VII型分泌系统分泌蛋白YukE的根际益生功能增效研究[D]. 南京:南京农业大学,2021. SHU X. Synergism of Protein YukE Secreted by Type VII Secretion System of Bacillus Velezensis SQR9 in Plant Growth Promotion[D]. Nanjing:Nanjing Agricultural University,2021. (in Chinese)
[20] KAI B,SIMON S,KATHARINA S,et al. antiSMASH 5.0:updates to the secondary metabolite genome mining pipeline[J]. Nucleic Acids Research (W1) :W1.
[21] SHEN L J,ZANG X L,SUN K,et al. Complete genome sequencing of Bacillus sp. TK-2,analysis of its cold evolution adaptability[J]. Scientific Reports,2021,11:4836. DOI: 10.1038/s41598-021-84286-7
[22] LI L F,WU Q X,WU H,et al. Complete genome sequence of Streptomyces sp. HP-A2021,a promising bacterium for natural product discovery[J]. Biochemical Genetics,2023,61(5) :2042−2055. DOI: 10.1007/s10528-023-10350-8
[23] DUAN Z D,LIAO L,CHEN B. Complete genome analysis reveals secondary metabolite biosynthetic capabilities of Streptomyces sp. R527F isolated from the Arctic Ocean[J]. Marine Genomics,2022,63:100949. DOI: 10.1016/j.margen.2022.100949
[24] LÓPEZ-MEZA J E,OCHOA-ZARZOSA A,BARBOZA-CORONA J E,et al. Antimicrobial peptides:Current and potential applications in biomedical therapies[J]. BioMed Research International,2015,2015:367243.
[25] 王菲,罗恩杰. CRISPR及其在原核生物防御系统中的作用[J]. 热带医学杂志,2008,8(10) :1104−1106. DOI: 10.3969/j.issn.1672-3619.2008.10.034 WANG F,LUO E J. CRISPR and its function in prokaryotic defense system[J]. Journal of Tropical Medicine,2008,8(10) :1104−1106. (in Chinese) DOI: 10.3969/j.issn.1672-3619.2008.10.034
[26] 曾珍,胡莹. CRISPR/Cas系统在抗菌治疗领域的研究进展[J]. 中国医药生物技术,2021,16(1) :64−67. ZENG Z,HU Y. Research progress of CRISPR/Cas system in the field of antibacterial therapy[J]. Chinese Medicinal Biotechnology,2021,16(1) :64−67. (in Chinese)





 下载:
下载: