Re-identification of Tomato AP2/ERF Transcription Factors and Screening of Genes Related to Stress Resistance
-
摘要:目的
本研究深入理解番茄AP2/ERF转录因子的组学特征及生物胁迫响应,促进其在番茄抗病育种中的应用。
方法利用最新公共数据进行AP2/ERF鉴定,结合番茄自交系在青枯菌侵染下的转录测序数据,挖掘青枯病抗性相关AP2/ERF。
结果173个番茄AP2/ERF转录因子家族成员被鉴定,主要成簇分布在染色体端部,50.3%的AP2/ERF具有共线性,尤其是3和8号染色体。基于系统进化关系,AP2亚家族包含22个(12.7%)成员,主要分为两类;ERF亚家族包含93个(53.8%)成员,可进一步分为六类;DREB亚家族包含49个(28.3%)成员,可进一步分为两类;RAV亚家族仅有3个成员。71对AP2/ERF同源基因的Ka/Ks值均小于1。26.8%的AP2/ERF主要在根中表达,几种生物胁迫下,ERF_4-6和DREB_2基因转录表达水平较高,尤其是Solyc03g005500.1(ERF_6)和Solyc08g078170.1(ERF_5)。青枯菌侵染下,在抗、感材料间筛选到10个差异表达AP2/ERF(分属AP2和ERF),其中Solyc06g068570.4(AP2)为正调控基因,其他9个为负调控基因。50.9%的AP2/ERF形成193对互作关系,尤其是Solyc01g096860.3(AP2_2)与Solyc08g078190.2(ERF_5),而Solyc06g068570.4(AP2)与Solyc03g093560.1(ERF_5)正负互作在番茄青枯病响应中可能发挥重要作用。
结论综合基因组分析和转录表达鉴定了番茄AP2/ERF转录因子,获得若干青枯病响应基因,丰富了番茄抗病育种基础理论和基因资源。
-
关键词:
- 番茄 /
- AP2/ERF转录因子 /
- 基因组特征 /
- 生物胁迫 /
- 转录表达
Abstract:ObjectiveTo promote the germplasm application in tomato resistance breeding by gaining better insights on the genomic characteristics and biotic stress response of AP2/ERF transcription factors (TFs).
MethodAP2/ERF TFs were identified building on the recent release of genome data and genes related to bacterial wilt resistance were excavated, combined with the transcriptome sequencing of tomato inbred lines with Ralstonia solanacearum infection (RsI).
ResultA total of 173 AP2/ERF TFs were identified in tomato, which were mainly distributed in clusters at the ends of chromosomes, and 50.3% of them had collinearity, especially on chromosomes 3 and 8. Based on phylogenetic relationship, the AP2 subfamily contained 22 (12.7%) genes, which could be divided into two main categories; the ERF subfamily contained 93 (53.8%) genes and were further divided into six categories; the DREB subfamily contained 49 (28.3%) genes and were divided into two categories; and the RAV subfamily had only three genes. The Ka/Ks values of 71 pairs of tomato AP2/ERF homologs were all less than 1. 26.8% of AP2/ERF TFs were mainly expressed in roots, and the expression levels of genes in ERF_4-6 and DREB_2 categories were higher under several biotic stresses, especially Solyc03g005500.1 (ERF_6) and Solyc08g078170.1 (ERF_5). Ten differentially expressed AP2/ERF TFs (belonging to AP2 and ERF) were screened between resistant and susceptible tomato lines with RsI, of which Solyc06g068570.4 (AP2) was a positively regulated gene and the other nine were negatively regulated genes. 50.9% of tomato AP2/ERF TFs formed 193 pairs of interactions, of which the interaction between Solyc01g096860.3 (AP2_2) and Solyc08g078190.2 (ERF_5) was the most reliable, whereas the positive and negative interaction between Solyc06g068570.4 (AP2) and Solyc03g093560.1 (ERF_5) may play an important role in tomato response to bacterial wilt.
ConclusionTomato AP2/ERF TFs were comprehensively identified using genomic information and transcriptional expression, and several bacterial wilt response genes were obtained, which enriched the basic theory and genetic resources for tomato resistance breeding.
-
0. 引言
【研究意义】随着全球气温升高和种植单一化,作物病害日趋严重,选育抗病品种是绿色生产和稳产的有效途径。各种逆境胁迫下基因表达的改变主要发生在转录调控水平,转录因子(Transcription factor,TF)通过特异性识别和结合启动子顺式作用元件(Cis-acting element),激活或抑制靶基因转录活性,决定其时空表达。作为植物中普遍存在的转录因子超家族,AP2/ERF(APETALA2/ethylene-responsive factor)通过与启动子GCC-box(AGCCGCC)和DRE/CRT(Dehydration-responsive element/C-repeat,A/GCCGAC)顺式作用元件结合,广泛参与植物的生长发育、次生代谢调控及生物与非生物胁迫响应[1],深入研究AP2/ERF转录因子,对促进其在作物遗传育种中的应用具有重要意义。【前人研究进展】AP2/ERF至少含有一个由60~70个氨基酸组成的AP2结构域,N端的YRG元件呈亲水性和碱性,可直接与DNA结合;C端的RAYD元件具有两亲性,可与蛋白互作[2]。根据AP2结构域的数量和特征,AP2/ERF可划分为AP2、ERF、DREB(DRE binding protein)、RAV(Related to AB13/VP)和其他(Soloist)5个亚家族[3]。目前AP2/ERF已在拟南芥(Arabidopsis thaliana)[4−5]、水稻(Oryza sativa)[5−6]、玉米(Zea mays)[7]、烟草(Nicotiana tabacum)[8]、马铃薯(Solanum tuberosum)[9]、番茄(Solanum lycopersicum)[10−12]、辣椒(Capsicum annuum)[13]、茄子(Solanum melongena)[14]、黄瓜(Cucumis sativus)[15]、萝卜(Raphanus sativus)[16]、油菜(Brassica napus)[17]、葡萄(Vitis vinfera L.)[18]等多种植物中被分离鉴定。研究表明番茄和烟草中过表达TSRF1,青枯病抗性增强,且TSRF1与GCC-box结合受脱落酸(Abscisic acid,ABA)调控[19−20]。番茄中过表达AtCBF1,通过SIERF5/SIRAV2在AtCBF1与PR5之间行使功能,提高青枯病抗性[21]。烟草中过表达GmERF3,能诱导病程相关(Pathogenesis-related,PR)基因的表达,提高植株对青枯病等的抗性[22]。拟南芥中过表达HvRAF,PDF1.2、JR3、PR1/5、KIN2和GSH1等抗逆相关基因上调,青枯病抗性增强[23]。大部分ERF属转录激活子,但有些则含有一个抑制因子结构域EAR(ERF-associated amphiphilic repression)或存在保守的磷酸化位点[5,24]。过表达番茄去除EAR的SlERF3能激活PR1/2/5基因表达,提高青枯病抗性和耐盐性[25]。但BrERF11含有完整EAR,却能使PR1a/1b/3/等PR基因表达显著提高,青枯病抗性增强[26]。【本研究切入点】在番茄基因组测序完成的基础上,前人已对AP2/ERF转录因子超家族进行了鉴定[10−12]。但随着番茄基因组数据的不断更新和完善,更全面地鉴定和分析番茄AP2/ERF,丰富其成员信息,挖掘未知基因功能成为可能。【拟解决的关键问题】利用新的番茄基因组公共数据和注释信息,以拟南芥为参照,比较鉴定番茄AP2/ERF转录因子家族成员,深入理解其组学特征和系统进化关系。在此基础上,通过挖掘抗、感青枯病番茄自交系转录测序数据,鉴定青枯病抗性相关AP2/ERF,为番茄抗性遗传育种中品种改良提供理论基础和基因资源。
1. 材料与方法
1.1 材料与处理
抗、感青枯病番茄为温州市农业科学研究院蔬菜科学研究所从长期搜集到的种质中选育的高代自交系。青枯菌(Ralstonia solanacearum)菌株从田间发病番茄植株上分离。番茄幼苗长至4~6片真叶时伤根接种青枯菌,48h后时取样地上部,设置3次生物学重复。
1.2 番茄AP2/ERF基因家族鉴定
番茄基因组序列及注释信息(SL4.0 ITAG4.0)下载自茄科基因组数据库SGN(https://solgenomics.net/)。在InterPro(https://www.ebi.ac.uk/interpro/)中下载AP2隐马尔科夫模型(Hidden Markov Model, HMM)PF00847,并以此和拟南芥AP2保守结构域作为种子序列进行Blastp(E-value < 1),获得候选基因。然后通过SMART(http://smart.embl-heidelberg.de/smart/batch.pl)和CDD(https://www.ncbi.nlm.nih.gov/cdd)逐一鉴定AP2结构域,参考植物转录因子数据库PlantTFDB v5.0(http://planttfdb.gao-lab.org/index.php),将不含目的结构域的候选基因排除。
利用在线程序ExPASy ProtParam(https://web.expasy.org/protparam)对AP2/ERF进行蛋白理化参数预测。用WoLF PSORT(https://wolfpsort.hgc.jp/)进行蛋白亚细胞定位预测。用NetPhos 3.1(https://services.healthtech.dtu.dk/services/NetPhos-3.1/)预测丝氨酸(Serine,S)、苏氨酸(Threonine,T)和酪氨酸(Tyrosine,Y)磷酸化修饰位点。
1.3 番茄AP2/ERF的染色体定位
根据番茄基因组注释信息,用TBtools软件[27]绘制AP2/ERF转录因子染色体分布图。随后进行基因簇和串联重复分析[28],划分基因簇的原则为:相邻AP2/ERF基因间距小于200 kb且其他基因不得多于8个;判断串联重复的原则为:相邻AP2/ERF基因距小于100 kb且序列相似度高于70%。先用TBtools中的MC ScanX程序进行基因共线性分析,再用Circos进行种内共线性可视化。基于NG算法计算基因间的选择压(Ka/Ks)。
1.4 番茄AP2/ERF基因系统聚类
利用TBtools的Muscle Wrapper工具对AP2/ERF氨基酸序列进行比对,用IQ-Tree基于最大似然法(Maximum likelihood, ML)构建系统发生树[27],然后用FigTree1.4.2软件对树进行可视化。
1.5 番茄AP2/ERF转录表达分析
从番茄功能基因组数据库TFGD(http://ted.bti.cornell.edu/cgi-bin/TFGD/digital/home.cgi)下载番茄RNA-seq测序数据(D004和D007),去除表达量较低(RPKM<1)的AP2/ERF,并对其余数据进行log2标准化,利用TBtools绘制基因的表达热图[27]。
使用植物Trizol总RNA提取试剂盒提取RNA,检测后送百迈客生物科技有限公司进行转录组测序。以差异倍数(Fold change,FC)≥ 2且错误发现率(False discovery rate,FDR)< 0.01作为标准,筛选差异表达(Differentially expressed,DE)AP2/ERF转录因子,并对其进行本体库(Gene ontology consortium,GO)和京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)注释。
1.6 番茄AP2/ERF互作网络构建
利用STRING11.5在线工具(https://cn.string-db.org/cgi/input.pl?sessionId=N1dkcvbzuhF4&input_page_show_search=on)构建番茄AP2/ERF转录因子互作网络,然后导入Cytoscape软件进行可视化。
2. 结果与分析
2.1 番茄AP2/ERF的分类与特征
基于AP2/ERF保守氨基酸序列,E值1为阈值,从番茄基因组中鉴定到173个AP2/ERF转录因子,其中27个基因具有2个及以上AP2保守结构域,3个基因同时具有1个AP2和1个B3保守结构域,其他基因仅具有1个AP2保守结构域。这些基因编码氨基酸数为56-908,中位数242;蛋白分子量为6.52-102.47 kD,中位数27.15 kD;理论等电点为4.40-11.31,中位数6.51;不稳定系数为21.51-85.07,中位数52.31;总平均亲水性为−1.212-(−0.303),中位数−0.716;磷酸化位点数为4-111,中位数31(图1A)。85%的基因定位于细胞核,其次是叶绿体(7%)和细胞质(5%)(图1B)。综上,编码蛋白主要以阴离子形式存在,整体呈不稳定性和亲水性,定位于细胞核,磷酸化修饰以丝氨酸为主。
2.2 番茄AP2/ERF的染色体分布
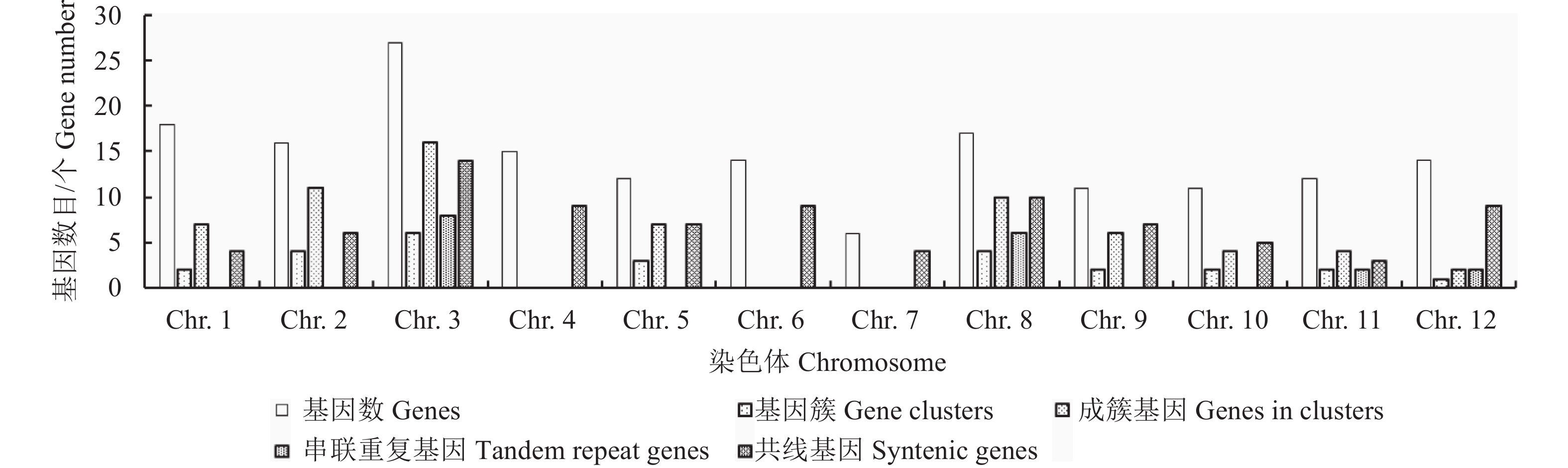
173个番茄AP2/ERF转录因子散布于各染色体,形成26个基因簇(67个基因)和12对串联重复(18个基因)。其中,3号染色体基因最多(27个),7号染色体基因最少(6个),其他染色体为11-18个,各染色体平均14个。串联重复AP2/ERF集中于3和8号染色体,占各自染色体总AP2/ERF的比例分别达到29.6%和35.3%;而基因簇主要形成于2、3、5、8和9号染色体,基因比例均超过50%(图2)。同时,发现87个(50.3%)AP2/ERF具有种内共线性,形成60对共线性基因。除1、2、10和11号染色体外,其他染色体共线基因比例均超过50%。3和8号染色体共线基因分别为14个和10个,比例分别达到51.9%和58.8%(图2)。Solyc03g026270.3、Solyc03g093540.1、Solyc08g007820.1、Solyc08g078170.1、Solyc08g078180.1和Solyc08g078410.2为串联重复和共线性所共有,Solyc08g078170.1与Solyc08g078180.1间虽形成串联重复但不具共线性。
2.3 番茄AP2/ERF系统进化关系
参考拟南芥AP2/ERF转录因子分类,结合保守基序分析,173个番茄AP2/ERF主要划分为11类(图3)。AP2亚家族包含22个成员,占比12.7%,主要分为AP2_1和AP2_2两类,基因数目分别为8和13,Solyc06g068570.4独立于其他基因;ERF亚家族包含93个成员,占比53.8%,可进一步分为ERF_1-6六类,基因数目分别为16、5、8、12、28和24;DREB亚家族包含49个成员,占比28.3%,可进一步分为DREB_1和DREB_2两类,基因数目分别为19和30;RAV亚家族仅有3个成员。Solyc01g008890.4具有2个AP2保守结构域,Solyc02g030210.4保守氨基酸序列更接近AP2,但两者在聚类上分属ERF_3和ERF_1类。6个基因并未明确聚类,但Solyc04g009450.1、Solyc08g081650.3和Solyc08g081670.3具有2个以上AP2保守结构域,Solyc03g119580.1、Solyc04g007170.3和Solyc05g009250.1保守氨基酸序列更接近ERF。
基于基因重复、共线性和系统进化关系,分析99个(57.2%)番茄AP2/ERF间的71对同源基因。其中,AP2_1和AP2_2同源基因数目分别为8和5(6和2对),占比100%和38.5%;ERF各类同源基因数目分别为5、3、3、5、16和18(4、3、3、4、12和10对),占比31.3%、60%、37.5%、41.7%、57.1%和75%;DREB各类同源基因数目分别为9和22(5和19对),占比47.4%和73.3%;1对RAV类同源基因,占比66.7%;另有1对同源基因未聚类。整体来看,AP2_1、ERF_2、ERF_6和DREB_2类同源基因比例较高,ERF_1和ERF_3比例较低。这些同源基因对Ka/Ks为0.077(Solyc03g095973.1 vs. Solyc03g095977.1,DREB_1)−0.586(Solyc11g045690.3 vs. Solyc11g045680.3,ERF_6),均值0.226,均小于1。
2.4 番茄AP2/ERF基因转录表达
基于已有转录组数据,番茄中分别有26.8%和14.9%的AP2/ERF转录因子主要在根和芽中表达(图4A)。细菌鞭毛蛋白侵染6 h,ERF_6类中的Solyc03g005500.1和Solyc09g089910.1,ERF_5类中的Solyc03g005520.1、Solyc02g077370.1和Solyc08g078170.1具有较高的基因表达水平(图4B3)。不同丁香假单胞菌菌株侵染6 h,ERF_6类中的Solyc03g005500.1和Solyc03g005510.2,ERF_5类中的Solyc08g078170.1、Solyc09g089930.3、Solyc03g005520.1和Solyc09g066360.1,DREB_2类中的Solyc12g056430.1具有较高的基因表达水平(图4B2和4B3)。荧光假单胞菌或恶臭假单胞菌侵染6 h,ERF_6类中的Solyc03g005500.1和ERF_5类中的Solyc08g078170.1具有较高的基因表达水平(图4B3)。根癌农杆菌侵染6 h,ERF_4类中的Solyc10g078610.1、Solyc03g116610.3和Solyc03g117130.3,ERF_6类中的Solyc05g013540.1具有较高的基因表达水平(图4B3)。整体来看,ERF_4-6和DREB_2类基因转录表达水平较高,尤其是Solyc03g005500.1(ERF_6)和Solyc08g078170.1(ERF_5),在4种病菌侵染下均有较高的表达水平,但ERF_6类内不同分支基因表达水平差异明显,而AP2类基因转录表达水平较低。
![]() 图 4 番茄AP2/ERF在不同组织部位(A)和病菌侵染下(B)的相对表达A. 栽培番茄Heinz1706芽、花、叶、根和不同发育阶段果实(1-3 cm、绿熟期、破色期和破色后10 d)中基因的表达。B. flgII-28 6 h: 细菌鞭毛蛋白侵染. 不同丁香假单胞菌菌株侵染.荧光假单胞菌侵染.恶臭假单胞菌侵染.根癌农杆菌侵染.Figure 4. Expression profile of AP2/ERF TFs in different tissues (A) and with pathogen infection (B) in tomatoA.The expression of genes in the buds, flowers, leaves, roots, and fruits at different developmental stages (1-3 cm, green maturity stage, color break stage, and 10 days after color break) of cultivated tomato Heinz1706. B.flgII-28 6 h: Bacterial flagellar protein infection. Infection of different strains of P. syringae. Infection by P. fluorescens. Infection by odorous pseudomonas. Infection of A. tumefaciens.
图 4 番茄AP2/ERF在不同组织部位(A)和病菌侵染下(B)的相对表达A. 栽培番茄Heinz1706芽、花、叶、根和不同发育阶段果实(1-3 cm、绿熟期、破色期和破色后10 d)中基因的表达。B. flgII-28 6 h: 细菌鞭毛蛋白侵染. 不同丁香假单胞菌菌株侵染.荧光假单胞菌侵染.恶臭假单胞菌侵染.根癌农杆菌侵染.Figure 4. Expression profile of AP2/ERF TFs in different tissues (A) and with pathogen infection (B) in tomatoA.The expression of genes in the buds, flowers, leaves, roots, and fruits at different developmental stages (1-3 cm, green maturity stage, color break stage, and 10 days after color break) of cultivated tomato Heinz1706. B.flgII-28 6 h: Bacterial flagellar protein infection. Infection of different strains of P. syringae. Infection by P. fluorescens. Infection by odorous pseudomonas. Infection of A. tumefaciens.分析青枯菌侵染前后抗、感番茄自交系的转录测序数据,筛选到10个差异表达AP2/ERF转录因子,其中ERF_3、ERF_5和AP2类基因各3个,ERF_6类基因1个。Solyc01g095500.3、Solyc03g007460.3、Solyc04g077490.3、Solyc06g051840.1、Solyc10g009110.1和Solyc11g010710.2在抗病材料中下调表达,但Solyc04g077490.3表达量变化仅达到显著水平(P < 0.05),Solyc11g010710.2表达量变化不显著,Solyc06g051840.1表达量在感病材料中显著(P < 0.05)下降但幅度较小;Solyc03g093560.1、Solyc03g093610.1和Solyc05g052040.1在感病材料中上调表达,但Solyc03g093560.1和Solyc05g052040.1表达量变化不显著;Solyc06g068570.4在感病材料中下调表达,但表达量变化仅达到显著水平(P < 0.05),其他表达各情况均未明显变化(图5)。这些DE AP2/ERF除在DNA结合条目(GO:0003677和GO:0003700)富集外,还涉及转录调控(GO:0006355)、生长素响应(GO:0009733)、乙烯合成(GO:0010364)、防御反应(GO:0006952)、果实成熟(GO:0009835)、乙烯信号途径调控(GO:0010104)和多细胞生物发育(GO:0007275)等生物过程。
![]() 图 5 番茄青枯菌侵染下差异表达AP2/ERFR和S分别代表抗、感番茄自交系,Mock和RsI分别表示对照和青枯菌侵染。*、**和***分别表示FDR<0.05、FDR<0.01和FDR<0.001,不同大、小写字母分别表示P<0.01和P<0.05水平上的差异显著性。Figure 5. Transcriptionally DE AP2/ERF TFs with RsI in tomatoR and S represent resistant and susceptible tomato inbred lines, Mock and RsI represent a control and R. solanacearum infection, respectively. *, **, and *** indicate FDR<0.05, FDR<0.01, and FDR<0.001, respectively. Different uppercase and lowercase letters indicate significant differences at P<0.01 and P<0.05 levels, respectively.
图 5 番茄青枯菌侵染下差异表达AP2/ERFR和S分别代表抗、感番茄自交系,Mock和RsI分别表示对照和青枯菌侵染。*、**和***分别表示FDR<0.05、FDR<0.01和FDR<0.001,不同大、小写字母分别表示P<0.01和P<0.05水平上的差异显著性。Figure 5. Transcriptionally DE AP2/ERF TFs with RsI in tomatoR and S represent resistant and susceptible tomato inbred lines, Mock and RsI represent a control and R. solanacearum infection, respectively. *, **, and *** indicate FDR<0.05, FDR<0.01, and FDR<0.001, respectively. Different uppercase and lowercase letters indicate significant differences at P<0.01 and P<0.05 levels, respectively.2.5 番茄AP2/ERF基因互作关系
88个番茄AP2/ERF转录因子间存在193对互作关系,其中Solyc04g049800.3(AP2_1)有31个互作因子,Solyc10g084340.2(AP2_1)、Solyc09g007260.4(AP2_1)、Solyc03g044300.3(AP2_1)、Solyc12g038440.1(ERF_6)、Solyc01g096860.3(AP2_2)和Solyc02g064960.3(AP2_1)互作因子均在10个以上,各转录因子互作基因平均数为4.4(图6)。Solyc01g096860.3(AP2_2)与Solyc08g078190.2(ERF_5)互作可信度最大(Score>0.6),另外27对互作关系可信度也较大(Score>0.5)。结合青枯菌诱导下的番茄转录组数据分析,Solyc06g068570.4(AP2)与9个基因存在互作,且在感病材料中下调表达,Solyc03g093560.1(ERF_5)和Solyc06g051840.1(ERF_3)分别与2个基因存在互作,且前者在感病材料中上调表达后者在抗病材料中下调表达。其中,Solyc06g068570.4(AP2)与Solyc03g093560.1(ERF_5)互作是一对重要调控关系。
3. 讨论
在作物抗病育种中,抑制或增强一个关键转录因子,从而调控多个相关基因表达,是提高作物抗性的有效途径。重鉴定到173个番茄AP2/ERF转录因子,丰富了其成员信息,筛选到若干青枯病抗性可能调控基因,为番茄抗性AP2/ERF挖掘和利用奠定了基础。
3.1 番茄AP2/ERF基因家族的基本特征
不同物种所含AP2/ERF转录因子家族成员不同。Nakano等[5]报道拟南芥含147个AP2/ERF家族成员,其中AP2亚家族基因18个、ERF/DREB亚家族基因122个、RAV亚家族基因6个。Rashid等[6]对水稻进行全基因组分析,鉴定出170个AP2/ERF家族成员,其中AP2亚家族基因23个、ERF/DREB亚家族基因141个、RAV亚家族基因4个。鉴于拟南芥和水稻基因组大小差异,推测AP2/ERF数量与蛋白组大小正相关,而与基因组大小没有必然联系。ERF和DREB亚家族成员主要区别在于AP2结构域第14和19位氨基酸残基不同,ERF分别是丙氨酸(Alanine, A)和天冬氨酸(Aspartic acid, D),而DREB分别是缬氨酸(Valine, V)和谷氨酸(Glutamic acid, E)[3, 29]。本研究鉴定到的AP2/ERF转录因子比徐志璇等 [30]的研究结果少3个,可能与不同数据版本测序质量、注释完整性和选用标准有关,如后者AP2亚家族成员更多,ERF/DREB总数相近但ERF多DREB少。15个原有ERF/DREB未被鉴定到,但其中12个在新的番茄基因组中未被注释到,Solyc03g123520.3不含AP2结构域,Solyc01g090320.4和Solyc08g007840.3因注释不完整或阈值所限无法确定。
AP2/ERF编码蛋白主要以阴离子形式存在,整体呈不稳定性和亲水性,定位于细胞核,AP2和部分ERF/DREB亚家族成员具有较多磷酸化修饰位点,且以丝氨酸为主(图1)。这些特性有利于基因信号传导、代谢调控和应激响应,从而促进植物的生长发育和逆境适应。85%的AP2/ERF转录因子定位于细胞核,这符合其调控下游基因表达的特性,但叶绿体定位基因的功能有待进一步研究。磷酸化修饰可增强AP2/ERF与顺式作用元件的结合能力,调控转录活性和蛋白质稳定性,如番茄Ser/Thr蛋白激酶Pto磷酸化Pti4 [5]。不同AP2/ERF磷酸化修饰类型和位点数目存在差异,可能与植物逆境胁迫响应中的信号传导有关[31]。
3.2 番茄AP2/ERF基因的系统进化关系
超过50%的番茄AP2/ERF转录因子具有共线性关系,尤其是3和8号染色体(图2),表明其进化过程的保守性,不同染色体上的共线基因可能源自片段重复。并非所有串联重复基因具有共线性,可能在进化过程中发生了结构变异。基于序列比对和系统进化关系,番茄AP2/ERF转录因子主要分为AP2_1-2、ERF_1-6、DREB_1-2和RAV共11类(图3)。整合以上分析,推测近60%的番茄AP2/ERF间71对旁系同源基因可能具有重要生物学功能。其中,AP2_1、ERF_2、ERF_6、DREB_2和RAV类同源基因比例均超过60%。这些同源基因对Ka/Ks值均小于1,表明其在进化过程中以纯化选择为主。
Solyc01g008890.4、Solyc02g030210.4、Solyc04g009450.1、Solyc08g081650.3和Solyc08g081670.3在不同研究中AP2和ERF归属并不一致。Solyc03g119580.1、Solyc04g007170.3和Solyc05g009250.1尽管在序列上更接近ERF,但分支独立。而Solyc06g068570.4在AP2亚家族中的进化地位,Solyc02g088310.1属于ERF还是DREB,也需进一步鉴定。本研究番茄与已有拟南芥、水稻、烟草、辣椒、茄子、马铃薯和番茄[32]AP2/ERF转录因子各亚家族成员数目相关系数分别为0.982、0.977、0.915、0.929、0.885、0.997和0.994,表明AP2/ERF在不同物种进化过程中虽存在扩张或收缩,但亚家族成员具有相对保守性。绝大多数AP2/ERF转录因子保守结构域均含YRG和RAYD序列,但也有少数基因发生了变异或丢失,表明转录因子结合活性及功能执行的进化选择。
3.3 番茄AP2/ERF基因青枯菌胁迫应答
番茄中超过25%的AP2/ERF转录因子在根中偏好性表达,说明其主要参与根部代谢调控。ERF_4-6和DREB_2类基因对几种生物胁迫反应强烈,尤其是Solyc03g005500.1(ERF_6)和Solyc08g078170.1(ERF_5)(图4)。研究表明,AtCBF1、BrERF11/SlERF3、CaERF3、CaERF5/SlERF5、ERF098、SlRAV2和TSRF1均能介导植物对青枯病的抗性,且以正调控为主[19−21, 25−26, 33−35]。这7个AP2/ERF转录因子在番茄中分别最优匹配于Solyc03g026280.3(DREB_2)、Solyc10g009110.1(ERF_6)、Solyc07g049490.3(ERF_6)、Solyc03g093560.1(ERF_5)、Solyc03g005500.1(ERF_6)、Solyc05g009790.1(RAV)和Solyc09g089930.3(ERF_5),可能参与番茄青枯病抗性响应。青枯菌诱导差异表达AP2/ERF转录因子中,ERF_5/6类基因占40%,且均为负调控(图5)。这种生物胁迫应答差异性,可能与各类AP2/ERF结合元件、活性大小和参与的激素信号通路不同有关[11]。SlERF3(Solyc10g009110.1)和SlERF5(Solyc03g093560.1)在不同研究中对青枯病的应答模式不同[21, 25],说明ERF同源基因功能机制的复杂性,需进一步验证。互作分析推测Solyc06g068570.4(AP2)作为转录调控网络重要节点,通过与Solyc03g093560.1(ERF_5)正负互作(图6),在番茄应答青枯病过程中起关键作用,靶向他们的novel_miR_54和sly-miR10534也可能发挥重要调控作用[36]。
AP2/ERF转录因子通过结合PR基因启动子中的GCC-box激活下游防御基因表达和H2O2积累,从而抵御各种生物胁迫。如拟南芥AT4G13040,作为Soloist亚家族的“孤儿”基因,通过在水杨酸(Salicylic acid, SA)信号通路PAD4下游发挥作用,促进SA积累,增强细菌性病害抗性[37]。该基因在番茄中最优匹配于Solyc09g059510.3,聚于同一分支,属于DREB_1类(图3)。
4. 结论
研究重鉴定到173个番茄AP2/ERF转录因子,包括AP2、ERF、DREB和RAV四个亚家族。这些AP2/ERF主要成簇分布在染色体端部,超过50%具有共线性,尤其是3和8号染色体,说明其进化具有保守性。重复是基因簇的重要成因,也是基因进化的重要动力。系统聚类可将AP2/ERF划分为11类,71对重要同源基因的Ka/Ks值均小于1,表明其进化过程以纯化选择为主。几种生物胁迫下,ERF_4-6和DREB_2基因转录表达水平较高,尤其是Solyc03g005500.1(ERF_6)和Solyc08g078170.1(ERF_5)。青枯菌诱导下,差异表达基因Solyc06g068570.4(AP2)为正调控,其他9个为负调控。超过50%的AP2/ERF存在互作关系,尤其是Solyc01g096860.3(AP2_2)与Solyc08g078190.2(ERF_5)。Solyc06g068570.4(AP2)作为转录调控网络节点,通过与Solyc03g093560.1(ERF_5)互作,可能在番茄青枯病响应中发挥重要作用。总之,重鉴定AP2/ERF转录因子,探索家族成员之间如何互作和参与激素信号传导进而介导下游基因表达,解析基因调控网络和作用机制,有助于AP2/ERF的挖掘和在作物广谱抗性中的应用。
-
图 4 番茄AP2/ERF在不同组织部位(A)和病菌侵染下(B)的相对表达
A. 栽培番茄Heinz1706芽、花、叶、根和不同发育阶段果实(1-3 cm、绿熟期、破色期和破色后10 d)中基因的表达。B. flgII-28 6 h: 细菌鞭毛蛋白侵染. 不同丁香假单胞菌菌株侵染.荧光假单胞菌侵染.恶臭假单胞菌侵染.根癌农杆菌侵染.
Figure 4. Expression profile of AP2/ERF TFs in different tissues (A) and with pathogen infection (B) in tomato
A.The expression of genes in the buds, flowers, leaves, roots, and fruits at different developmental stages (1-3 cm, green maturity stage, color break stage, and 10 days after color break) of cultivated tomato Heinz1706. B.flgII-28 6 h: Bacterial flagellar protein infection. Infection of different strains of P. syringae. Infection by P. fluorescens. Infection by odorous pseudomonas. Infection of A. tumefaciens.
图 5 番茄青枯菌侵染下差异表达AP2/ERF
R和S分别代表抗、感番茄自交系,Mock和RsI分别表示对照和青枯菌侵染。*、**和***分别表示FDR<0.05、FDR<0.01和FDR<0.001,不同大、小写字母分别表示P<0.01和P<0.05水平上的差异显著性。
Figure 5. Transcriptionally DE AP2/ERF TFs with RsI in tomato
R and S represent resistant and susceptible tomato inbred lines, Mock and RsI represent a control and R. solanacearum infection, respectively. *, **, and *** indicate FDR<0.05, FDR<0.01, and FDR<0.001, respectively. Different uppercase and lowercase letters indicate significant differences at P<0.01 and P<0.05 levels, respectively.
-
[1] FENG K, HOU X L, XING G M, et al. Advances in AP2/ERF super-family transcription factors in plant [J]. Critical Reviews in Biotechnology, 2020, 40(6): 750−776. DOI: 10.1080/07388551.2020.1768509
[2] OKAMURO J K, CASTER B, VILLARROEL R, et al. The AP2 domain of APETALA2 defines a large new family of DNA binding proteins in Arabidopsis [J]. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(13): 7076−7081.
[3] SAKUMA Y, LIU Q, DUBOUZET J G, et al. DNA-binding specificity of the ERF/AP2 domain of Arabidopsis DREBs, transcription factors involved in dehydration- and cold-inducible gene expression [J]. Biochemical and Biophysical Research Communications, 2002, 290(3): 998−1009. DOI: 10.1006/bbrc.2001.6299
[4] RIECHMANN J L, HEARD J, MARTIN G, et al. Arabidopsis transcription factors: Genome-wide comparative analysis among eukaryotes [J]. Science, 2000, 290(5499): 2105−2110. DOI: 10.1126/science.290.5499.2105
[5] NAKANO T, SUZUKI K, FUJIMURA T, et al. Genome-wide analysis of the ERF gene family in Arabidopsis and rice [J]. Plant Physiology, 2006, 140(2): 411−432. DOI: 10.1104/pp.105.073783
[6] RASHID M, HE G Y, YANG G X, et al. AP2/ERF transcription factor in rice: Genome-wide canvas and syntenic relationships between monocots and eudicots [J]. Evolutionary Bioinformatics Online, 2012, 8: 321−355.
[7] ZHUANG J, DENG D X, YAO Q H, et al. Discovery, phylogeny and expression patterns of AP2-like genes in maize [J]. Plant Growth Regulation, 2010, 62(1): 51−58. DOI: 10.1007/s10725-010-9484-7
[8] GAO Y, HAN D, JIA W, et al. Molecular characterization and systematic analysis of NtAP2/ERF in tobacco and functional determination of NtRAV-4 under drought stress [J]. Plant Physiology and Biochemistry, 2020, 156: 420−435. DOI: 10.1016/j.plaphy.2020.09.027
[9] CHARFEDDINE M, SAÏDI M N, CHARFEDDINE S, et al. Genome-wide analysis and expression profiling of the ERF transcription factor family in potato (Solanum tuberosum L. ) [J]. Molecular Biotechnology, 2015, 57(4): 348−358. DOI: 10.1007/s12033-014-9828-z
[10] SHARMA M K, KUMAR R, SOLANKE A U, et al. Identification, phylogeny, and transcript profiling of ERF family genes during development and abiotic stress treatments in tomato [J]. Molecular Genetics and Genomics, 2010, 284(6): 455−475. DOI: 10.1007/s00438-010-0580-1
[11] PIRRELLO J, PRASAD B C, ZHANG W S, et al. Functional analysis and binding affinity of tomato ethylene response factors provide insight on the molecular bases of plant differential responses to ethylene [J]. BMC Plant Biology, 2012, 12: 190. DOI: 10.1186/1471-2229-12-190
[12] ZHU X L, WEI X H, WANG B Q, et al. Identification and analysis of AP2/ERF gene family in tomato under abiotic stress [J]. Biocell, 2020, 44(4): 777−803. DOI: 10.32604/biocell.2020.010153
[13] JIN J H, WANG M, ZHANG H X, et al. Genome-wide identification of the AP2/ERF transcription factor family in pepper (Capsicum annuum L. ) [J]. Genome, 2018, 61(9): 663−674. DOI: 10.1139/gen-2018-0036
[14] LI D L, HE Y J, LI S H, et al. Genome-wide characterization and expression analysis of AP2/ERF genes in eggplant (Solanum melongena L. ) [J]. Plant Physiology and Biochemistry, 2021, 167: 492−503. DOI: 10.1016/j.plaphy.2021.08.006
[15] HU L F, LIU S Q. Genome-wide identification and phylogenetic analysis of the ERF gene family in cucumbers [J]. Genetics and Molecular Biology, 2011, 34(4): 624−633. DOI: 10.1590/S1415-47572011005000054
[16] KARANJA B K, XU L, WANG Y, et al. Genome-wide characterization of the AP2/ERF gene family in radish (Raphanus sativus L. ): Unveiling evolution and patterns in response to abiotic stresses [J]. Gene, 2019, 718: 144048. DOI: 10.1016/j.gene.2019.144048
[17] GHORBANI R, ZAKIPOUR Z, ALEMZADEH A, et al. Genome-wide analysis of AP2/ERF transcription factors family in Brassica napus [J]. Physiology and Molecular Biology of Plants, 2020, 26(7): 1463−1476. DOI: 10.1007/s12298-020-00832-z
[18] LICAUSI F, GIORGI F M, ZENONI S, et al. Genomic and transcriptomic analysis of the AP2/ERF superfamily in Vitis vinifera [J]. BMC Genomics, 2010, 11: 719. DOI: 10.1186/1471-2164-11-719
[19] ZHANG H B, ZHANG D B, CHEN J, et al. Tomato stress-responsive factor TSRF1 interacts with ethylene responsive element GCC box and regulates pathogen resistance to Ralstonia solanacearum [J]. Plant Molecular Biology, 2004, 55(6): 825−834. DOI: 10.1007/s11103-005-2140-3
[20] ZHOU J X, ZHANG H B, YANG Y H, et al. Abscisic acid regulates TSRF1-mediated resistance to Ralstonia solanacearum by modifying the expression of GCC box-containing genes in tobacco [J]. Journal of Experimental Botany, 2008, 59(3): 645−652. DOI: 10.1093/jxb/erm353
[21] LI C W, SU R C, CHENG C P, et al. Tomato RAV transcription factor is a pivotal modulator involved in the AP2/EREBP-mediated defense pathway [J]. Plant Physiology, 2011, 156(1): 213−227. DOI: 10.1104/pp.111.174268
[22] ZHANG G Y, CHEN M, LI L C, et al. Overexpression of the soybean GmERF3 gene, an AP2/ERF type transcription factor for increased tolerances to salt, drought, and diseases in transgenic tobacco [J]. Journal of Experimental Botany, 2009, 60(13): 3781−3796. DOI: 10.1093/jxb/erp214
[23] JUNG J, WON S Y, SUH S C, et al. The barley ERF-type transcription factor HvRAF confers enhanced pathogen resistance and salt tolerance in Arabidopsis [J]. Planta, 2007, 225(3): 575−588. DOI: 10.1007/s00425-006-0373-2
[24] MCGRATH K C, DOMBRECHT B, MANNERS J M, et al. Repressor- and activator-type ethylene response factors functioning in jasmonate signaling and disease resistance identified via a genome-wide screen of Arabidopsis transcription factor gene expression [J]. Plant Physiology, 2005, 139(2): 949−959. DOI: 10.1104/pp.105.068544
[25] PAN I C, LI C W, SU R C, et al. Ectopic expression of an EAR motif deletion mutant of SlERF3 enhances tolerance to salt stress and Ralstonia solanacearum in tomato [J]. Planta, 2010, 232(5): 1075−1086. DOI: 10.1007/s00425-010-1235-5
[26] LAI Y, DANG F F, LIN J, et al. Overexpression of a Chinese cabbage BrERF11 transcription factor enhances disease resistance to Ralstonia solanacearum in tobacco [J]. Plant Physiology and Biochemistry, 2013, 62: 70−78. DOI: 10.1016/j.plaphy.2012.10.010
[27] CHEN C J, CHEN H, ZHANG Y, et al. TBtools: An integrative toolkit developed for interactive analyses of big biological data [J]. Molecular Plant, 2020, 13(8): 1194−1202. DOI: 10.1016/j.molp.2020.06.009
[28] HUANG S X, GAO Y F, LIU J K, et al. Genome-wide analysis of WRKY transcription factors in Solanum lycopersicum [J]. Molecular Genetics and Genomics, 2012, 287(6): 495−513. DOI: 10.1007/s00438-012-0696-6
[29] GUTTERSON N, REUBER T L. Regulation of disease resistance pathways by AP2/ERF transcription factors [J]. Current Opinion in Plant Biology, 2004, 7(4): 465−471. DOI: 10.1016/j.pbi.2004.04.007
[30] 徐志璇, 任仲海. 番茄AP2/ERF超家族重鉴定及过表达SlERF. D. 3株系表型分析 [J]. 园艺学报, 2020, 47(4):653−664. XU Z X, REN Z H. Re-identification of tomato AP2/ERF transcription factor superfamily and phenotypic analysis of the overexpressing SlERF. D. 3 lines [J]. Acta Horticulturae Sinica, 2020, 47(4): 653−664. (in Chinese)
[31] 练从龙, 兰金旭, 杨晶凡, 等. 基于冬凌草全长转录组的TIFY基因家族鉴定与表达分析 [J]. 福建农业学报, 2024, 39(3):290−301. LIAN C L, LAN J X, YANG J F, et al. Identification and expressions of TIFY family based on the full-length transcriptome in Isodon rubescens [J]. Fujian Journal of Agricultural Sciences, 2024, 39(3): 290−301. (in Chinese)
[32] 张麒, 陈静, 李俐, 等. 植物AP2/ERF转录因子家族的研究进展 [J]. 生物技术通报, 2018, 34(8):1−7. ZHANG Q, CHEN J, LI L, et al. Research progress on plant AP2/ERF transcription factor family [J]. Biotechnology Bulletin, 2018, 34(8): 1−7. (in Chinese)
[33] LAI Y, DANG F F, LIN J, et al. Overexpression of a pepper CaERF5 gene in tobacco plants enhances resistance to Ralstonia solanacearum infection [J]. Functional Plant Biology, 2014, 41(7): 758−767. DOI: 10.1071/FP13305
[34] SHI L P, LI X, WENG Y H, et al. The CaPti1–CaERF3 module positively regulates resistance of Capsicum annuum to bacterial wilt disease by coupling enhanced immunity and dehydration tolerance [J]. The Plant Journal, 2022, 111(1): 250−268. DOI: 10.1111/tpj.15790
[35] LIU K S, SHI L P, LUO H L, et al. Ralstonia solanacearum effector RipAK suppresses homodimerization of the host transcription factor ERF098 to enhance susceptibility and the sensitivity of pepper plants to dehydration [J]. The Plant Journal, 2024, 117(1): 121−144. DOI: 10.1111/tpj.16479
[36] 史建磊, 宰文珊, 苏世闻, 等. 番茄青枯病抗性相关miRNA的鉴定与表达分析 [J]. 生物技术通报, 2023, 39(5):233−242. SHI J L, ZAI W S, SU S W, et al. Identification and expression analysis of miRNA related to bacterial wilt resistance in tomato [J]. Biotechnology Bulletin, 2023, 39(5): 233−242. (in Chinese)
[37] GIRI M K, SWAIN S, GAUTAM J K, et al. The Arabidopsis thaliana At4g13040 gene, a unique member of the AP2/EREBP family, is a positive regulator for salicylic acid accumulation and basal defense against bacterial pathogens [J]. Journal of Plant Physiology, 2014, 171(10): 860−867. DOI: 10.1016/j.jplph.2013.12.015





 下载:
下载: