Cloning and Biological Characteristics of PpCaMK1 in Pseudocohnilembus persalinus
-
摘要:目的
钙调素依赖蛋白激酶(calmodulin-dependent protein kinases, CaMKs)是钙离子介导的信号转导通路中的关键组成部分,可能成为治疗盾纤虫病的药物靶点。本研究试图克隆水滴伪康纤虫(Pseudocohnilembus persalinus)的钙调素依赖蛋白激酶基因,分析其蛋白结构、功能,并基于该蛋白的氨基酸序列构建相关纤毛虫的系统进化树,以阐明水滴伪康纤虫CaMK的进化特征及与相关纤毛虫的系统发育关系。
方法参考水滴伪康纤虫RNA-seq测序结果,利用cDNA末端快速克隆(RACE)技术获得水滴伪康纤虫钙调素依赖蛋白激酶基因的全长cDNA序列,将其命名为PpCaMK1;利用生物信息学方法对该基因的序列特征、基因结构进行分析,预测该基因编码蛋白的理化性质、结构域及蛋白结构等,并构建其与相关纤毛虫CaMK的系统发育树。
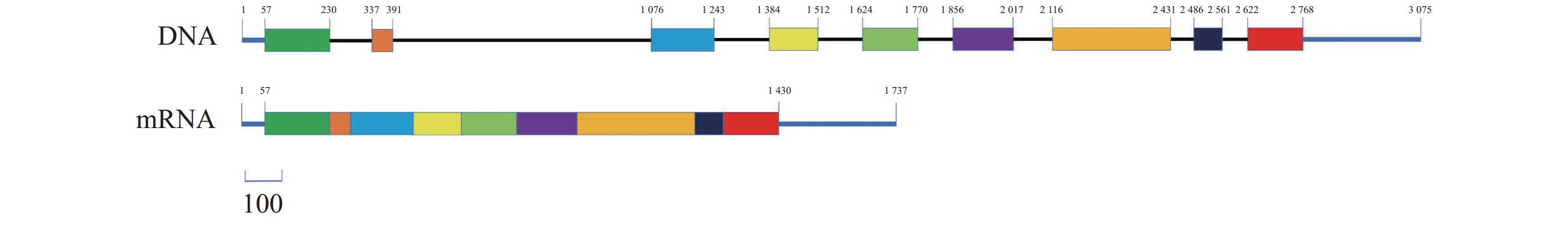
结果PpCaMK1基因包含有9个外显子和8个内含子,其cDNA全长为1 737 bp(GenBank序列号:PQ278249),其5′-UTR长度为56 bp,3′-UTR长度为307 bp,ORF总长度为1 374 bp,编码457个氨基酸残基。PpCaMK1总亲水性平均值为−0.802,不稳定指数为54.78,为亲水性不稳定蛋白。亚细胞定位预测分析表明PpCaMK1存在于细胞质-细胞核中。该蛋白无跨膜结构域,无信号肽。蛋白质的二级结构分析结果显示该蛋白含有的无规卷曲占比达50.33%。构建的系统发育树显示,水滴伪康纤虫PpCaMK1与寡膜纲纤毛虫同源蛋白具有较近的亲缘关系。
结论本研究克隆了水滴伪康纤虫PpCaMK1基因的全长cDNA,阐明该基因的结构,并初步分析PpCaMK1蛋白的基本理化性质及结构特征,结果可作为进一步研究PpCaMK1功能的基础,并为深入探究水滴伪康纤虫钙调素依赖蛋白激酶基因家族提供参考。
Abstract:ObjectiveGene associated with the calmodulin-dependent protein kinases (CaMKs) from Pseudocohnilembus persalinus was cloned to study its protein structure and function as well as genetic relation and evolutionary characteristics for the development of treatment for the parasitic scuticociliatosis in aquaculture species.
MethodBased on the RNA-seq of P. persalinus, the full-length cDNA sequence of PpCaMK1 was obtained using the Rapid Amplification of cDNA Ends (RACE) technique. Bioinformatic methods were employed to analyze the features, structure, physicochemical properties, domains and phylogenetic relationships with other ciliates of the gene.
ResultPpCaMK1 contained 9 exons and 8 introns. The full cDNA length was 1 737 bp (GenBank accession number: PQ278249) including a 5'-UTR of 56 bp, a 3'-UTR of 307 bp, and an ORF of 1 374 bp and encoded 457 amino acids. It was a hydrophilic and unstable protein with an average overall hydrophilicity of −0.802 and an instability index of 54.78. Distributed within the cytoplasm and nucleus, PpCaMK1 notably lacked the transmembrane domains and signal peptides and had a secondary structure composed of a significant proportion (i.e., 50.33%) of random coils. P. persalinus closely related to other ciliates in the class Oligohymenophorea.
ConclusionThe full-length cDNA of PpCaMK1 was successfully cloned and the basic biological and physicochemical characteristics studied paving the way for further research on the development of an effective treatment of the devastating disease on marine lives in aquacultural environment caused by the parasite P. persalinus.
-
Keywords:
- Pseudocohnilembus persalinus /
- CaMK /
- gene cloning /
- bioinformatics
-
0. 引言
【研究意义】水滴伪康纤虫(Pseudocohnilembus persalinus)隶属纤毛门(Ciliophora),盾纤目(Scuticociliatida),伪康纤虫属(Pseudocohnilembus)。盾纤毛虫是水产养殖中常见、多发且具有危害性的原生动物[1],属兼性寄生虫,不但能摄食细菌、微藻等,也能寄生于贝类外套腔、鱼类和甲壳动物的组织中,以其细胞和组织残留物为食[2],在感染时通常会伴随其他细菌性疾病的发生[3]。水滴伪康纤虫在富营养化的水体中极易繁殖,造成水体环境的急剧恶化[4]。克隆水滴伪康纤虫重要基因并了解其功能,应用生物信息学技术预测候选抗原,筛选药物靶点,可为水滴伪康纤虫的防治研究提供重要参考。【前人研究进展】钙/钙调素依赖性蛋白激酶(calcium/calmodulin-dependent protein kinases, CaMKs)是一类丝氨酸/苏氨酸蛋白激酶,是钙离子介导的信号转导通路中的关键组成部分,是钙调素下游的一类重要靶点蛋白,被Ca2+-CaM复合体激活[5]。关于CaMK的研究主要集中于脊椎动物和真菌中,在原生动物和植物中的研究较少。对CaMK参与真菌生长、次生代谢和致病性调控的研究表明,其调控作用因病原种类、寄生部位等因素不同而存在差异 [6−8]。Kato等[9]研究与人钙调素依赖性蛋白激酶(CaMK)同源的恶性疟原虫(Plasmodium falciparum)蛋白激酶2(PfPK2)的表征,PfPK2 位于恶性疟原虫培养物的裂殖子中,纯化的PfPK2 具有Ca2+和钙调蛋白依赖性方式的蛋白激酶活性,它可能与分裂子入侵过程中Ca2+信号的调节有关。在刚地弓形虫(Toxoplasma gondii)中,研究者发现了一种CaMK 同源物(CaMK-related kinase, TgCaMKrk)[10],并证实其在受感染的细胞中表达,定位于速殖子的顶端,与弓形虫对宿主的入侵相关,因此被认为是一种潜在的药物靶点。鉴于CaMKs在Ca2+信号转导中的重要作用,并与寄生虫入侵宿主过程相关,因此,亟需对水滴伪康纤虫的CaMKs基因进行深入的研究。【本研究切入点】尽管水滴伪康纤虫的全基因组测序已完成[11],也构建了其全长cDNA文库[12],但仍有大量的表达序列标签(expressed sequence tag,EST),序列并未得到有效注释,同时也未见对其重要功能基因进行克隆的研究报道。由于CaMK蛋白在生命活动中执行重要的生理功能,本研究拟应用克隆全长cDNA序列常用的cDNA末端快速扩增(rapid-amplification of cDNA ends, RACE)技术[13],对水滴伪康纤虫的CaMK基因进行全长cDNA克隆测序,并对其基因结构进行解析和蛋白结构、功能的预测分析,为深入研究水滴伪康纤虫基因及其功能提供基础信息。【拟解决的关键问题】利用RACE技术克隆水滴伪康纤虫CaMK基因家族其中1个基因的全长,阐明该基因的序列特征、基因结构,并对该基因所编码蛋白的理化特性、结构域及蛋白高级结构进行预测,为进一步研究该蛋白家族的功能提供基础资料;同时构建基于该CaMK蛋白序列的系统发育树,以阐明水滴伪康纤虫CaMK蛋白的进化特征及相关纤毛虫的系统发育关系。
1. 材料与方法
1.1 水滴伪康纤虫的培养和收集
水滴伪康纤虫由中国海洋大学原生动物教研室惠赠,本研究室保存。培养和收集参照林能锋等[12] 的方法,并稍做调整。具体方法如下:将1 mL的水滴伪康纤虫传代保存虫液接种于50 mL的过滤海水中,滴加500 μL过夜培养的E.coli DH5α菌液,于18 ℃培养箱中培养3~4 d,待密度大于4×104 ind·mL−1后进行营养虫的收集, 4 000 r·min−1离心5 min,弃上清,加入1 mL过滤海水重悬虫体沉淀至1.5 mL离心管中,8 000 r·min−1离心3 min,弃上清,得到营养虫,用于总RNA的提取。
1.2 总RNA的提取与cDNA合成
用TAKARA MiniBEST Universal RNA Extraction Kit(code No.9767)试剂盒进行总RNA的提取,方法参照试剂盒说明。超微量紫外可见分光光度计DS-11(DeNovix)测定总RNA浓度和质量。使用PrimeScriptTM RT reagent Kit(TAKARA code No.RR037A)试剂盒进行反转录试验,反转录的反应液的配制按试剂盒说明书进行,使用总RNA 1 μg,总反应体积为20 μL。反应程序为37 ℃ 15 min,85 ℃ 5 s。合成的cDNA置于−40 ℃冰箱中保存。
1.3 水滴伪康纤虫CaMK1基因核心片段的克隆
通过实验室前期RNAseq结果(NCBI序列号:SRX20359988),得到可能为蛋白激酶相关基因c15077的部分序列,根据该序列,应用在线引物设计工具Primer3(https://primer3.ut.ee/)设计c15077F1、c15077R1、c15077F2、c15077R2和c15077F3等5条引物(表1),以水滴伪康纤虫cDNA为模板,使用Premix Taq™ (Ex Taq™ Version 2.0)(RR003A)扩增3段水滴伪康纤虫蛋白激酶的核心片段。PCR的反应体系25 μL:12.5 μL Premix Taq,上下游引物各1 μL(10 μmol·L−1),cDNA模板1 μL,ddH2O 9.5 μL。PCR反应程序:98 ℃ 15 s,52 ℃ 30 s,72 ℃延伸1 min,30个循环。1.2%琼脂糖凝胶电泳检测PCR产物。使用SanPrep 柱式DNA胶回收试剂盒(上海生工生物工程有限公司)对目的片段进行胶回收,并送至福州尚亚生物技术公司进行测序。
表 1 水滴伪康纤虫CaMK1基因克隆中应用的引物Table 1. Primers used for cloning PpCaMK1引物名称
Primer name引物序列(5′-3′)
Primer sequence(5′-3′)目的
Purposec15077 F1 GGTTTAGAGTTTAGACGCTGA PpCaMK1核心片段1克隆 c15077 R1 CTTCGTCCCAGTCGATTAAC PpCaMK1核心片段1克隆 c15077 F2 TTGGTGTTTCTATGGTCGCA PpCaMK1核心片段2克隆 c15077 R2 CTTCTTCGTTTACCTCCTCC PpCaMK1核心片段2、3克隆 c15077 F3 GCTGATGATGGACAGTTAATCG PpCaMK1核心片段3克隆 c15077 3′ F1 GATTACGCCAAGCTTCGACTGGGACGAAGAGGATTCAAAGTGG 第一轮3′ RACE 克隆 c15077 3′ F2 GATTACGCCAAGCTTGTTCAAAGGGTTACTCTGGTAAAGCTGC 第二轮3′ RACE克隆 c15077 5′ R1 GATTACGCCAAGCTTCCACTTTGAATCCTCTTCGTCCCAGTCG 第一轮5′ RACE克隆 c15077 5′ R2 GATTACGCCAAGCTTTTACCATCAGCGTCTAAACTCTAAACCAT 第二轮5′ RACE克隆 UPM(long) SMARTer RACE 5′/3′Kit(TAKARA code No.050421)提供 通用长引物 UPM(short) SMARTer RACE 5′/3′Kit(TAKARA code No.050421)提供 通用短引物 1.4 水滴伪康纤虫CaMK1 5′端和3′端序列扩增
RACE试验参照SMARTer RACE 5′/3′Kit(TAKARA code No.050421)试剂盒说明,将RNA反转录成5′/3′ RACE-Ready cDNA。根据1.3中获得的PpCaMK1基因核心片段设计RACE引物(表1),对该基因的5′端和3′端序列分别扩增。
根据试剂盒说明书配制41.5 μL的 Master Mix,第一轮PCR的反应体系50 μL:2.5 μL 5′-/ 3′-RACE-Ready cDNA、1 μL c15077 3′ F1/ c15077 5′ R1(10 μmol·L−1)、5 μL UPM Mix、1 μL ddH2O、41.5 μL Master Mix。UPM对照组不加c15077 3′ F1和c15077 5′ R1引物,GSP对照组不加UPM引物,两个对照组反应最终都使用ddH2O定容至50 μL。3′端的扩增采用Touch down PCR,反应程序:94 ℃ 30 s,72 ℃ 3 min,5个循环;94 ℃ 30 s,68 ℃ 30 s,72 ℃ 3 min,5个循环;94 ℃ 30 s,65.5 ℃ 30 s,72 ℃ 3 min,25个循环。5′端扩增的反应程序:94 ℃ 30 s,68 ℃ 30 s,72 ℃ 3 min,25个循环。
第二轮巢式PCR:取5 μL第1次PCR的产物到245 μL的Tricine-EDTA缓冲液中,将其稀释50倍。反应体系50 μL:5 μL稀释的第一次PCR产物,1 μL c15077 3′ F2/c15077 5′ R2(10 μmol·L−1)、1 μL UPM(short)、1.5 μL H2O、41.5 μL Master Mix,反应程序:94 ℃ 30 s,60 ℃ 30 s,72 ℃ 3 min,20个循环。PCR产物于琼脂糖凝胶电泳(1.5%)检测,使用SanPrep 柱式DNA胶回收试剂盒(上海生工)对其进行胶回收。
1.5 水滴伪康纤虫CaMK1 5′端和3′端序列的In-Fusion克隆
参照SMARTer RACE 5′/3′Kit(TAKARA code No.050421)试剂盒中关于In-Fusion克隆的部分,配制10 μL的In-Fusion 克隆反应体系:2 μL 5×In-Fusion 快速组装克隆预混液、1 μL 线性化载体、7 μL 纯化的 PCR 片段,混匀;在50 ℃下孵育反应15 min。将感受态细胞置于冰上解冻,解冻后轻轻混合以确保均匀分布,然后将50 μL的感受态细胞转入1.5 mL的EP管中,向感受态细胞中加入2.5 μL的In-Fusion 克隆反应体系,将试管置于冰上30 min;在42 ℃下对细胞进行45 s热休克;将试管置于冰上1~2 min;SOC培养基加热至37 ℃后,加入1.5 mL的EP管中至总体积为500 μL;于37 ℃,220 r·min−1振荡1 h;取100 μL在含氨苄青霉素的LB平板中涂板,平板在37 ℃培养箱孵育过夜;第2天从平板中挑取8~10个单菌落,在含氨苄青霉素的LB培养基中过夜培养,进行菌液PCR验证后提取质粒(SanPrep 柱式质粒 DNA 小量抽提试剂盒,上海生工),送至福州尚亚生物技术公司测序。
1.6 水滴伪康纤虫CaMK1生物信息学及系统进化分析
通过Bioedit软件[14]对所测得的DNA序列进行拼接,得到完整的cDNA序列。通过Expasy的Translate tool(https://web.expasy.org/translate/)将cDNA翻译成蛋白质序列。通过Expasy 的ProtParam tool(https://web.expasy.org/protparam/)对蛋白质的分子量和等电点进行预测。使用在Expasy的ProtScale分析软件(https://web.expasy.org/protscale/)对该蛋白质的疏水性进行分析。使用NetPhos-3.1(https://services.healthtech.dtu.dk/services/NetPhos-3.1/)对蛋白磷酸化位点进行预测。使用NetOGlyc-4.0(https://services.healthtech.dtu.dk/services/NetOGlyc-4.0/)对蛋白质糖基化位点进行预测。使用SignalP-6.0(https://services.healthtech.dtu.dk/services/SignalP-6.0/)对蛋白信号肽进行预测。使用TMHMM-2.0(https://services.healthtech.dtu.dk/services/TMHMM-2.0/)和WoLFP SORT(https://wolfpsort.hgc.jp)分别预测PpCaMK1的跨膜结构和亚细胞定位情况。使用NCBI的CD-SEARCH工具(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)的和ScanProsite(https://prosite.expasy.org/)对蛋白质保守结构域进行预测。使用SOPAM(https://npsa.lyon.inserm.fr/cgi-bin/secpred_sopma.pl)预测蛋白质的二级结构。使用SWISS-MODEL(https://swissmodel.expasy.org/interactive)程序预测蛋白质的三级结构。
构建系统发育树:应用NCBI中的BLAST程序搜索相似序列,DNAMAN9.0(Lynnon Corporation, Quebec, Canada)对PpCaMK1蛋白进行多重序列比。应用MEGA11.0[15]软件中的邻接法(Neighbor-Joining, NJ)构建系统进化树, Bootstrap值设为
1000 。2. 结果与分析
2.1 水滴伪康纤虫PpCaMK1序列克隆
以水滴伪康纤虫营养期cDNA为模板,扩增3条PpCaMK1的核心片段,PCR产物经1.2%的琼脂糖凝胶电泳鉴定为单一扩增带,且条带大小与预期片段长度一致(图1A),3条核心片段的长度分别为365、426、649 bp。RACE扩增基因的3′端片段大小约700 bp,5′端的片段大小约300 bp(图1B)。
2.2 PpCaMK1序列全长和编码蛋白质的理化性质
通过Bioedit拼接序列,水滴伪康纤虫CaMK1基因的cDNA全长为1 737 bp(GenBank序列号:PQ278249),其中,5′-UTR长度为56 bp,3′-UTR长度为307 bp,开放阅读片段ORF总长度为1 374 bp,编码457个氨基酸(图2),通过Snapgene软件将cDNA与水滴伪康纤虫全基因组DNA (NCBI基因组序列号:GCA_001447515.1)比对绘制条形图,PpCaMK1基因有9个外显子、8个内含子,所有的内含子都符合GT-AG规则(图3)。
![]() 图 2 水滴伪康纤虫CaMK1基因全长cDNA序列及对应的氨基酸序列ORF用大写字母表示,5′-UTR和3′-UTR用小写字母表示。起始密码子加框显示,终止密码子以星号标注。Figure 2. Full-length cDNA sequence and corresponding amino acid sequence of PpCaMK1ORF domain shown in uppercase letters; 5′-UTR and 3′-UTR domains in lowercase letters; start codon in box; stop codon with asterisk.
图 2 水滴伪康纤虫CaMK1基因全长cDNA序列及对应的氨基酸序列ORF用大写字母表示,5′-UTR和3′-UTR用小写字母表示。起始密码子加框显示,终止密码子以星号标注。Figure 2. Full-length cDNA sequence and corresponding amino acid sequence of PpCaMK1ORF domain shown in uppercase letters; 5′-UTR and 3′-UTR domains in lowercase letters; start codon in box; stop codon with asterisk.预测结果显示PpCaMK1基因编码的蛋白质分子量为53.1 kDa,理论等电点为8.96。该蛋白质由碳、氢、氧、氮、硫5种元素组成,其分子式为C
2346 H3740 N658O712S16。它由常见的20种氨基酸组成,其中含量最高的为赖氨酸(11.6%),其次是谷氨酰胺(8.8%),含量最低的是半胱氨酸(1.1%),其次是色氨酸(1.3%)。负电荷残基数(天冬氨酸+谷氨酸)为61,正电荷残基数(精氨酸+赖氨酸)为70。PpCaMK1蛋白质的总亲水性平均值为−0.802,不稳定指数为54.78,为亲水性不稳定蛋白。根据ProtScale分析得到的亲疏水信号图显示,PpCaMK1蛋白N端和C端都含有亲水头部,中部氨基酸也表现出较强的亲水性,整体来看,峰值分布于0以下,亦证明其为亲水性蛋白。2.3 PpCaMK1编码蛋白质的修饰位点预测
磷酸化和糖基化等蛋白翻译后修饰常影响蛋白激酶的活性。应用NetPhos-3.1软件对蛋白的磷酸化位点预测结果显示,PpCaMK1蛋白共有38个磷酸化位点,其中有21个丝氨酸(Serine)磷酸化位点,11个苏氨酸(Threonine)磷酸化位点,6个酪氨酸(Tyrosine)磷酸化位点。使用NetOGlyc-4.0对蛋白质的糖基化位点进行预测,PpCaMK1蛋白有16个潜在的糖基化位点。
2.4 PpCaMK1编码蛋白质的信号肽、跨膜结构域及亚细胞定位预测
蛋白的信号肽预测结果显示PpCaMK1蛋白无信号肽序列,是一类非分泌性蛋白。蛋白的跨膜结构预测结果表明PpCaMK1不存在跨膜结构,非膜蛋白。对PpCaMK1的亚细胞定位预测结果显示其主要定位于细胞质-细胞核(cyto-nucl)中。
2.5 PpCaMK1编码蛋白质的结构域预测
对PpCaMK1蛋白的结构域预测结果表明该蛋白属于类PK超级蛋白激酶家族(PKc_like Superfamily),具有STKc_LKB1_CaMKK结构域,该结构域含有蛋白激酶的特征性ATP结合激酶区(ATP binding site)和活性结合位点。经过ScanProsite进一步分析可知PpCaMK1蛋白84~371位氨基酸为蛋白激酶保守结构域,其中90~113位氨基酸为ATP结合位点保守基序LGRGAFGKVKrAvqkstgqtYAVK,230~242位氨基酸为丝苏氨酸活性位点保守基序IiHrDIKpqNILF。
2.6 PpCaMK1编码蛋白质的结构特征
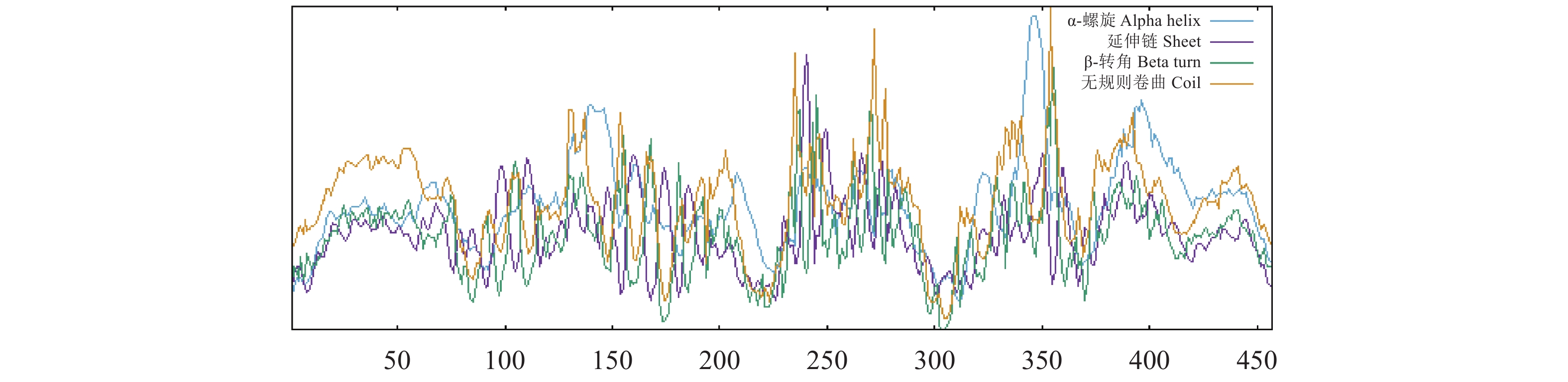
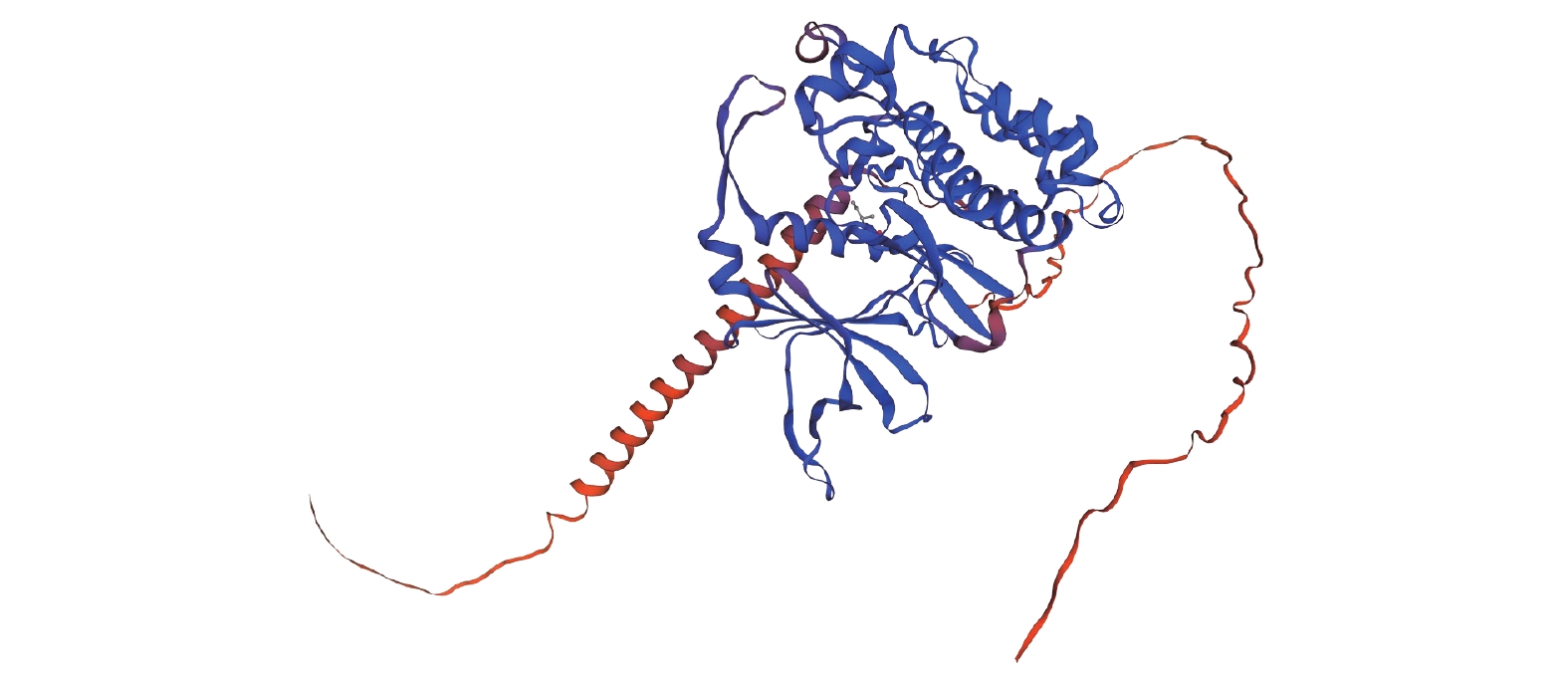
蛋白质的二级结构预测结果显示该蛋白由32.39%的α-螺旋、4.81%的β-转角、50.33%无规卷曲和12.47%延伸链组成(图4)。使用SWISS-MODEL预测得到的蛋白质三级结构如图5所示,该模型的GMQE值(全球模型质量估计)为0.76,表明该预测模型质量较好。
2.7 PpCaMK1编码蛋白质的系统进化分析
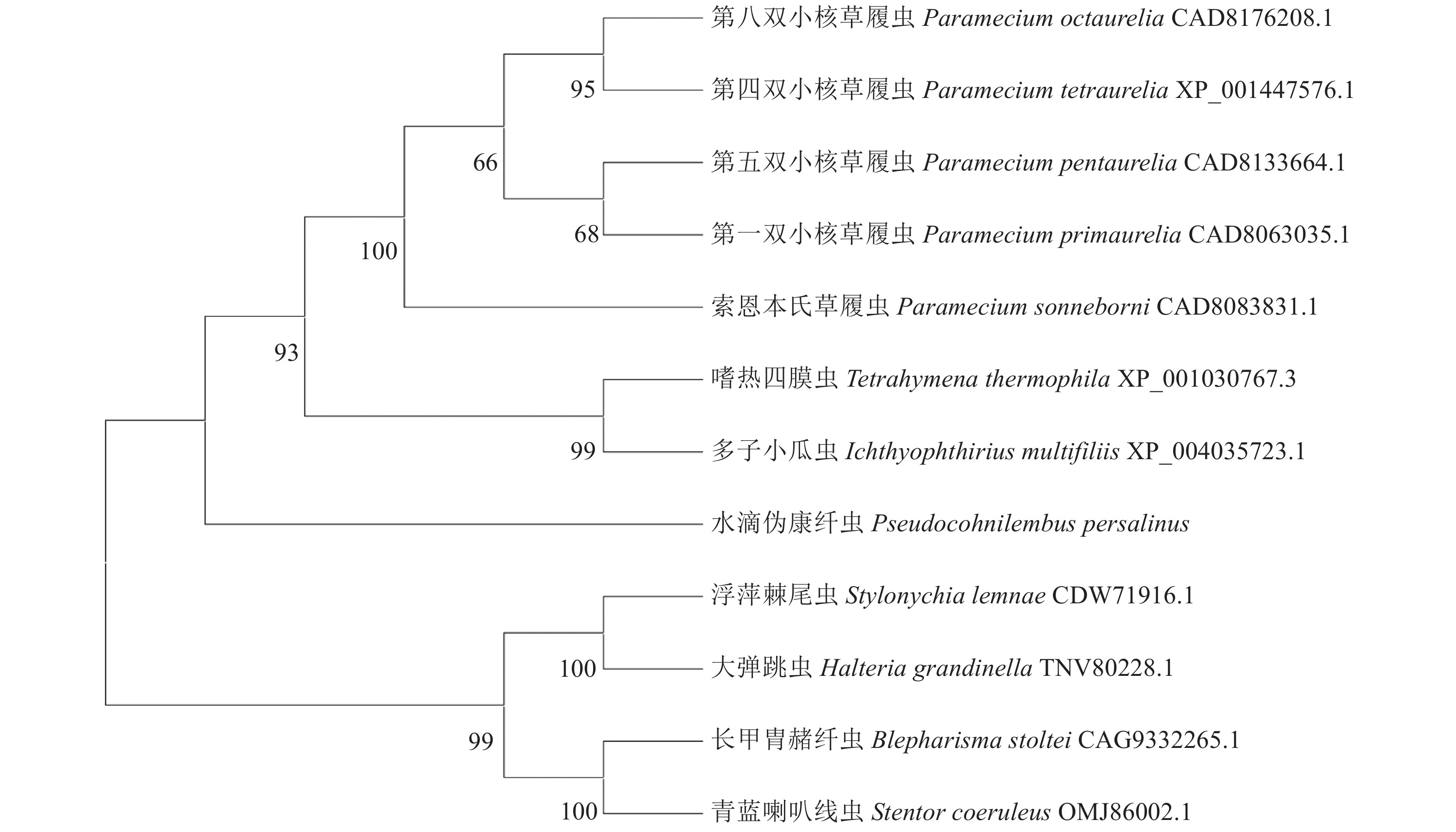
在NCBI中搜索与PpCaMK1具有较高相似度的蛋白质序列。得到嗜热四膜虫(Tetrahymena thermophila,XP_001030767.3)、多子小瓜虫(Ichthyophthirius multifiliis,XP_004035723.1)、第八双小核草履虫(Paramecium octaurelia,CAD8176208.1)、第四双小核草履虫(Paramecium tetraurelia,XP_001447576.1)、索恩本氏草履虫(Paramecium sonneborni,CAD8083831.1)、第五双小核草履虫(Paramecium pentaurelia,CAD8133664.1)、第一双小核草履虫(Paramecium primaurelia,CAD8063035.1)、浮萍棘尾虫(Stylonychia lemnae,CDW71916.1)、长甲胄赭纤虫(Blepharisma stoltei,CAG9332265.1)、大弹跳虫(Halteria grandinella,TNV80228.1)、青蓝喇叭线虫(Stentor coeruleus,OMJ86002.1)的蛋白质序列。通过DNAMAN进行上述序列进行蛋白质序列同源比对(图6),将多重序列比对图和结构域预测图进行对照,可以发现在PpCaMK1的第280位氨基酸之前,序列相似度高的基本是ATP结合激酶区和活性结合位点。
构建PpCaMK1蛋白与几种纤毛虫类的CaMK蛋白的NJ(Neighbor-Joining)进化树(图7)的结果显示,水滴伪康纤虫与草履虫、嗜热四膜虫和多子小瓜虫处在同一进化支上,具有较近的亲缘关系,与分类上同属寡膜纲一致。
3. 讨论
目前对盾纤虫病的防治措施有使用外用消毒剂药浴浸泡[16]、中草药浸泡[17]、中草药混饲和口服专用药物[18]等,这些措施能有效杀灭鱼类体表的纤毛虫,但却不能有效杀灭入侵体内组织器官的纤毛虫,或对鱼体体质有害[3]。同时,一些药物的滥用,会使虫体产生抗药性并有可能造成水环境的污染,依靠生物技术研制基因工程疫苗或者核酸疫苗是盾纤虫免疫防制方法的一个重要方向。选择合适的具有免疫原性的蛋白质为抗原则是研制疫苗的关键步骤。蛋白质的亲水性和二级结构是判断其能否作为抗原结构基础的重要指标[19],亲水性氨基酸位于蛋白质表面,连在一起形成抗原表位,亲水性同时也是判断B细胞抗原表位的主要参数[20]。本研究预测得到PpCaMK1的总亲水性平均值为−0.802,说明其亲水性较高。在PpCaMK1蛋白质的二级结构中,β-转角和无规卷曲超过总氨基酸的50%,这些结构主要位于蛋白表面,结构相对松散,是潜在的抗原表位位点。因此,从PpCaMK1的结构来看,该蛋白有可能成为抗盾纤虫的鱼类疫苗的候选抗原。
目前,关于原生动物的钙依赖蛋白激酶集中于对顶复门动物CDPKs(calcium dependent protein kinases)的研究,而CaMKs相关报道相对较少。CDPK是由一个编码CaMK的基因与一个编码钙调素的基因在进化中融合而形成的[21],CDPK结构主要由N端可变结构域、Ser/Thr激酶结构域、自抑制连接结构域和类钙调激素结构域构成[22−23],一般来说,类钙调激素结构域具有与钙离子结合的EF-hand钙结合结构域[24],CaMKs不存在EFF-hand结构,它不与Ca2+结合,而是被Ca2+-CaM复合体激活。弓形虫TgCaMKrk具有CaMKs家族保守的催化结构域,但缺乏调节结构域,该激酶活性既不需要Ca2+也不需要钙调蛋白,其具有自磷酸化和组蛋白磷酸化活性[10]。本研究预测的水滴伪康纤虫PpCaMK1蛋白没有EF-hand调节结构域,但具有STKc_LKB1_CaMKK结构域,属于典型的类PK超级蛋白激酶家族(PKc_Like Superfamily)的催化结构域,该结构域主要由丝/苏氨酸特异性蛋白激酶催化结构域组成。LKB1/CaMKK 亚家族是 CaMKs 超家族的一部分,CaMKK 与 LKB1 均为腺苷酸活化蛋白激酶 (AMPK)的上游激酶,其活性均依赖于 Ca2+/CaM 复合物[8],而PpCaMK1也具有相似的结构域,因此推测其也可被Ca2+/CaM 复合物激活,因此,PpCaMK1属于CaMKs 超家族成员。蛋白预测结果显示PpCaMK1不存在跨膜结构域和信号肽,但是有多个磷酸化位点,具有催化下游效应蛋白的功能,因而推测其在水滴伪康纤虫生命活动及在入侵宿主过程中起到细胞信号转导的功能。
多种亚型的CaMK已被确定定位在细胞质和细胞核中,这说明CaMKs执行重要的生物学功能。CaMKs具有多种底物蛋白,根据CaMK的底物特异性,常分为两大类:一类以CaMKIII为底物特异性钙调蛋白激酶;而另一类,如CaMKI、CaMKII、CaMKIV、CaMKK均可磷酸化多种底物蛋白[25]。因此了解CaMK的功能需要独立对每个CaMK进行功能分析。由于钙/钙调素依赖蛋白激酶参与的信号调节网络非常复杂,对其参与的信号通路及不同通路间的相互作用机制的解析是对其功能研究的重要方面,这些研究可全面了解 CaMK 的信号级联从而有助于设计出更好的寄生虫靶向药物。目前,尚未见有对水滴伪康纤虫的钙调素依赖蛋白激酶的研究,与PpCaMK1互作的蛋白及其作用底物、其在细胞中的定位及其对水滴伪康纤虫生命活动的调控机制还有待进一步研究。
-
图 2 水滴伪康纤虫CaMK1基因全长cDNA序列及对应的氨基酸序列
ORF用大写字母表示,5′-UTR和3′-UTR用小写字母表示。起始密码子加框显示,终止密码子以星号标注。
Figure 2. Full-length cDNA sequence and corresponding amino acid sequence of PpCaMK1
ORF domain shown in uppercase letters; 5′-UTR and 3′-UTR domains in lowercase letters; start codon in box; stop codon with asterisk.
表 1 水滴伪康纤虫CaMK1基因克隆中应用的引物
Table 1 Primers used for cloning PpCaMK1
引物名称
Primer name引物序列(5′-3′)
Primer sequence(5′-3′)目的
Purposec15077 F1 GGTTTAGAGTTTAGACGCTGA PpCaMK1核心片段1克隆 c15077 R1 CTTCGTCCCAGTCGATTAAC PpCaMK1核心片段1克隆 c15077 F2 TTGGTGTTTCTATGGTCGCA PpCaMK1核心片段2克隆 c15077 R2 CTTCTTCGTTTACCTCCTCC PpCaMK1核心片段2、3克隆 c15077 F3 GCTGATGATGGACAGTTAATCG PpCaMK1核心片段3克隆 c15077 3′ F1 GATTACGCCAAGCTTCGACTGGGACGAAGAGGATTCAAAGTGG 第一轮3′ RACE 克隆 c15077 3′ F2 GATTACGCCAAGCTTGTTCAAAGGGTTACTCTGGTAAAGCTGC 第二轮3′ RACE克隆 c15077 5′ R1 GATTACGCCAAGCTTCCACTTTGAATCCTCTTCGTCCCAGTCG 第一轮5′ RACE克隆 c15077 5′ R2 GATTACGCCAAGCTTTTACCATCAGCGTCTAAACTCTAAACCAT 第二轮5′ RACE克隆 UPM(long) SMARTer RACE 5′/3′Kit(TAKARA code No.050421)提供 通用长引物 UPM(short) SMARTer RACE 5′/3′Kit(TAKARA code No.050421)提供 通用短引物 -
[1] 常志强,陈钊,李泽鹏,等. 中兽药驱虫复方制剂防治海水养殖中盾纤毛虫的临床试验研究[J]. 中兽医医药杂志,2019,38(5) :5−9. CHANG Z Q,CHEN Z,LI Z P,et al. Clinical assessment of traditional Chinese medicine compound on the ciliate parasite in marine aquaculture[J]. Journal of Traditional Chinese Veterinary Medicine,2019,38(5) :5−9. (in Chinese)
[2] IGLESIAS R,PARAMÁ A,ALVAREZ M F,et al. Philasterides dicentrarchi (Ciliophora,Scuticociliatida) as the causative agent of scuticociliatosis in farmed turbot Scophthalmus maximus in Galicia (NW Spain) [J]. Diseases of Aquatic Organisms,2001,46(1) :47−55.
[3] 崔龙波,刘冉,王琛,等. 大菱鲆盾纤毛虫病的流行病学调查[J]. 科学养鱼,2015(3) :54−56. CUI L B,LIU R,WANG C,et al. Epidemiological investigation of Scophthalmus maximus shield ciliary disease[J]. Scientific Fish Farming,2015(3) :54−56. (in Chinese)
[4] 潘旭明,马洪钢,邵晨,等. 海洋纤毛虫:水滴伪康纤虫的口器发生及形态学重描述[J]. 水生生物学报,2012,36(3) :489−495. DOI: 10.3724/SP.J.1035.2012.00489 PAN X M,MA H G,SHAO C,et al. Stomatogenesis and morphological redescription of Pseudocohnilembus persalinus (Ciliophora:Scuticociliatida) [J]. Acta Hydrobiologica Sinica,2012,36(3) :489−495. (in Chinese) DOI: 10.3724/SP.J.1035.2012.00489
[5] BRAUN A P,SCHULMAN H. The multifunctional calcium/calmodulin-dependent protein kinase:From form to function[J]. Annual Review of Physiology,1995,57:417−445. DOI: 10.1146/annurev.ph.57.030195.002221
[6] 肖圣燕,程隆基,许凯,等. 桑褐斑壳丰孢菌pmcamk基因的克隆与表达时相分析[J]. 蚕业科学,2023,49(1) :15−22. XIAO S Y,CHENG L G,XU K,et al. Cloning and expression analysis of calcium /calmodulin-dependent protein kinases gene of Phloeospora maculans[J]. Acta Sericologica Sinica,2023,49(1) :15−22. (in Chinese)
[7] 吴迪,赵梓彤,李志勇,等. 玉米大斑病菌钙/钙调素依赖性蛋白激酶(CaMK) 基因家族的鉴定与表达模式分析[J]. 农业生物技术学报,2015,23(8) :1020−1030. WU D,ZHAO Z T,LI Z Y,et al. Identification and expression pattern analysis of calcium/calmodulin dependent protein kinases (CaMKs) gene family in Setosphaeria turcica[J]. Journal of Agricultural Biotechnology,2015,23(8) :1020−1030. (in Chinese)
[8] 蒋倩倩,毛仁燕,李永才,等. 梨果黑斑病菌AaCaMK基因克隆、生物信息学分析及其在侵染结构分化中的表达分析[J]. 微生物学通报,2021,48(12) :4664−4676. JIANG Q Q,MAO R Y,LI Y C,et al. Cloning,bioinformatics and expression analysis of AaCaMK gene on infection structure differentiation of Alternaria alternate,causal agent of pear black spot[J]. Microbiology China,2021,48(12) :4664−4676. (in Chinese)
[9] KATO K,SUDO A,KOBAYASHI K,et al. Characterization of Plasmodium falciparum protein kinase 2[J]. Molecular and Biochemical Parasitology,2008,162(1) :87−95. DOI: 10.1016/j.molbiopara.2008.07.007
[10] KATO K,SUGI T,TAKEMAE H,et al. Characterization of a Toxoplasma gondii calcium calmodulin-dependent protein kinase homolog[J]. Parasites &Vectors,2016,9(1) :405.
[11] XIONG J,WANG G Y,CHENG J,et al. Genome of the facultative scuticociliatosis pathogen Pseudocohnilembus persalinus provides insight into its virulence through horizontal gene transfer[J]. Scientific Reports,2015,5:15470.
[12] 林能锋,曾红. 水滴伪康纤虫全长cDNA文库的构建及EST分析[J]. 福建农业学报,2017,32(9) :921−925. LIN N F,ZENG H. Full-length cDNA Library and EST Analysis on Pesudocohnilembus persalinus[J]. Fujian Journal of Agricultural Sciences,2017,32(9) :921−925. (in Chinese)
[13] 贺斌,黄兴奇,余腾琼,等. cDNA末端快速扩增技术及其方法的改进[J]. 浙江农业科学,2012,53(9) :1352−1357. DOI: 10.3969/j.issn.0528-9017.2012.09.045 HE B,HUANG X Q,YU T Q,et al. Rapid amplification technology of cDNA terminal and its improvement[J]. Journal of Zhejiang Agricultural Sciences,2012,53(9) :1352−1357. (in Chinese) DOI: 10.3969/j.issn.0528-9017.2012.09.045
[14] HALL T A. BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series,1999,41(2) :95−98.
[15] KUMAR S,TAMURA K,NEI M. MEGA:Molecular Evolutionary Genetics Analysis software for microcomputers[J]. Computer Applications in the Biosciences,1994,10(2) :189−191.
[16] 宫春光,李凤晨. 牙鲆盾纤毛虫病的药物防治试验[J]. 水产科学,2007,26(9) :509−511. DOI: 10.3969/j.issn.1003-1111.2007.09.009 GONG C G,LI F C. Chemical treatment on protozoon Paralembus digitiformis in farmed Japanese flounder[J]. Fisheries Science,2007,26(9) :509−511. (in Chinese) DOI: 10.3969/j.issn.1003-1111.2007.09.009
[17] 林能锋,郑晨艳,王侯杰,等. 中草药浸出液体外驱杀两种水产致病性盾纤虫的研究[J]. 福建畜牧兽医,2017,39(5) :13−15. DOI: 10.3969/j.issn.1003-4331.2017.05.006 LIN N F,ZHENG C Y,WANG H J,et al. Repellent effect of Chinese herbal leachate to two species of scuticociliate in vitro[J]. Fujian Journal of Animal Husbandry and Veterinary Medicine,2017,39(5) :13−15. (in Chinese) DOI: 10.3969/j.issn.1003-4331.2017.05.006
[18] 崔青曼,袁春营,李春岭,等. 主要海水养殖鱼类白点病和盾纤毛虫病防治技术[J]. 水利渔业,2007,27(6) :85−87,94. DOI: 10.3969/j.issn.1003-1278.2007.06.037 CUI Q M,YUAN C Y,LI C L,et al. Prevention and control technology of white spot disease and scuticociliatosis in major marine aquaculture fish[J]. Journal of Hydroecology,2007,27(6) :85−87,94. (in Chinese) DOI: 10.3969/j.issn.1003-1278.2007.06.037
[19] 刘荣荣,张进顺,张晓磊,等. 刚地弓形虫棒状体蛋白10的生物信息学分析[J]. 中国病原生物学杂志,2017,12(9) :855−859. LIU R R,ZHANG J S,ZHANG X L,et al. Bioinformatic analysis of rhoptry protein 10 of Toxoplasma gondii[J]. Journal of Pathogen Biology,2017,12(9) :855−859. (in Chinese)
[20] SIVALINGAM G N,SHEPHERD A J. An analysis of B-cell epitope discontinuity[J]. Molecular Immunology,2012,51(3/4) :304−309.
[21] HARMON A C,GRIBSKOV M,HARPER J F. CDPKs–a kinase for every Ca2+ signal?[J]. Trends in Plant Science,2000,5(4) :154−159. DOI: 10.1016/S1360-1385(00)01577-6
[22] KLIMECKA M,MUSZYŃSKA G. Structure and functions of plant calcium-dependent protein kinases[J]. Acta Biochimica Polonica,2007,54(2) :219−233. DOI: 10.18388/abp.2007_3242
[23] LIU H L,CHE Z J,ZENG X R,et al. Genome-wide analysis of calcium-dependent protein kinases and their expression patterns in response to herbivore and wounding stresses in soybean[J]. Functional &Integrative Genomics,2016,16(5) :481−493.
[24] 费小钰,李红丽,王俊皓. 植物钙依赖蛋白激酶CDPK基因功能综述[J]. 吉林农业,2017(9) :104−105. FEI X Y,LI H L,WANG J H. Review on the function of plant calcium-dependent protein kinase CDPK gene[J]. Agriculture of Jilin,2017(9) :104−105. (in Chinese)
[25] SWULIUS M T,WAXHAM M N. Ca2+/calmodulin-dependent protein kinases[J]. Cellular and Molecular Life Sciences,2008,65(17) :2637−2657. DOI: 10.1007/s00018-008-8086-2




 下载:
下载: