Mapping of Early Senescence-related OsBRCA1 in Rice by BSA-seq Technique
-
摘要:目的 水稻早衰突变体是研究水稻衰老机制的良好载体。定位和克隆水稻早衰相关基因,有助于理解水稻早衰的遗传规律和分子机制,为相关基因的作用机制研究奠定基础。方法 用EMS化学诱变处理粳稻云引,获得一个稳定遗传的早衰突变体w14,与日本晴杂交构建F2分离群体,并在群体中各选择100个隐性单株和显性单株的DNA等量混合,利用BSA-seq分析两个DNA池之间的差异位点,定位水稻早衰相关基因。结果 突变体w14的早衰表型受单隐性基因控制,对两个DNA池测序结果进行单基因定位和突变基因的鉴定,发现单基因主峰位于Chr3,候选区间为 Chr3:27.5~29.5 Mb,进一步在目标区域内找到2个符合条件的候选因果变异。结论 候选基因 LOC_Os03g49210突变位点是由野生型的C突变为T,且该突变位于候选基因的第2 外显子上,属于错义突变,造成了该基因编码的第20 个氨基酸由T(苏氨酸)变成I(异亮氨酸),从而可能导致了基因功能的改变,因此,将该基因定为本研究的候选基因,因与人类BRCA1同源,命名为 OsBRCA1
。 Abstract:Objective Molecular mechanism of rice senescence was studied by locating and cloning the specific gene from a mutant artificially induced with early senescence.Methods Japonica rice Yunyin was treated with EMS to artificially induce a genetically stable early senescence w14. The mutant was crossed with Nipponbare to construct an isolated F2 population. Subsequently, equal parts of the DNA of 100 recessive and dominant plants in the population were mixed. The differential loci between these two DNA pools were analyzed by BSA-seq to locate the target early senescence-related gene.Results The early senescence phenotype of w14 was found to be controlled by a single recessive gene. The mutagenic gene mapped and identified by the specific sequence showed the main peak located in Chr 3 and the candidate interval Chr 3: 27.5–29.5 Mb. In the region, two qualified candidate causal variants were found.Conclusion The mutation site of the candidate gene Loc_ Os03g49210 located in the 2nd exon was determined to have changed in the wild-type from C to T. It was a non-synonymous mutation that resulted in the 20th amino acid encoded by the gene changed from T (threonine) to I (isoleucine) and led to the functional alternation. Being homologous with human BRCA1, the target gene was named OsBRCA1.-
Keywords:
- rice /
- mutant /
- early senescence /
- gene clone /
- BSA-seq
-
0. 引言
【研究意义】水稻(Oryza sativa)是全世界最主要的粮食作物之一,全球有超过50%的人口以大米为主食,水稻的高产和稳产是保障粮食安全的重要因素。而在农业生产中,水稻等禾谷类作物的产量95%来自叶片光合作用[1]。近年来,随着不断增长的人口压力、各种生物和非生物逆境胁迫以及城镇化带来的耕地面积不断缩小等因素均对水稻的产量构成了严重的威胁[2,3]。植物的生长发育的过程受到各种内外因素的影响。外因方面,某些物理和化学因素,如射线、强光、高温、盐、碱及干旱等环境因素可能会引起植物细胞的渗透胁迫,导致叶片的枯萎甚至早衰,进而影响作物的光合作用和代谢,造成作物的产量受到影响[4]。在遗传因素上,植物叶片早衰突变症状是常见的突变类型,这是因为某些与植物叶绿体的合成、发育和降解相关的重要基因发生了突变,失去了相应的基因功能,最后致使水稻叶色异常或早衰,进而导致水稻产量的下降。水稻作为单子叶植物分子研究的模式植物,其全基因组测序已经完成[5],我们可从中获取大量可利用的全长cDNA相关信息用于指导育种和生产。因此,开展水稻早衰突变体相关基因的克隆并进行其早衰机理的研究,深入了解水稻叶片衰老所参与的信号通路和调控机制,对于选育延缓衰老、抗衰老的水稻品种,保证粮食安全具有重要意义。【前人研究进展】衰老同时受到遗传基因与外界环境的影响,但在植物衰老过程中,遗传往往占据主导因素,这是植物在漫长的进化过程中建立的一种自我保护形式[6]。从植物叶片早衰数据库查询结果显示,在水稻中已鉴定出近160个与叶片衰老相关的基因,其中经遗传转化验证或互补验证的与叶片早衰相关的调控基因仅占三分之一[7]。在其中21个已克隆的水稻叶片早衰相关基因中,多数基因参与叶绿素降解[8]、过氧化反应[9] 、激素合成及信号传导[10]和逆境胁迫[11]等途径。研究结果显示,早衰突变体多数是因为植物抗氧化系统功能下降,导致叶片衰老。因此,克隆和研究更多导致水稻早衰突变体的相关基因,并进行其遗传机理的深入研究,有利于构建和完善植物早衰的遗传调控网络。然而,当前这些早衰相关基因大多是通过经典的正向遗传和图位克隆的方法获得的,传统的图位克隆方法存在研究周期长、通量低、工作量大等缺点。【本研究切入点】为加快水稻早衰基因的定位和克隆,本研究在前期工作通过传统图位克隆方法,已获得了早衰突变体w14的目标基因精细定位区间,因区间内包含有8个功能基因,无法最终确定其候选基因[12]。为了进一步确定早衰突变体的目标候选基因,同时也对图位克隆的结果进行验证,本研究通过BSA-Seq的方法从中寻找可能影响水稻出现早衰突变的位点。混合群体分离分析法(Bulked segregant analysis,BSA)是基于临时(如F1、F2分离群体)或永久群体(如重组自交系、近等基因系)表现出明显差异的个体,通过构建DNA混池进行基因定位。BSA已被应用于水稻质量性状基因[13]和数量性状基因(QTL)[14-15]的定位。但利用BSA对水稻早衰突变体的相关基因进行定位和遗传学分析相关研究鲜见报道。【拟解决的关键问题】本研究以w14和日本晴分别作为早衰突变体和野生型,配制 F2 代遗传群体,种植于田间到抽穗期开始调查表型。依据w14和野生型日本晴作为调查早衰表型的标准,选出极端材料构建基因池。 通过BSA-seq分析,初步确定w14中早衰相关基因的连锁区间,为早衰突变体w14的衰老相关基因的精细定位及基因克隆奠定基础。
1. 材料与方法
1.1 试验材料
日本晴为本课题组保有的粳稻常规品种。野生型粳稻品种云引(YY)自云南省农业科学院引进,经0.8% EMS诱变处理后从中筛选得到一个稳定遗传的早衰突变体,命名为w14。
1.2 试验方法
1.2.1 BSA-Seq群体构建
将早衰突变体w14与粳稻品种日本晴杂交得到F1杂交种,F1套袋自交并分单株收获得到F2种子,F2群体种植于田间生长至孕穗期时进行表型调查和取样,早衰突变型单株和野生型单株各取100个,分别提取DNA,进行DNA浓度测定后稀释成相同的浓度再等量混合,分别构成隐性基因池和显性基因池,再加突变体w14共3个样品委托广州基迪奥生物科技有限公司进行重测序分析,日本晴利用参考基因组用模块版本:IRGSP 1.0。基因集注释版本:MSU 7.0。水稻参考基因组大小为374 471 240 bp,共有12条染色体。水稻12条染色体的长度信息将用于统计量拟合和可视化,我们将染色体长度的信息保存在04.mapping/00.chr_length.tab文件中。
1.2.2 测序数据的评估和处理
对测序得到的原始测序读段(Sequenced reads或者Raw reads),里面含有带接头的、低质量的数据。为了保证信息分析质量,必须对原始测序读段过滤,得到待分析数据,后续分析都基于待分析数据。数据处理的步骤如下:
(1)去除带接头(Adapter)的数据对;
(2)当单端测序数据中含有的N的含量超过该条数据长度比例的10%时,需要去除此对成对数据;
(3)当单端测序数据中含有的低质量(Q≤5)碱基数超过该条数据长度比例的50%时,需要去除此对成对数据。
1.2.3 测序数据的质量修剪
先经过FASTQC的质控评估后如果发现待分析数据中依然含有一些接头与低质量的序列,利用TRIMMOMATIC对待分析数据进行质量修剪,修剪后的读段称为删节数据(Trimmed data)。
TRIMMOMATIC的处理内容主要包括以下方面:
(1)继续去除数据中的接头(Adapter)序列;
(2)以7 bp的滑窗扫描数据,若滑窗的Qphred均值低于20,则去除该窗口及其之后的序列(默认采用Q20标准);
(3)在成对数据中,当某一单端数据修剪后长度低于30 bp时,需要去除此成对数据。
1.2.4 读段比对
通过bwa mem程序将分析数据比对到参考基因组,利用samtools sort工具对匹配项(Alignment)按参考序列的顺序进行排序,利用Picard的Mark Duplicate去除比对结果中的重复,屏蔽PCR duplication的影响,并通过GATK进行InDel Realignment,即对存在插入缺失的位点附近进行局部重新比对,校正由于插入缺失引起的比对错误。
将各样本的比对结果(bam文件)存放在01.bam目录中。在Linux操作系统下,安装了samtools之后,用samtools view及less命令浏览bam文件,命令如下:samtools view-h sample.bam less或者用IGV等基因组浏览器载入bam文件,可视化浏览比对结果。
1.2.5 基因组序列扩增与比对分析
应用NCBI及http://rice.plantbiology.msu.edu等网站的数据库分析水稻LOC_Os03g49200的基因序列,利用引物设计软件Primer Premier 6设计引物扩增目标基因的基因组序列全长,并在上述网站下载该基因序列,在DNAMAN中进行序列比对分析。所设计的分段扩增目标基因的基因组序列全长的引物序列如表1。
表 1 用于 OsBRCA1基因组全长分段扩增的引物序列Table 1. Primer sequences for genome full-length fragment amplification on OsBRCA1序号
Number标记
Marker上游引物
Forward primer(5′-3′)下游引物
Reverse primer(5′-3′)1 49210-1 AATACCATATCGCCGTTTTCTT AACTTTTCCATCACATCGTTCTAA 2 49210-2 TTGCCCGCATAATGTGACTG AAGACTGGAGACTTGGGAGGTG 3 49210-3 TTTGCTATGCTGAACTCCCG TCCCTTCCACCTTCTTCTTTG 4 49210-4 GATATGTTGGGTCATTTTGGAGC TCAAGTTTGTAAGTTGGTGGTCG 5 49210-5 CAAGGTCCACAGGAAGAAGGT AAGATTGTTGCACTCAAAGAAATG 6 49210-6 CTGGCTAACTCCACATGCTCTT TTCCACTGGCCTACCTCACG 7 49210-7 CCAACTAATGAACCTGGAGCG CAATATGTGCAACCAAAATGTGAA 8 49210-8 GGGCAGCCTTCACTAATGACA TTACGCTGAAACAGGGAAATG 9 49210-9 GCTGAGGATGGAATACGAAGGA GGTGGAGGGAAATCGAGGAG 2. 结果与分析
2.1 BSA-Seq及测序数据质量分析
在之前的工作中,我们通过传统的基因图位克隆方法,获得了早衰突变体w14目标基因所在的精细定位区间,但该区间包括8个可能的候选基因,因而仍无法最终确定候选基因[12]。为了进一步确定目标基因的候选基因同时也对图位克隆的结果进行验证,我们通过BSA-Seq的方法,即通过分别对F2群体中的显性单株构成的野生型基因池和隐性单株构成的突变型基因池(各取100个单株)进行重测序分析,拟从中寻找可能影响水稻出现早衰突变的位点。
重测序分析SNP位点是如今进行基因序列差异分析和基因克隆的重要辅助手段。但是基因组测序的数据质量往往会对后续的分析造成重要的影响。在本研究中,除了其中一个亲本使用日本晴的网络数据库序列信息之外,我们分别对突变体w14、隐性基因池和显性基因池进行了测序分析,同时对这3个样本的测序数据量及测序质量进行了统计分析,结果表明,剔除低质量的数据后获得的过滤数据都在95%以上,其中Phred数值大于20、30的碱基占总体碱基的百分比分别超过95%和91%(表2)。
表 2 测序数据和测序质量的统计分析Table 2. Statistical analysis on sequencing data and quality样本
Sample原始数据
Raw base/bp过滤数据
Clean base/bp比例
Rate/%Q20/% Q30/% GC/% w14 7 057 752 750 6 731 469 250 95.38 96.10 91.15 44.27 隐性基因池 Recessive gene pool 12 841 555 750 12 418 982 750 96.71 96.00 91.04 44.43 显性基因池 Dominant gene pool 9 239 019 250 8 888 096 500 96.20 95.96 91.00 42.56 注: Q20及Q30:Phred数值大于20、30的碱基占总体碱基的百分比,其中,Phred=-10log10(e),e为错误率。
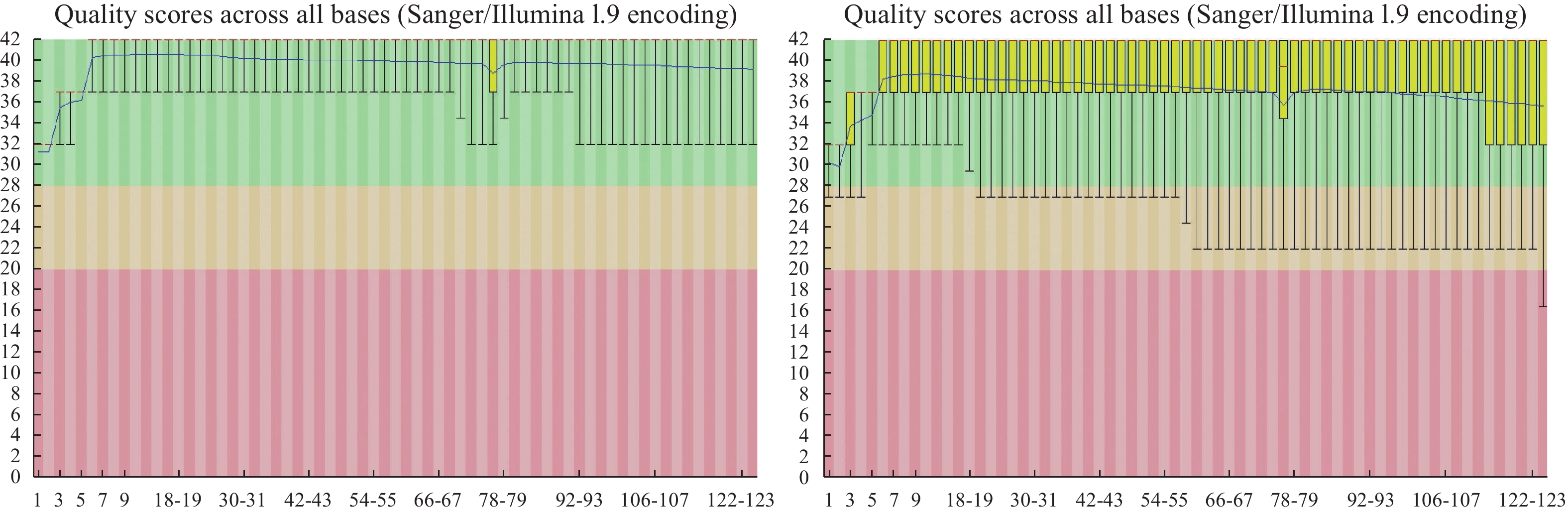
Note: Q20 and Q30: percentages of bases with Phred values greater than 20 and 30, respectively, in total number of bases; Phred=-10log10(e); e: error rate.在测序质量评估上,以样本w14的测序结果为例,Qphred值的箱线图(zip文件中Images/per_base_quality.png)(图1),其中图1A是正向数据的Qphred值,图1B是反向数据的Qphred值。从图可知,测序的质量值总体在Q30以上,测序的质量值分布正常,可用于进一步的分析。
2.2 BSA-Seq突变位点分析
根据设计的BSA-seq分析的基因克隆方案,进行了单基因定位和突变基因的鉴定。对显性基因池和隐性基因池进行基因频率分析结果可知,单基因主峰位于Chr3(图2)。进一步对Chr3基因统计量拟合曲线分析,候选区间初步估计为Chr3:27.5~29.5 Mb(图3)。这包含了我们前述的利用传统分子标记技术所定位的区间,并形成相互验证的结果。但为保守起见,上下游各扩0.5 Mb,即挑选Chr3的27 Mb至30 Mb的范围进行因果变异的鉴定。
综合精细定位的区间,在目标区域内共找到2个符合条件的候选因果变异(表3)。并且利用snpEff对候选因果变异进行注释,注释结果如表4所示,在Chr3上的两个候选基因中,其中LOC_Os03g48626位于染色体27726166的物理位置上,突变位点由野生型YY的C突变为T,但该突变发生在内含子中。另一候选基因LOC_Os03g49210位于Chr3的28129631物理位置上,突变位点也是由野生型的C突变为T,且该突变位于候选基因的第3外显子上,属于错义突变,造成了该基因编码的第20个氨基酸由T(苏氨酸)变成I(异亮氨酸),从而可能导致了基因功能的改变。遂将该基因定位本研究的目标基因,因与人BRCA1同源,命名为OsBRCA1。
表 3 目标染色体的因果变异鉴定结果Table 3. Causal variant identification on target chromosome染色体
Chromosome物理位置
Physical location野生型
Wild type隐性池
Hidden pool显性池
Dominant pool野生型参考基因组
WT-IRGSP5Chr3 27726166 1/1∶8∶1∶7 1/1∶41∶0∶41 0/1∶25∶19∶6 0/0∶1∶1∶0 Chr3 28129631 0/1∶11∶2∶9 0/1∶39∶2∶37 0/1∶31∶20∶11 0/0∶13∶13∶0 注:表中数据0/0表示参考基因组序列的纯合,1/1表示变异序列的纯合。
Note: * 0/0 and 1/1 represent homozygous of reference and variant genome sequence, respectively.表 4 利用snpEff对候选因果变异进行注释的结果Table 4. Annotated candidate causal variant by snpEff差异位点
Variable site差异位点1
Variable site 1差异位点2
Variable site 2物理位置 Physical location 27726166 28129631 突变基因 Mutant gene LOC_Os03g48626 LOC_Os03g49210 变异结果 Consequence 变异发生在内含子中 错义突变aCc(T) → aTc(I)由于C变异为T,导致第20个氨基酸T变为I 基因功能注释 Functional annotation 表达蛋白 含有BRCA1 C末端结构域的蛋白质 2.3 突变基因差异位点验证
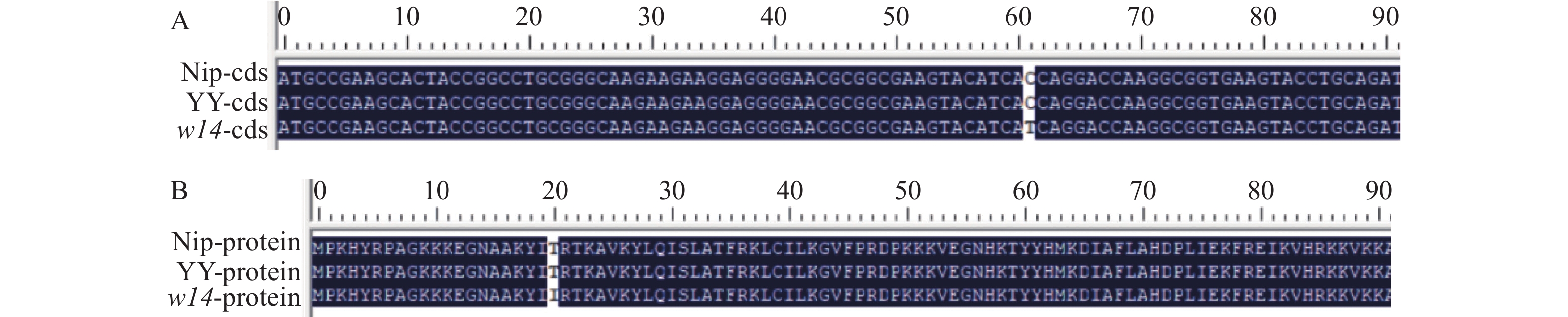
根据BSA-Seq结果,通过数据库查找目标基因OsBRCA1的基因组序列,设计9对引物(表1)分别分段扩增突变体w14、野生型云引及日本晴的OsBRCA1基因组序列,以对BSA-Seq结果进行验证。电泳后回收片段进行DNA测序。调取NCBI数据库中水稻的LOC_Os03g49210序列,用DNAMAN软件进行分段比对,将测序结果拼接比对LOC_Os03g49210的CDS序列在日本晴、YY及w14中是否存在差异。对比结果表明在突变体w14中该基因CDS的第62位碱基处有一个SNP位点,由野生型YY的C突变成T(图4A),造成了其编码的氨基酸序列的第21位由苏氨酸突变为异亮氨酸(图4B)。该测序结果与上述BSA-seq分析结论相一致,这也进一步说明测序和分析结论的具有较高的可靠性。
3. 讨论与结论
水稻叶片的衰老直接影响水稻的产量和品质,水稻早衰的研究成为热点领域之一。当前,在水稻中已经鉴定了超过150个的衰老相关基因,这些早衰相关基因分布在除了第1、8和12染色体之外的其他9条染色体,其中已被克隆的仅占三分之一。与前人研究相比较,本研究中的突变体w14的表型与已报道的早衰突变体均不同。在前期工作中,我们通过传统图位克隆方法,获得了早衰突变体w14的目标基因精细定位区间,但该区间内包含有8个功能基因,因此无法最终确定其候选基因[12]。为了进一步确定早衰突变体的目标候选基因,同时也对图位克隆的结果进行验证,本研究中我们通过BSA-Seq的方法从中寻找可能影响水稻出现早衰突变的位点,并最终在目标区域内找到2个符合条件的候选因果变异,其中LOC_Os03g48626该突变发生在内含子中,另一候选基因LOC_Os03g49210的突变位于第2外显子上,属于错义突变,造成该基因编码的氨基酸发生改变。BSA-seq分析结论与传统图位克隆方法所得到的结果相一致,这也进一步说明BSA-seq及分析结论具有较高的可靠性。错义突变基因与人BRCA1同源,命名为OsBRCA1,该基因为一个保守基因,目前已在13650多个物种上被发现,在水稻中尚未见该基因的相关报道。在拟南芥中也存在BRCA1的同源基因,AtBRCA1,包含一个N端环状结构域,两个C端BRCT和p300/CBP互作结构域,其结构域与分子特性都与BRCA1高度相似,极易受伽马射线诱导,在DNA修复和细胞周期控制中起重要作用[16] 。拟南芥中存在两个BRCA1
的互作基因即AtBRCC36A和AtBRCC36B,这两个基因功能同时存在或同时敲除时,拟南芥都是完全可育的且并无发育缺陷,而单突变体则具有明显的HR缺陷[17] 。BRCA2 同源基因不仅参与减数分裂中DNA重组,还通过与链交换蛋白RAD51的互作参与病原相关(PR)基因的转录调控[18] 。暴露在X射线遭受高剂量辐射时,拟南芥中BRCA1和RAD51在慢阶段DNA损伤修复过程中表达滞后[19] ,从而影响植物的生长发育。因此OsBRCA1的突变也可能造成水稻DNA复制时的错配、双链DNA断裂和DNA损伤修复受阻,从而引起水稻的发育异常,导致水稻的叶片早衰症状。 -
表 1 用于 OsBRCA1基因组全长分段扩增的引物序列
Table 1 Primer sequences for genome full-length fragment amplification on OsBRCA1
序号
Number标记
Marker上游引物
Forward primer(5′-3′)下游引物
Reverse primer(5′-3′)1 49210-1 AATACCATATCGCCGTTTTCTT AACTTTTCCATCACATCGTTCTAA 2 49210-2 TTGCCCGCATAATGTGACTG AAGACTGGAGACTTGGGAGGTG 3 49210-3 TTTGCTATGCTGAACTCCCG TCCCTTCCACCTTCTTCTTTG 4 49210-4 GATATGTTGGGTCATTTTGGAGC TCAAGTTTGTAAGTTGGTGGTCG 5 49210-5 CAAGGTCCACAGGAAGAAGGT AAGATTGTTGCACTCAAAGAAATG 6 49210-6 CTGGCTAACTCCACATGCTCTT TTCCACTGGCCTACCTCACG 7 49210-7 CCAACTAATGAACCTGGAGCG CAATATGTGCAACCAAAATGTGAA 8 49210-8 GGGCAGCCTTCACTAATGACA TTACGCTGAAACAGGGAAATG 9 49210-9 GCTGAGGATGGAATACGAAGGA GGTGGAGGGAAATCGAGGAG 表 2 测序数据和测序质量的统计分析
Table 2 Statistical analysis on sequencing data and quality
样本
Sample原始数据
Raw base/bp过滤数据
Clean base/bp比例
Rate/%Q20/% Q30/% GC/% w14 7 057 752 750 6 731 469 250 95.38 96.10 91.15 44.27 隐性基因池 Recessive gene pool 12 841 555 750 12 418 982 750 96.71 96.00 91.04 44.43 显性基因池 Dominant gene pool 9 239 019 250 8 888 096 500 96.20 95.96 91.00 42.56 注: Q20及Q30:Phred数值大于20、30的碱基占总体碱基的百分比,其中,Phred=-10log10(e),e为错误率。
Note: Q20 and Q30: percentages of bases with Phred values greater than 20 and 30, respectively, in total number of bases; Phred=-10log10(e); e: error rate.表 3 目标染色体的因果变异鉴定结果
Table 3 Causal variant identification on target chromosome
染色体
Chromosome物理位置
Physical location野生型
Wild type隐性池
Hidden pool显性池
Dominant pool野生型参考基因组
WT-IRGSP5Chr3 27726166 1/1∶8∶1∶7 1/1∶41∶0∶41 0/1∶25∶19∶6 0/0∶1∶1∶0 Chr3 28129631 0/1∶11∶2∶9 0/1∶39∶2∶37 0/1∶31∶20∶11 0/0∶13∶13∶0 注:表中数据0/0表示参考基因组序列的纯合,1/1表示变异序列的纯合。
Note: * 0/0 and 1/1 represent homozygous of reference and variant genome sequence, respectively.表 4 利用snpEff对候选因果变异进行注释的结果
Table 4 Annotated candidate causal variant by snpEff
差异位点
Variable site差异位点1
Variable site 1差异位点2
Variable site 2物理位置 Physical location 27726166 28129631 突变基因 Mutant gene LOC_Os03g48626 LOC_Os03g49210 变异结果 Consequence 变异发生在内含子中 错义突变aCc(T) → aTc(I)由于C变异为T,导致第20个氨基酸T变为I 基因功能注释 Functional annotation 表达蛋白 含有BRCA1 C末端结构域的蛋白质 -
[1] LIM P O, KIM H J, NAM H G. Leaf senescence [J]. Annual Review of Plant Biology, 2007, 58: 115−136. DOI: 10.1146/annurev.arplant.57.032905.105316
[2] BECK C I, ULRICH T H. Environmental release permits [J]. Bio/Technology, 1993, 11(12): 1524−1528.
[3] SWAMINATHAN M S. Towards a hunger-free India [J]. The Indian Journal of Nutrition and Dietetics, 1999, 36(4): 108−117.
[4] DAI L Y, LIU X L, XIAO Y H, et al. Recent advances in cloning and characterization of disease resistance genes in rice [J]. Journal of Integrative Plant Biology, 2007, 49(1): 112−119. DOI: 10.1111/j.1744-7909.2006.00413.x
[5] PROJECT I R G S. The map-based sequence of the rice genome [J]. Nature, 2005, 436(7052): 793−800. DOI: 10.1038/nature03895
[6] YOSHIDA S. Molecular regulation of leaf senescence [J]. Current Opinion in Plant Biology, 2003, 6(1): 79−84. DOI: 10.1016/S1369526602000092
[7] 王备芳, 陈玉宇, 张迎信, 等. 水稻早衰突变体es5的鉴定及其突变基因的精细定位 [J]. 中国农业科学, 2018, 51(4):613−625. DOI: 10.3864/j.issn.0578-1752.2018.04.002 WANG B F, CHEN Y Y, ZHANG Y X, et al. Identification and fine mapping of an early senescent leaf mutant Es5 in Oryza sativa L [J]. Scientia Agricultura Sinica, 2018, 51(4): 613−625.(in Chinese) DOI: 10.3864/j.issn.0578-1752.2018.04.002
[8] SAKURABA Y, RAHMAN M L, CHO S H, et al. The rice faded green leaf locus encodes protochlorophyllide oxidoreductase B and is essential for chlorophyll synthesis under high light conditions [J]. The Plant Journal:for Cell and Molecular Biology, 2013, 74(1): 122−133. DOI: 10.1111/tpj.12110
[9] LIN A H, WANG Y Q, TANG J Y, et al. Nitric oxide and protein S-nitrosylation are integral to hydrogen peroxide-induced leaf cell death in rice [J]. Plant Physiology, 2011, 158(1): 451−464.
[10] LIANG C Z, WANG Y Q, ZHU Y N, et al. OsNAP connects abscisic acid and leaf senescence by fine-tuning abscisic acid biosynthesis and directly targeting senescence-associated genes in rice [J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(27): 10013−10018. DOI: 10.1073/pnas.1321568111
[11] KONG Z S, LI M N, YANG W Q, et al. A novel nuclear-localized CCCH-type zinc finger protein, OsDOS, is involved in delaying leaf senescence in rice [J]. Plant Physiology, 2006, 141(4): 1376−1388. DOI: 10.1104/pp.106.082941
[12] 朱永生, 蒋家焕, 蔡秋华, 等. 水稻早衰突变体w14的生理学特性分析及其基因的精细定位 [J]. 科学通报, 2021, 66(32):4144−4156. DOI: 10.1360/TB-2021-0012 ZHU Y S, JIANG J H, CAI Q H, et al. Analysis of physiological characteristics of early leaf senescence mutant w14 and its gene mapping for rice [J]. Chinese Science Bulletin, 2021, 66(32): 4144−4156.(in Chinese) DOI: 10.1360/TB-2021-0012
[13] 梁廷敏, 郭新睿, 陈子强, 等. 水稻材料IR65482抗稻瘟病基因鉴定与定位 [J]. 分子植物育种, 2018, 16(13):4308−4313. LIANG T M, GUO X R, CHEN Z Q, et al. Identification and mapping of a blast disease resistance gene in rice line IR65482 [J]. Molecular Plant Breeding, 2018, 16(13): 4308−4313.(in Chinese)
[14] SUN J, YANG L M, WANG J G, et al. Identification of a cold-tolerant locus in rice (Oryza sativa L. ) using bulked segregant analysis with a next-generation sequencing strategy [J]. Rice (New York, N Y ), 2018, 11(1): 24.
[15] SALUNKHE A S, POORNIMA R, PRINCE K S J, et al. Fine mapping QTL for drought resistance traits in rice (Oryza sativa L. ) using bulk segregant analysis [J]. Molecular Biotechnology, 2011, 49(1): 90−95. DOI: 10.1007/s12033-011-9382-x
[16] LAFARGE S, MONTANÉ M H. Characterization of Arabidopsis thaliana ortholog of the human breast cancer susceptibility gene 1: AtBRCA1, strongly induced by gamma rays [J]. Nucleic Acids Research, 2003, 31(4): 1148−1155. DOI: 10.1093/nar/gkg202
[17] BLOCK-SCHMIDT A S, DUKOWIC-SCHULZE S, WANIECK K, et al. BRCC36A is epistatic to BRCA1 in DNA crosslink repair and homologous recombination in Arabidopsis thaliana [J]. Nucleic Acids Research, 2010, 39(1): 146−154.
[18] TRAPP O, SEELIGER K, PUCHTA H. Homologs of breast cancer genes in plants [J]. Frontiers in Plant Science, 2011, 2: 19.
[19] EINSET J, COLLINS A R. DNA repair after X-irradiation: Lessons from plants [J]. Mutagenesis, 2014, 30(1): 45−50.
-
期刊类型引用(3)
1. 苗龙,舒阔,李娟,黄茹,王业杏,Soltani Muhammad YOUSOF,许竞好,吴传磊,李佳佳,王晓波,邱丽娟. 大豆根茎过渡区弯曲突变体M_(rstz)的鉴定与基因定位. 作物学报. 2024(05): 1091-1103 .  百度学术
百度学术
2. 刘继虎,于雪然,刘爱婷,徐强,马静,姚鑫杰,张哲,李培富. 粳稻种质资源粒长全基因组关联分析及候选基因筛选. 种子. 2024(10): 98-103 .  百度学术
百度学术
3. 赵宇晶,张滨烁,苏安玉,于振海,李佳欢,林洋,张艳婷,武小霞,赵莹. 基于BSA-seq方法挖掘大豆再生相关候选基因. 作物学报. 2023(11): 2935-2948 .  百度学术
百度学术
其他类型引用(7)




 下载:
下载: