Mitochondrial Genome and Phylogenetics of Philonthus spinipes (Sharp, 1874)
-
摘要:目的 探究大赤隐翅虫Philonthus spinipes(Sharp, 1874)的线粒体基因组结构特征及隐翅虫亚科的系统发育关系。方法 利用高通量测序技术获得大赤隐翅虫线粒体基因组的全序列。在系统发育分析中,选择隐翅虫亚科的21个物种和毒隐翅虫亚科的8个物种作为内群,并选择2个蚁甲亚科Pselaphinae的物种作为外群,利用最大似然法和贝叶斯法重建隐翅虫亚科的系统发育关系。结果 大赤隐翅虫的线粒体基因组包含37个基因(13个蛋白质编码基因、22个tRNA基因和2个rRNA基因)和一段非编码控制区。整个线粒体基因组全长为16 219 bp(GenBank登录号:OL998729)。大赤隐翅虫线粒体基因组的13个蛋白质编码基因的起始密码子除nad6和nad1利用ATC和TTG开头外,其余蛋白质编码基因都是以ATT、ATA和ATG开头。4个蛋白质编码基因(cox1、cox2、cox3和nad5)以不完整的终止密码子T或TA结尾,其余9个蛋白质编码基因以完整的终止密码子TAA或TAG结尾。除trnS1因缺少DHU臂而形成一个简单的环,无法形成稳定的三叶草结构外,其余tRNA基因均能形成典型的三叶草结构。rrnL基因的全长为1 275 bp,A+T含量为79.84%。rrnS基因全长为765 bp,A+T含量为76.47%。结论 两种不同的系统发育分析方法构建的隐翅虫亚科的系统发育关系是基本一致的,均表明隐翅虫亚科为非单系群;毒隐翅虫亚科为单系群;毒隐翅虫亚科嵌套在隐翅虫亚科中。Abstract:Objective Mitochondrial genome and phylogenetic relationship in Staphylininae subfamily of Philonthus spinipes (Sharp, 1874) were determined.Method The high-throughput sequencing technique was applied to secure the full length mitochondrial genome of P. spinipes. A phylogenetic analysis was conducted to decipher the beetle’s relationship in the Staphylininae subfamily. Using 21 exemplars of Staphylininae and 8 of Paederinae as ingroups as well as two Pselaphinae species as outgroups, the phylogeny was reconstructed with the maximum likelihood (ML) and Bayesian inference (BI) methods.Result The mitochondrial genome of P. spinipes was a circular molecule of 16 219 bp with the GenBank accession number of OL998729. It contained 13 protein-coding genes, 22 transfer RNA genes, two ribosomal RNA genes, and a non-coding control region. Most of the protein-coding genes started with ATT, ATA, or ATG, but nad6 did with ATC and nad1 with TTG. And cox1, cox2, cox3, and nad5 had an incomplete T or TA as the stop codon, while the remaining 9 genes terminated with TAA or TAG. All tRNA genes, except trnS1 which lacked the DHU arm, could be folded into a typical cloverleaf-like structure. The lengths of rrnL and rrnS were 1 275 bp and 765 bp with the A+T contents of 79.84% and 76.47%, respectively.Conclusion The two applied phylogenetic inference methods produced a similar tree topological structure that showed Paederinae to be monophyletic, Staphylininae non-monophyletic, and Paederinae nested within the Staphylininae subfamily.
-
Keywords:
- Coleoptera /
- Staphylininae /
- Philonthus spinipes /
- mitochondrial genome /
- phylogeny
-
0. 引言
【研究意义】大赤隐翅虫Philonthus spinipe (Sharp 1874)属于鞘翅目Coleoptera隐翅虫科Staphylinidae隐翅虫亚科Staphylininae。隐翅虫亚科是隐翅虫科中种群数量较多、物种较为丰富的一个类群。目前,隐翅虫亚科有350属,超过8 700个现存的已知种,为隐翅虫科第三大类群[1-2]。由于隐翅虫亚科的一些物种具有鲜艳的体色而经常被昆虫爱好者所收集。因此,对隐翅虫亚科的系统发育学研究有助于更好地了解其系统发育关系。【前人研究进展】传统分类中,隐翅虫亚科被分类学家广泛认为是一个自然群[3-7]。因为这类甲虫的成虫、幼虫和蛹具有独特的形态学特征。并且,一些基于分子数据的研究支持隐翅虫亚科的单系性[8-10]。然而,也有一些分子系统研究不支持隐翅虫亚科的单系性[11, 12]。此外,BRUNKE等基于6个基因片段的系统发育分析不支持隐翅虫亚科的单系性,因为隐翅虫亚科的一些族被嵌入到毒隐翅虫亚科中[11]。【本研究切入点】作为一种分子遗传标记,昆虫的线粒体基因组因其具有结构简单以及相对保守的基因组组织结构等特点,被广泛应用于昆虫系统学研究中[13-15]。但是,关于大赤隐翅虫的线粒体基因组研究仍未有报道。截至目前,在NCBI的GenBank中仅有37条部分或完整的隐翅虫亚科昆虫的线粒体基因组序列被公布。有限的线粒体基因组序列阻碍了人们从基因组角度研究隐翅虫亚科昆虫的系统发育关系。【拟解决的关键问题】本研究利用高通量测序技术获得大赤隐翅虫P. spinipes的线粒体基因组全序列,并对其线粒体基因组结构特点进行分析,构建线粒体基因组中的37个线粒体基因序列矩阵、13个蛋白质编码基因的核苷酸序列矩阵、13个蛋白质编码基因的第1、2密码子的核苷酸序列矩阵。结合已经公布的隐翅虫亚科的21个物种和毒隐翅虫亚科的8个物种的线粒体基因组序列,并选择蚁甲亚科Pselaphinae的两个物种作为外群,利用最大似然法(Maximum likelihood)和贝叶斯法(Bayesian infernece)重建隐翅虫亚科的系统发育关系,以期为研究隐翅虫亚科的系统发育关系提供线粒体基因组数据。
1. 材料与方法
1.1 标本采集与DNA提取
大赤隐翅虫标本于2019年7月采集于河南省郑州市人民公园(34°72′N, 114°01′E)。将采集到的昆虫标本浸泡在95%的乙醇中,并置于−20 ℃冰箱保存。使用天根公司的动物基因组DNA提取试剂盒(TIANamp Genomic DNA Kit,China)提取大赤隐翅虫样本的总DNA。经NanoDrop 2000分光光度计和1%的琼脂糖凝胶电泳进行总DNA的质量和浓度检测。
1.2 高通量测序与线粒体基因组组装
将检测质量合格且质量浓度大于20 ng·μL-1的DNA送往上海派森诺生物科技有限公司进行高通量测序。首先,利用全基因组鸟枪法(Whole Genome Shotgun,WGS)构建文库,并基于Illumina HiSeq 2500测序平台进行双端(2 × 150 bp PE)测序。最后,将下机的原始数据去除接头,进行质控。利用GetOrganelle v1.7.5.2软件[16]进行线粒体基因组的组装和拼接,从而得到高质量的线粒体基因组序列。
1.3 线粒体基因组的注释和分析
将组装得到的大赤隐翅虫的线粒体基因组序列在MITOS[17]网站上进行初步的注释,Genetic Code参数选择为05-inverterbrate,其余均按照MITOS默认参数进行设置。22个tRNA和两个RNA的二级结构通过MITOS预测获得。然后,通过序列比对,手动矫正13个蛋白质编码基因和2个RNA基因的基因边界。将注释好的线粒体基因组序列上传至GenBank,得到大赤隐翅虫的GenBank登录号:OL998729。利用MEGA 7.0[18]软件分析大赤隐翅虫线粒体基因组13个蛋白质编码基因的碱基组成以及密码子的使用频率。
1.4 多序列比对
利用TranslatorX程序中的MAFFT软件分别对不同的线粒体基因的核苷酸序列矩阵进行比对[19-20]。用trimAl v1.4对序列进行修剪[21],将比对模糊的片段删除。最后,使用FASconCAT-G_v1.04进行串联得到矩阵[22]。
1.5 系统发育分析
本研究使用测序获得大赤隐翅虫线粒体基因组的37个基因序列、13个蛋白质编码基因的核苷酸序列、13个蛋白质编码基因的第1、2密码子的核苷酸序列,结合NCBI中已经公布的隐翅虫亚科的21个物种和毒隐翅虫亚科的8个物种作为内群,Pselaphinae的2个物种作为外群构建数据矩阵。利用最大似然法(Maximum likelihood)和贝叶斯法(Bayesian inference)重建隐翅虫亚科的系统发育树。利用 IQ-TREE 2.0.6[23] 进行最大似然法分析:选择GTR+F+I+G4模型,系统发育树的节点支持率通过自举检验置信度进行评估(运行次数设置为10000)。使用PhyloBayes v3.3 进行贝叶斯法分析[24]。首先,执行运算两次,每次包含4个链。使用CAT-GTR位点异质模型来进行分析,利用PhyloBayes软件包中的bpcomp程序评估链间的趋同一致性。以最大差异值小于0.1作为分析达到有效的判定标准。最后,使用bpcomp程序构建50%合意树。
2. 结果与分析
2.1 线粒体基因组分析
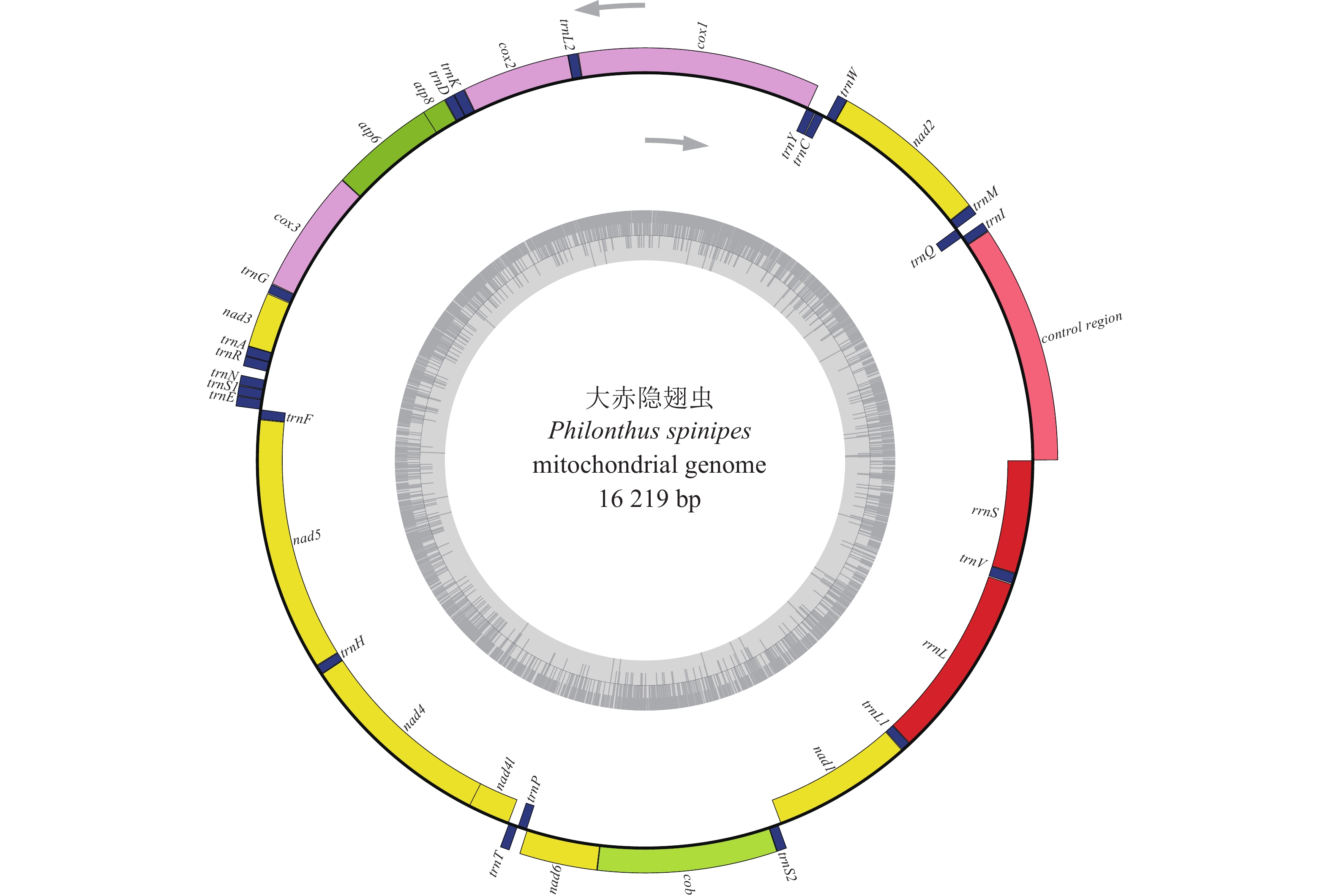
利用OGDRAW Version 1.1[25]绘制大赤隐翅虫的线粒体基因组结构图(图1)。大赤隐翅虫的线粒体基因全长16 219 bp,其中A+T含量为73.56%,G+C含量为26.44%。该线粒体基因组包含37个基因和1个非编码控制区(表1)。
表 1 大赤隐翅虫线粒体基因组注释Table 1. Annotation of P. spinipes mitochondrial genome基因
Gene基因长度
Gene length/bp起始位置
Start position/bp终止位置
Stop position/bp起始密码子
Start codon终止密码子
Stop codon编码链
Coding strand控制区Control region 1 521 1 1 521 非编码序列Non-coding sequence trnI 64 1 522 1 585 H trnQ 69 1 651 1 583 L trnM 69 1 651 1 719 H nad2 1 017 1 720 2 736 ATT TAA H trnW 67 2 735 2 801 H trnC 62 2 878 2 817 L trnY 64 2 944 2 881 L cox1 1 540 2 937 4 476 ATT T H trnL2 65 4 477 4 541 H cox2 688 4 540 5 227 ATG T H trnK 71 5 228 5 298 H trnD 66 5 298 5 363 H atp8 156 5 364 5 519 ATA TAA H atp6 669 5 513 6 181 ATG TAA H cox3 788 6 181 6 968 ATG TA H trnG 61 6 970 7 030 H nad3 354 7 033 7 386 ATT TAG H trnA 65 7 385 7 449 H trnR 66 7 449 7 514 H trnN 66 7 571 7 636 H trnS1 66 7 637 7 702 H trnE 68 7 703 7 770 H trnF 67 7 835 7 769 L nad5 1 717 9 552 7 836 ATT T L trnH 65 9 617 9 553 L nad4 1 338 10 954 9 617 ATG TAA L nad4l 288 11 235 10 948 ATG TAA L trnT 63 11 238 11 300 H trnP 64 11 364 11 301 L nad6 498 11 366 11 863 ATC TAA H Cob 1 143 11 863 13 005 ATG TAG H trnS2 66 13 004 13 069 H nad1 951 14 037 13 087 TTG TAG L trnL1 65 14 103 14 039 L rrnL 1 275 15 378 14 104 L trnV 73 15 454 15 382 L rrnS 765 16 219 15 455 L H:重链;L:轻链;T或TA表示不完全密码子。
H: Heavy strand; L: light strand; T or TA: incomplete stop codon.在新测定的线粒体基因组中,H链(Heavy strand)编码23个基因,包括9个蛋白质编码基因,14个tRNA基因;L链(Light strand)编码14个基因,包含4个蛋白质编码基因,8个tRNA基因和2个rRNA基因。非编码控制区位于rrnS和trnI之间,长度1 521 bp,其A+T含量为78.83%。
2.2 蛋白质编码基因分析
大赤隐翅虫线粒体基因组的13个蛋白质编码基因全长为10 410 bp,其中A+T含量为71.78%,G+C含量为28.22%。对于13个蛋白质编码基因的起始密码子,除nad6和nad1利用ATC和TTG开头外,其余11个蛋白质编码基因都是以ATT、ATA和ATG开头。4个蛋白质编码基因,即cox1、cox2、cox3和nad5是以不完整的终止密码子T或TA结尾,其余9个蛋白质编码基因以完整的终止密码子TAA或TAG结尾。在所有蛋白质编码基因的氨基酸使用频率中,丙氨酸(Ala)为1.31%、半胱氨酸(Cys)为2.67%、天冬氨酸(Asp)为2.48%、谷氨酸(Glu)为2.76%、苯丙氨酸(Phe)为11.20%、甘氨酸(Gly)为2.67%、组氨酸(His)为2.36%、异亮氨酸(Ile)为9.36%、赖氨酸(Lys)为4.08%、亮氨酸(Leu)为13.96%、甲硫氨酸(Met)为5.46%、天冬酰胺(Asn)为6.29%、脯氨酸(Pro)为3.92%、谷氨酰胺(Gln)为2.27%、精氨酸(Arg)为1.22%、丝氨酸(Ser)为11.20%、苏氨酸(Thr)为4.05%、缬氨酸(Val)为2.94%、色氨酸(Trp)为2.82%、酪氨酸(Thr)为9.04%。13个蛋白质编码基因的相对密码子使用频率见表2。
表 2 大赤隐翅虫线粒体基因组13个蛋白质编码基因的相对密码子使用频率Table 2. Relative synonymous codon usage of 13 protein-coding genes of P. spinipes mitochondrial genome密码子
Codon次数
CountRSCU 密码子
Codon次数
CountRSCU 密码子
Codon次数
CountRSCU 密码子
Codon次数
CountRSCU UUU(F) 280 1.53 UCU(S) 72 1.58 UAU(Y) 173 1.54 UGU(C) 51 1.17 UUC(F) 85 0.47 UCC(S) 42 0.92 UAC(Y) 52 0.46 UGC(C) 36 0.83 UUA(L) 185 2.44 UCA(S) 73 1.6 UAA(*) 127 1.23 UGA(W) 60 1.3 UUG(L) 61 0.8 UCG(S) 22 0.48 UAG(*) 80 0.77 UGG(W) 32 0.7 CUU(L) 78 1.03 CCU(P) 57 1.78 CAU(H) 59 1.53 CGU(R) 12 1.2 CUC(L) 38 0.5 CCC(P) 34 1.06 CAC(H) 18 0.47 CGC(R) 4 0.4 CUA(L) 53 0.7 CCA(P) 30 0.94 CAA(Q) 51 1.38 CGA(R) 16 1.6 CUG(L) 40 0.53 CCG(P) 7 0.22 CAG(Q) 23 0.62 CGG(R) 8 0.8 AUU(I) 240 1.57 ACU(T) 54 1.64 AAU(N) 159 1.55 AGU(S) 36 0.79 AUC(I) 65 0.43 ACC(T) 33 1 AAC(N) 46 0.45 AGC(S) 27 0.59 AUA(M) 122 1.37 ACA(T) 39 1.18 AAA(K) 99 1.49 AGA(S) 46 1.01 AUG(M) 56 0.63 ACG(T) 6 0.18 AAG(K) 34 0.51 AGG(S) 47 1.03 GUU(V) 35 1.46 GCU(A) 17 1.58 GAU(D) 60 1.48 GGU(G) 17 0.78 GUC(V) 15 0.63 GCC(A) 11 1.02 GAC(D) 21 0.52 GGC(G) 13 0.6 GUA(V) 26 1.08 GCA(A) 12 1.12 GAA(E) 56 1.24 GGA(G) 33 1.52 GUG(V) 20 0.83 GCG(A) 3 0.28 GAG(E) 34 0.76 GGG(G) 24 1.1 RSCU:相对密码子使用频率; *终止密码子。
RSCU: Relative synonymous codon usage; *: stop codon.2.3 tRNA和rRNA基因分析
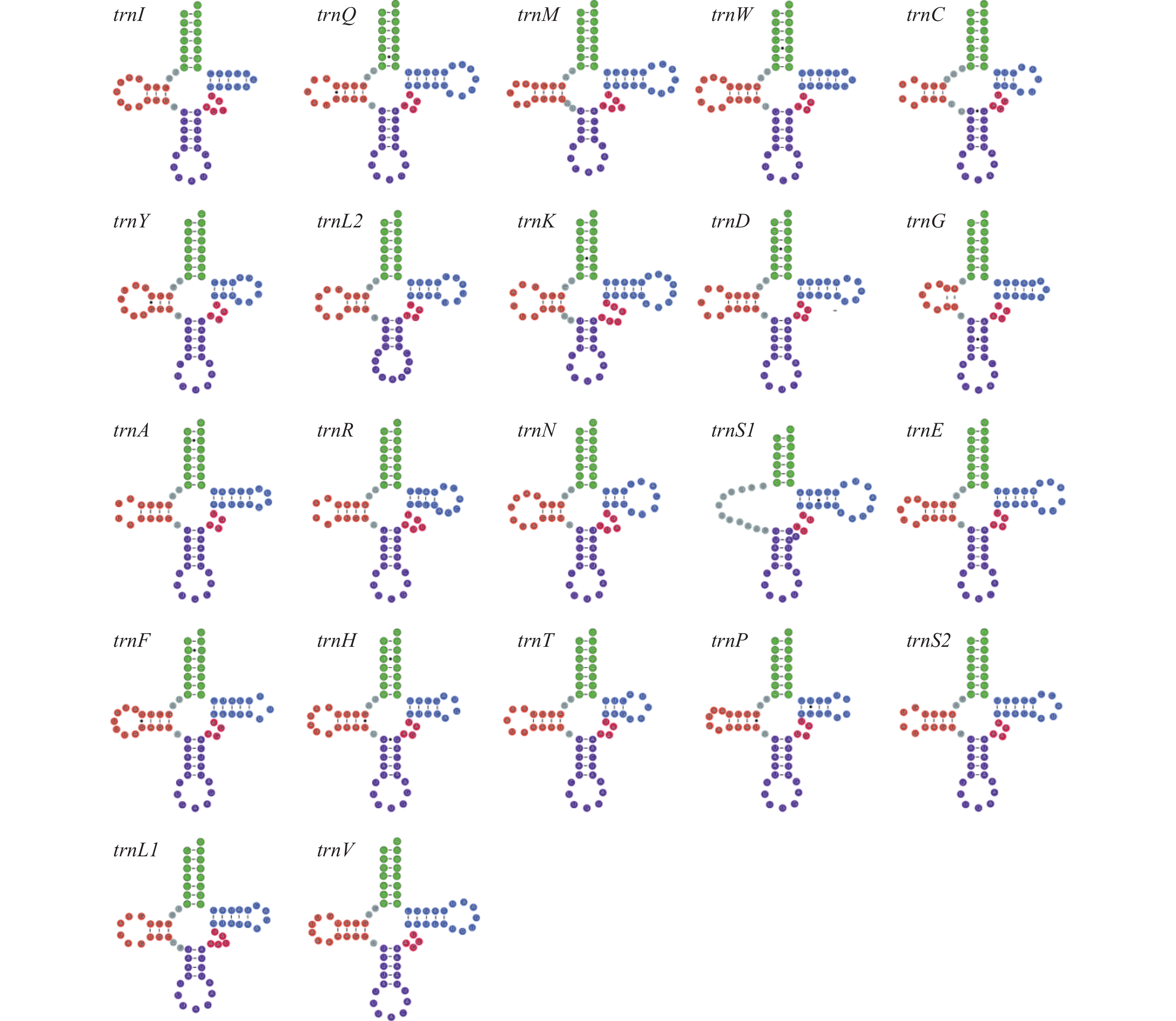
大赤隐翅虫线粒体基因组的22个tRNA基因的序列长度为61~73 bp。trnG基因最短(61 bp),trnV 基因最长(73 bp)。其中14个tRNA基因位于H链,8个tRNA基因位于L链, rrnL和rrnS基因位于L链。除trnS1因缺少DHU臂而形成一个简单的环,无法形成稳定的三叶草结构外,其余tRNA基因均能形成典型的三叶草结构(图2)。此外,trnS1 的反密码子不是常见的GCU,而是UCU。大赤隐翅虫线粒体的线粒体基因组rrnL的全长为1 275 bp,A+T含量为79.84%,G+C含量为20.16%。rrnS的全长为765 bp,A+T含量为76.47%,G+C含量为23.53%。本研究参考果蝇的rrnL和rrnS基因的二级结构图,对大赤隐翅虫的两个核糖体基因的二级结构进行预测,详见图3和图4。 rrnL基因包含5个结构区域(I-II和IV-VI),共50个螺旋结构。rrnS基因有3个结构区域(I、II和III),由31个螺旋结构组成。
2.4 系统发育分析
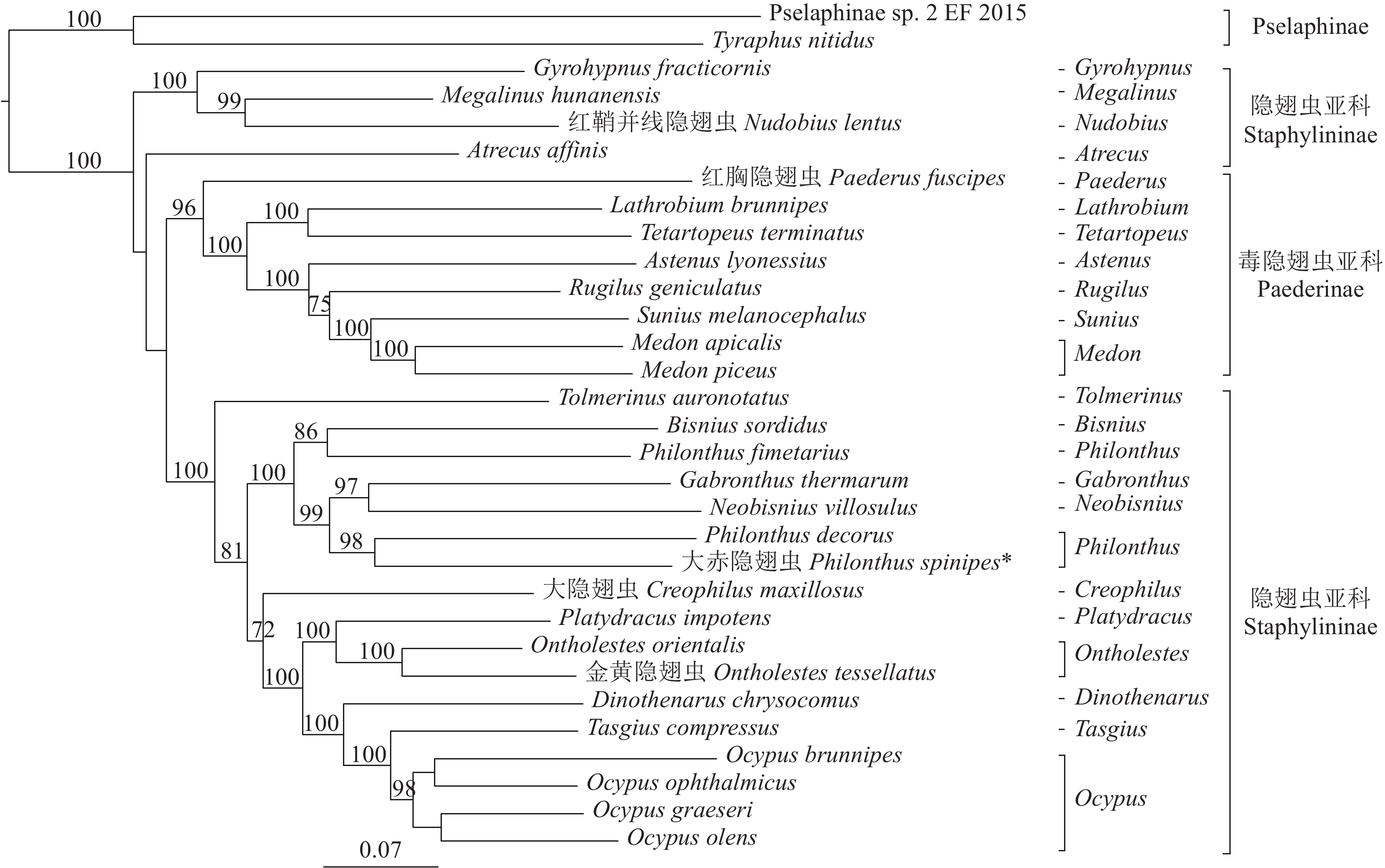
本研究结合已经公布的隐翅虫亚科的21个物种和毒隐翅虫亚科的8个物种作为内群以及蚁甲亚科Pselaphinae的2个物种作为外群,利用最大似然法和贝叶斯法,基于它们的线粒体基因组的37个基因的序列矩阵、13个蛋白质编码基因的核苷酸序列矩阵、13个蛋白质编码基因的第1、2密码子的核苷酸序列矩阵重建隐翅虫亚科的系统发育关系(图5~6)。两种系统发育分析方法构建的树拓扑结构都支持隐翅虫亚科为非单系群,毒隐翅虫亚科为单系群。毒隐翅虫亚科嵌套在隐翅虫亚科中。在最大似然法分析中,毒隐翅虫亚科的单系性也得到了很好的支持(BS = 95)。此外,在最大似然法分析中,本研究测定的大赤隐翅虫与P. decorus构成姐妹群,节点支持率最高(BS = 98)。在贝叶斯法分析中,线粒体基因组37个基因的序列矩阵与13个蛋白质编码基因的第1、2密码子的核苷酸序列矩阵发育树结果显示大赤隐翅虫与P. decorus与Neobisnius villosulus、 Gabronthus thermarum分别构成姐妹群。13个蛋白质编码基因的核苷酸序列矩阵的发育树结果显示大赤隐翅虫与P. decorus构成姐妹群。但是,贝叶斯法分析中关于大赤隐翅虫的姐妹群关系里只有线粒体基因组37个基因的序列矩阵发育树结果对大赤隐翅虫与N. villosulus、G. thermarum构成姐妹群有较高的节点支持率(PP=0.97),与P. decorus的姐妹群关系的节点支持率较低(PP<0.6)。其他两个矩阵的发育分析结果显示,大赤隐翅虫与P. decorus与N. villosulus、 G. thermarum分别构成姐妹群的节点支持率也都较低(PP<0.6)。
3. 讨论与结论
本研究利用高通量测序技术获得了大赤隐翅虫的线粒体基因组的完整序列。大赤隐翅虫线粒体基因组的37个基因的排列顺序与典型的昆虫线粒体基因组排列顺序一致[14],没有出现基因重排或基因丢失的现象。
虽然以前基于形态学特征和一些分子数据的系统发育研究恢复了隐翅虫亚科的单系性 [3-10],近期的一些研究却并不支持这一假设,还有研究显示隐翅虫亚科的一些族应该被转入到毒隐翅虫亚科类群中[11-12]。鞘翅目昆虫线粒体基因组是真核细胞中具有为细胞活动提供能量,参与细胞信息传递等特点,由37个相对保守基因构成的双链、环状的DNA,因其结构简单和进化速率慢等特点,常用于昆虫的系统发育研究中[26-30]。本研究基于线粒体基因组的37个基因的序列数据、13个蛋白质编码基因的核苷酸序列数据以及13个蛋白质编码基因的第1、2密码子的核苷酸序列数据的系统发育分析结果均显示隐翅虫亚科的4个属Atrecus、Gyrohypnus、Nudobius和Megalinus单独形成一支,导致隐翅虫亚科为非单系群。毒隐翅虫亚科嵌套在隐翅虫亚科中。这一结果与Brunke等和Schomann等的研究结果[11-12]一致。
Chatzimanolis等基于4个基因(cox1、wingless、Topoisomerase I和28S)片段,对隐翅虫亚科的49个物种和毒隐翅虫亚科的2个物种进行系统发育分析,结果支持隐翅虫亚科的单系性[9]。Mckenna等基于8个核基因序列(18S、28S、AK、AS、CAD、EF1-α、PEPCK 和 WG)对整个鞘翅目进行系统发育关系的重建,其分类取样也包含了隐翅虫亚科,结果支持该亚科为单系群[8]。Mckenna等利用28S和CAD两个基因序列对金龟下目Scarabaeiformia和隐翅虫下目Staphyliniformia进行系统发育关系的重建,其结果支持隐翅虫亚科和毒隐翅虫亚科的单系性[10]。以上研究结果与本研究对于隐翅虫亚科系统发育分析结果不同在于:Mckenna等基于核基因研究鞘翅目的高级系统发育关系为主要目的,虽然取样较多,但是对于隐翅虫亚科的取样有限;而本研究基于线粒体基因组数据,集中隐翅虫亚科和毒隐翅虫亚科的取样,着力解决这两者之间的相互关系。在两个研究中,不同的分子标记和不同的分类阶元取样可能导致隐翅虫亚科的系统发育结果不同。在今后的研究中,系统分类学者应该包含更多的隐翅虫亚科和毒隐翅虫亚科的昆虫样本以及更加综合的外群取样,结合形态学特征、转录组数据或基因组规模的数据等,以期获得接近或真实反映隐翅虫进化历史的系统发育关系。
-
表 1 大赤隐翅虫线粒体基因组注释
Table 1 Annotation of P. spinipes mitochondrial genome
基因
Gene基因长度
Gene length/bp起始位置
Start position/bp终止位置
Stop position/bp起始密码子
Start codon终止密码子
Stop codon编码链
Coding strand控制区Control region 1 521 1 1 521 非编码序列Non-coding sequence trnI 64 1 522 1 585 H trnQ 69 1 651 1 583 L trnM 69 1 651 1 719 H nad2 1 017 1 720 2 736 ATT TAA H trnW 67 2 735 2 801 H trnC 62 2 878 2 817 L trnY 64 2 944 2 881 L cox1 1 540 2 937 4 476 ATT T H trnL2 65 4 477 4 541 H cox2 688 4 540 5 227 ATG T H trnK 71 5 228 5 298 H trnD 66 5 298 5 363 H atp8 156 5 364 5 519 ATA TAA H atp6 669 5 513 6 181 ATG TAA H cox3 788 6 181 6 968 ATG TA H trnG 61 6 970 7 030 H nad3 354 7 033 7 386 ATT TAG H trnA 65 7 385 7 449 H trnR 66 7 449 7 514 H trnN 66 7 571 7 636 H trnS1 66 7 637 7 702 H trnE 68 7 703 7 770 H trnF 67 7 835 7 769 L nad5 1 717 9 552 7 836 ATT T L trnH 65 9 617 9 553 L nad4 1 338 10 954 9 617 ATG TAA L nad4l 288 11 235 10 948 ATG TAA L trnT 63 11 238 11 300 H trnP 64 11 364 11 301 L nad6 498 11 366 11 863 ATC TAA H Cob 1 143 11 863 13 005 ATG TAG H trnS2 66 13 004 13 069 H nad1 951 14 037 13 087 TTG TAG L trnL1 65 14 103 14 039 L rrnL 1 275 15 378 14 104 L trnV 73 15 454 15 382 L rrnS 765 16 219 15 455 L H:重链;L:轻链;T或TA表示不完全密码子。
H: Heavy strand; L: light strand; T or TA: incomplete stop codon.表 2 大赤隐翅虫线粒体基因组13个蛋白质编码基因的相对密码子使用频率
Table 2 Relative synonymous codon usage of 13 protein-coding genes of P. spinipes mitochondrial genome
密码子
Codon次数
CountRSCU 密码子
Codon次数
CountRSCU 密码子
Codon次数
CountRSCU 密码子
Codon次数
CountRSCU UUU(F) 280 1.53 UCU(S) 72 1.58 UAU(Y) 173 1.54 UGU(C) 51 1.17 UUC(F) 85 0.47 UCC(S) 42 0.92 UAC(Y) 52 0.46 UGC(C) 36 0.83 UUA(L) 185 2.44 UCA(S) 73 1.6 UAA(*) 127 1.23 UGA(W) 60 1.3 UUG(L) 61 0.8 UCG(S) 22 0.48 UAG(*) 80 0.77 UGG(W) 32 0.7 CUU(L) 78 1.03 CCU(P) 57 1.78 CAU(H) 59 1.53 CGU(R) 12 1.2 CUC(L) 38 0.5 CCC(P) 34 1.06 CAC(H) 18 0.47 CGC(R) 4 0.4 CUA(L) 53 0.7 CCA(P) 30 0.94 CAA(Q) 51 1.38 CGA(R) 16 1.6 CUG(L) 40 0.53 CCG(P) 7 0.22 CAG(Q) 23 0.62 CGG(R) 8 0.8 AUU(I) 240 1.57 ACU(T) 54 1.64 AAU(N) 159 1.55 AGU(S) 36 0.79 AUC(I) 65 0.43 ACC(T) 33 1 AAC(N) 46 0.45 AGC(S) 27 0.59 AUA(M) 122 1.37 ACA(T) 39 1.18 AAA(K) 99 1.49 AGA(S) 46 1.01 AUG(M) 56 0.63 ACG(T) 6 0.18 AAG(K) 34 0.51 AGG(S) 47 1.03 GUU(V) 35 1.46 GCU(A) 17 1.58 GAU(D) 60 1.48 GGU(G) 17 0.78 GUC(V) 15 0.63 GCC(A) 11 1.02 GAC(D) 21 0.52 GGC(G) 13 0.6 GUA(V) 26 1.08 GCA(A) 12 1.12 GAA(E) 56 1.24 GGA(G) 33 1.52 GUG(V) 20 0.83 GCG(A) 3 0.28 GAG(E) 34 0.76 GGG(G) 24 1.1 RSCU:相对密码子使用频率; *终止密码子。
RSCU: Relative synonymous codon usage; *: stop codon. -
[1] THAYER M K. Morphology and Systematics (Archostemata, Adephaga, Myxophaga, Polyphaga partim)[M]. Berlin: Walter de Gruyter, 2016: 394-442.
[2] CAI C Y, WANG Y L, LIANG L, et al. Congruence of morphological and molecular phylogenies of the rove beetle subfamily Staphylininae (Coleoptera: Staphylinidae) [J]. Scientific Reports, 2019, 9(1): 1−11. DOI: 10.1038/s41598-018-37186-2
[3] KASULE F K. The larvae of Paederinae and Staphylininae (Coleoptera: Staphylinidae) with keys to the known British Genera [J]. Transactions of the Royal Entomological Society of London, 2009, 122(2): 49−80. DOI: 10.1111/j.1365-2311.1970.tb00527.x
[4] NEWTON A F, THAYER M K. In American Beetles (Archostemata, Myxophaga, Adephaga, Polyphaga: Staphyliniformia)[M]. London: CRC Press, 2000: 272–418.
[5] GREBENNIKOV V V, NEWTON A F. Good-bye Scydmaenidae, or why the ant-like stone beetles should become megadiverse Staphylinidae sensu latissimo (Coleoptera) [J]. European Journal of Entomology, 2009, 106(2): 275−301. DOI: 10.14411/eje.2009.035
[6] NEWTON A F, THAYER M K, CHICAGO N N H M. Current classification and family-group names in Staphyliniformia (Coleoptera)[M]. Chicago: Field Museum of Natural History, 1992.
[7] SOLODOVNIKOV A Y, NEWTON A F. Phylogenetic placement of Arrowinini trib. n. within the subfamily Staphylininae (Coleoptera: Staphylinidae), with revision of the relict South African genusArrowinusand description of its larva [J]. Systematic Entomology, 2005, 30(3): 398−441. DOI: 10.1111/j.1365-3113.2004.00283.x
[8] MCKENNA D D, WILD A L, KANDA K, et al. The beetle tree of life reveals that Coleoptera survived end-Permian mass extinction to diversify during the Cretaceous terrestrial revolution [J]. Systematic Entomology, 2015, 40(4): 835−880. DOI: 10.1111/syen.12132
[9] CHATZIMANOLIS S, COHEN I M, SCHOMANN A, et al. Molecular phylogeny of the mega-diverse rove beetle tribe Staphylinini (Insecta, Coleoptera, Staphylinidae) [J]. Zoologica Scripta, 2010, 39(5): 436−449. DOI: 10.1111/j.1463-6409.2010.00438.x
[10] MCKENNA D D, FARRELL B D, CATERINO M S, et al. Phylogeny and evolution of Staphyliniformia and Scarabaeiformia: Forest litter as a stepping stone for diversification of nonphytophagous beetles [J]. Systematic Entomology, 2015, 40(1): 35−60. DOI: 10.1111/syen.12093
[11] BRUNKE A J, CHATZIMANOLIS S, SCHILLHAMMER H, et al. Early evolution of the hyperdiverse rove beetle tribe Staphylinini (Coleoptera: Staphylinidae: Staphylininae) and a revision of its higher classification [J]. Cladistics, 2016, 32(4): 427−451. DOI: 10.1111/cla.12139
[12] SCHOMANN A M, SOLODOVNIKOV A. Phylogenetic placement of the austral rove beetle genus Hyperomma triggers changes in classification of Paederinae (Coleoptera: Staphylinidae) [J]. Zoologica Scripta, 2017, 46(3): 336−347. DOI: 10.1111/zsc.12209
[13] CAMERON S L. Insect mitochondrial genomics: Implications for evolution and phylogeny [J]. Annual Review of Entomology, 2014, 59: 95−117. DOI: 10.1146/annurev-ento-011613-162007
[14] BOORE J L. Animal mitochondrial genomes [J]. Nucleic Acids Research, 1999, 27(8): 1767−1780. DOI: 10.1093/nar/27.8.1767
[15] SONG N, ZHANG H, LI H, et al. All 37 mitochondrial genes of aphid Aphis craccivora obtained from transcriptome sequencing: Implications for the evolution of aphids [J]. PLoS One, 2016, 11(6): e0157857. DOI: 10.1371/journal.pone.0157857
[16] JIN J J, YU W B, YANG J B, et al. GetOrganelle: A fast and versatile toolkit for accurate de novo assembly of organelle genomes [J]. Genome Biology, 2020, 21(1): 241. DOI: 10.1186/s13059-020-02154-5
[17] BERNT M, DONATH A, JÜHLING F, et al. MITOS: Improved de novo metazoan mitochondrial genome annotation [J]. Molecular Phylogenetics and Evolution, 2013, 69(2): 313−319. DOI: 10.1016/j.ympev.2012.08.023
[18] KUMAR S, STECHER G, TAMURA K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets [J]. Molecular Biology and Evolution, 2016, 33(7): 1870−1874. DOI: 10.1093/molbev/msw054
[19] KATOH K, STANDLEY D M. MAFFT multiple sequence alignment software version 7: Improvements in performance and usability [J]. Molecular Biology and Evolution, 2013, 30(4): 772−780. DOI: 10.1093/molbev/mst010
[20] ABASCAL F, ZARDOYA R, TELFORD M J. TranslatorX: Multiple alignment of nucleotide sequences guided by amino acid translations [J]. Nucleic Acids Research, 2010, 38(S2): W7−13. DOI: 10.1093/nar/gkq291
[21] CAPELLA-GUTIÉRREZ S, SILLA-MARTÍNEZ J M, GABALDÓN T. trimAl: A tool for automated alignment trimming in large-scale phylogenetic analyses [J]. Bioinformatics, 2009, 25(15): 1972−1973. DOI: 10.1093/bioinformatics/btp348
[22] KÜCK P, LONGO G C. FASconCAT-G: Extensive functions for multiple sequence alignment preparations concerning phylogenetic studies [J]. Frontiers in Zoology, 2014, 11(1): 1−8. DOI: 10.1186/1742-9994-11-1
[23] NGUYEN L T, SCHMIDT H A, VON HAESELER A, et al. IQ-TREE: A fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies [J]. Molecular Biology and Evolution, 2015, 32(1): 268−274. DOI: 10.1093/molbev/msu300
[24] LARTILLOT N, LEPAGE T, BLANQUART S. PhyloBayes 3: A Bayesian software package for phylogenetic reconstruction and molecular dating [J]. Bioinformatics, 2009, 25(17): 2286−2288. DOI: 10.1093/bioinformatics/btp368
[25] GREINER S, LEHWARK P, BOCK R. OrganellarGenomeDRAW (OGDRAW) version 1.3. 1: Expanded toolkit for the graphical visualization of organellar genomes [J]. Nucleic Acids Research, 2019, 47(W1): W59−64. DOI: 10.1093/nar/gkz238
[26] BAE J S, KIM I, SOHN H D, et al. The mitochondrial genome of the firefly, Pyrocoelia rufa: Complete DNA sequence, genome organization, and phylogenetic analysis with other insects [J]. Molecular Phylogenetics and Evolution, 2004, 32(3): 978−985. DOI: 10.1016/j.ympev.2004.03.009
[27] 林爱丽, 李欣欣, 赵新成, 等. 黑毛皮蠹线粒体基因组分析及皮蠹科系统发育分析 [J]. 昆虫学报, 2018, 61(4):477−487. DOI: 10.16380/j.kcxb.2018.04.010 LIN A L, LI X X, ZHAO X C, et al. Analysis of the mitochondrial genome of Attagenus unicolor japonicus (Coleoptera: Dermestidae) and a phylogenetic analysis of Dermestidae [J]. Acta Entomologica Sinica, 2018, 61(4): 477−487.(in Chinese) DOI: 10.16380/j.kcxb.2018.04.010
[28] SONG N, ZHANG H, ZHAO T. Insights into the phylogeny of Hemiptera from increased mitogenomic taxon sampling [J]. Molecular Phylogenetics and Evolution, 2019, 137: 236−249. DOI: 10.1016/j.ympev.2019.05.009
[29] SONG N, ZHAI Q, ZHANG Y L. Higher-level phylogenetic relationships of rove beetles (Coleoptera, Staphylinidae) inferred from mitochondrial genome sequences [J]. Mitochondrial DNA Part A, 2021, 32(3): 98−105. DOI: 10.1080/24701394.2021.1882444
[30] 林兴雨, 翟卿, 宋南, 等. 锯谷盗线粒体基因组及扁甲总科系统发育分析 [J]. 河南农业大学学报, 2023, 57(1):109−117. DOI: 10.3969/j.issn.1000-2340.2023.1.hennannydxxb202301012 LIN X Y, ZHAI Q, SONG N, et al. The mitochondrial genome of Oryzaephilus surinamensis and a phylogenetic analysis of cucujoidea [J]. Journal of Henan Agricultural University, 2023, 57(1): 109−117.(in Chinese) DOI: 10.3969/j.issn.1000-2340.2023.1.hennannydxxb202301012
-
期刊类型引用(2)
1. 李梦然,许学慧,赵萌莉. 不同形态氮肥对苗期玉米镉富集的影响. 中国土壤与肥料. 2021(04): 289-294 .  百度学术
百度学术
2. 冯文静,高巍,刘红恩,聂兆君,秦世玉,李畅,睢福庆,赵鹏. 植物生长调节剂促进小麦幼苗生长及降低镉吸收转运的研究. 河南农业大学学报. 2021(06): 1036-1044 .  百度学术
百度学术
其他类型引用(2)




 下载:
下载: