Molecular Identification of Juvenile Eels by DNA Fingerprints
-
摘要: 养殖鳗鲡种苗来源日趋多样,确定鳗苗种类是决定养殖成败的关键因素之一。应用鳗鲡属鱼类mtDNA COI基因的保守序列设计一对特异性引物,对21批次,共109个白苗期鳗鲡样品进行种类鉴定。结果显示,应用该引物可以在所采集到的鳗鲡样品中扩增出652 bp特异性基因片段,将样品COI基因序列与BOLD数据库中鳗鲡COI基因标准序列进行比对,共鉴定得7个种类归属,分别为日本鳗鲡A.japonica、美洲鳗鲡A.rostrata、欧洲鳗鲡A.anguilla、花鳗鲡A.marmorata、莫桑比克鳗鲡A.mossambica、吕宋鳗鲡A.luzonensis和太平洋双色鳗A.bicolor pacifica。对COI基因序列进行聚类分析,得到7个有很高节点支持率的主要类群。结果证实mtDNA COI基因在鳗鲡种类鉴定上具有很好的实用性。此外,在种类的鉴定中发现其中1批次的样品定种错误,有1个批次的鳗鲡样品中存在有2个种类的混杂,提示在实际的苗种养殖及交易中,对鳗鲡的种类鉴定上仍存在一定的错误和混杂,有必要引入DNA分子标记技术进行更加方便和准确的鉴定。Abstract: Accurate species identification is crucial for eel aquaculture, especially when increasing taxa of the fish were raised in the eel farms in China. This study applied a pair of specific primers from the conservative fragment of mtDNA COI gene in Anguilla to amplify 109 DNA samples extracted from 21 batches of eel juveniles for taxon identification. It was found that a specific 652bp fragment could be amplified from all of the samples. The BOLD Identification System (Barcoding of Life Data Systems: www.boldsystems.org) was used for sequence similarity search on the COI genes. Seven species, including A. japonica, A. rostrata, A.anguilla, A.marmorata, A.mossambica, A.luzonensis and A.bicolor pacifica, were identified. The cluster analysis showed 7 main clades with high rate of support at the nodes. It suggested that the COI identification method could be satisfactorily employed for eel juveniles identification. Incidentally, during the course of this study a former mistaken identification on a batch of juvenile fish was discovered. Therefore, the DNA barcode-based identification method was highly recommended for implementation to benefit the commercial trade and eel aquaculture.
-
Keywords:
- eel /
- juveniles /
- COI /
- molecular identification
-
鳗鲡养殖业大量的苗种需求导致传统养殖鳗鲡种类, 如日本鳗鲡Anguilla japonica和欧洲鳗鲡A.anguilla自然资源量急剧下降,为保护自然资源,产苗国也对这些鳗鲡种类进行了一定程度的保护,更加剧了市场上的鳗鲡种苗供不应求、价格高企的局面。在这种情况下,养殖业必然将目光转向那些种苗资源尚未被开发的种类。鳗鲡苗种的多样化,养殖业者难以准确鉴别苗种,给鳗鲡的养殖和销售造成了很大的困扰。
研究发现,在大多数的脊椎动物中,线粒体细胞色素c氧化酶亚基Ⅰ(mitochondrial cytochrome coxidase subunit I,COI) 基因序列差异能够成功地区分大多数脊椎动物的物种,可以作为鉴定物种的DNA条形码[1]。FISH-BOL (Fish Barcode of Life) 组织已开展大量的国际合作,旨在建立所有鱼类将近3万个种的DNA条形码数据库,重点对1.5万种海洋鱼类的DNA条形码数据进行收集[2],到2016年8月,已获得9 700多种鱼类的DNA条形码数据 (http://www.fishbol.org/)。因此有条件应用DNA条形码技术对引进的鳗苗种类进行准确的鉴定。
本研究拟应用DNA条形码技术对收集自福建省几个鳗鲡养殖场的鳗鲡种类样品进行种类鉴别,以期对福建省养殖鳗鲡苗种的现状做初步的分析。
1. 材料与方法
1.1 样品来源及保存
鳗苗样品收集自福建省长乐、莆田、福清、龙岩等地鳗鲡养殖场。试验中取新鲜鳗鲡白苗尾部肌肉样品或保存于95%乙醇 (-20℃) 的固定的肌肉样品。每批次鳗苗样品至少随机取4只作为鉴定样品。
1.2 试验方法
1.2.1 鳗鲡基因组DNA的提取
基因组DNA提取方法参照林能锋等[3]并略有修改,具体方法如下:剪取新鲜或95%乙醇保存的肌肉样品,加入STE缓冲液 (0.1 mol·L-1NaCl,10 mmol·L-1Tris,100 mmol·L-1EDTA,pH=8.0),剪碎肌肉,离心,去上清,加入600 μL DNA抽提缓冲液 (10 mmol·L-1Tris,100 mmol·L-1EDTA,0.5%SDS,pH=8.0) 和3 μL蛋白酶K (20 mg·mL-1),55℃水浴消化至管内液体澄清,加等体积酚:氯仿:异戊醇 (25:24:1) 抽提2次,每次10 min,12 000 r·min-1离心10 min,加氯仿抽提10 min,12 000 r·min-1离心5 min,取上清,加入1/25体积的5 mol·L-1NaCl溶液,混匀后加入2倍体积预冷的无水乙醇沉淀DNA。4℃,12 000 r·min-1离心10 min,去上清。70%乙醇溶液洗涤1次,4℃,12 000 r·min-1离心10 min,去上清。于无菌操作台上吹干沉淀后加100 μL无菌超纯水,4℃溶解过夜。0.7%琼脂糖凝胶电泳检测DNA样品质量,Nanodrop2000测定样品DNA浓度。调整样品DNA的浓度大约在100 ng·μL-1,-20℃保存。
1.2.2 鳗鲡mtDNA COI基因片段的扩增及测序
比对多种鳗鲡mtDNA COI基因序列,以Primer3引物设计软件[4-5]在其保守区设计引物。扩增的正向引物序列为:5′-cct ata tct cgt att tgg tgc ct-3′,反向引物序列为:5′-taa gtg ttg gta gag gat tgg gt-3′。PCR反应体系为:模板DNA 50~100 ng,正、反向引物各0.5 μmol·L-1,dNTP 200 μmol·L-1,Mg2+1.5 mmol·L-1,Taq DNA聚合酶 (TaKaRa)1 U,反应总体积为20 μL。PCR反应条件为:94℃预变性5 min后进入循环体系,94℃ 45 s,53℃ 45 s,72℃ 45 s,共30个循环,循环结束后于72℃进行延伸反应10 min,扩增产物于4℃保存。1.0%琼脂糖凝胶电泳分析PCR结果。PCR产物送上海生工或是福州博尚生物技术公司测序。
1.2.3 种类归属的判定
将鳗鲡幼苗DNA的COI基因PCR扩增产物的测序结果与BOLD数据库 (http://www.boldsystems.org/index.php/IDS_OpenIdEngine) 中的物种COI基因的标准序列进行自动搜索比对,以相似度高于98%为判定种类的标准来确认鳗苗样品的种类归属[6]。
1.2.4 鳗鲡COI基因序列的聚类分析
所得测序结果用Clustal X软件进行比对,并辅以人工校正。用MEGA7.0软件[7]计算种内和种间遗传距离。分子系统树的构建采用邻接法 (neighbour-joining, NJ) 和最大简约法 (maximum parsimony, MP)。邻接法分析采用Kimura-2-parameter遗传距离,进行1 000次自展分支检验。最大简约法分析使用启发式搜索,构树方法采用SPR法 (Subtree-Pruning-Regrafting),可靠性由1 000次自展分支检验。
2. 结果与分析
2.1 PCR引物的扩增有效性
对2007-2015年的14个鳗鲡养殖场的21批次共109个鳗鲡苗种样品DNA进行线粒体COI基因652 bp片段的扩增。结果表明,应用本试验中的PCR引物,所有的样品均能够得到有效的扩增,且扩增条带的琼脂糖电泳条带单一,特异性好。
2.2 鳗鲡样品COI基因序列的比对
109个样品的PCR扩增产物测序结果进行整理后,在BLOD数据库中搜索相似序列,所有的样品COI基因片段序列均可以在BOLD数据库中找到相似性达99%以上的标准序列,可判定样品中有7个种。其中日本鳗鲡A.japonica 1批次6尾,美洲鳗鲡A.rostrata 7批次36尾,欧洲鳗鲡A.anguilla 4批次20尾,花鳗鲡A.marmorata 5批次24尾,莫桑比克鳗鲡A.mossambica 2批次11尾,吕宋鳗鲡A.luzonensis 1批次6尾,太平洋双色鳗A.bicolor pacifica1批次6尾。其中在2012年2月长乐某鳗场的1个批次6个样品中鉴定出5尾欧洲鳗鲡,1尾花鳗鲡。长乐某鳗场2011年1月的样品批次误判美洲鳗鲡为非洲鳗鲡。
2.3 聚类分析及系统发育分析结果
采用MEGA7.0软件,基于Kimura-2-parameter法计算7种鳗鲡的种内和种间遗传距离。结果如表 1所示。7种鳗鲡种内平均遗传距离为0.006(0.003~0.019),种间平均遗传距离为0.073(0.033~0.094)。种间平均遗传距离为种内平均遗传距离的12倍。
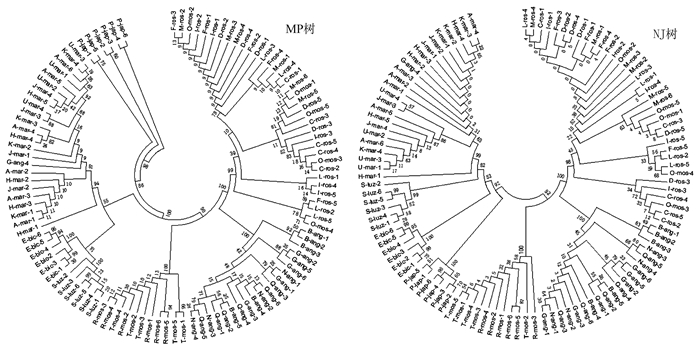
表 1 基于mtDNA COI基因序列的7种鳗鲡种内及种间遗传距离Table 1. Genetic distances among 7 eel species according to partial mtDNA COI gene sequenceeel A.rostrata A.anguilla A.mossambica A.marmorata A.luzonensis A.bicolor pacifica A.japonica A.rostrata 0.004 A.anguilla 0.033 0.007 A.mossambica 0.075 0.081 0.003 A.marmorata 0.073 0.082 0.070 0.004 A.luzonensis 0.078 0.082 0.076 0.035 0.005 A.bicolor pacifica 0.087 0.090 0.081 0.053 0.061 0.019 A.japonica 0.087 0.094 0.086 0.058 0.062 0.087 0.003 本研究采用MP法和NJ法对7种鳗鲡的109个COI基因序列构建了分子系统发育树 (图 1),2种构树法得到具有大致相似的拓扑结构,同一种类的不同个体均可聚在同一分支内,7个种均可聚为独立的分支,形成单系。并且在种间分支节点上具有较高的支持率。
从N_J树来看,亚洲种A.marmorata、A.luzonensis、A.bicolor pacifica和A.japonica聚成一个大的分支,大西洋种A.rostrata则和A.anguilla互为姊妹群,印度洋种A.mossambica与其他2支独立开。分支群间的关系显示它们符合于鳗鲡的地理分布范围。其中印度洋-大西洋区有3种,印度洋-太平洋区有4种,本研究所构建的系统发育树与以16S rRNA和Cyt b基因序列所描绘的系统发育树[8-9]相类似。
3. 讨论与结论
据世界鱼类数据库Fishbase (http://www.fishbase.org/) 记录的鳗鲡属鱼类有效种名 (valid name) 共有21种。其中日本鳗鲡、欧洲鳗鲡、美洲鳗鲡、大鳗鲡、澳洲鳗鲡、新澳鳗鲡和花鳗鲡等7种鳗鲡有较为成功的人工养殖历史。目前,日本鳗鲡、欧洲鳗鲡、美洲鳗鲡和花鳗鲡是我国的主要养殖种类,但这些种类的现有苗种资源量已不足以满足鳗鲡养殖业者的需求,因此在业界对养殖鳗鲡种类的选择和养殖尝试也日渐多元,不断探索引进多种鳗鲡进行试养,如莫桑比克鳗A.mossambica、太平洋双色鳗A.bicolor pacifica,等等。由于不同鳗鲡苗种的养殖特性和价格有着较大的差别,因此,鳗鲡苗种的准确鉴定的重要性显得尤为突出。
目前,鳗鲡苗种鉴定的最主要方法就是依据外形和脊椎骨数目作为分类的判别依据,但不同种鳗鲡的脊椎骨数目也多有重叠,且同一种鳗鲡的脊椎骨数目也会随着纬度的升高而增加。也曾有学者用背鳍起点到肛门间的脊椎骨数目作为鉴定鳗苗种类的方法,省去计数全部脊椎骨节数的麻烦,也取得了一定的快速鉴定的效果,但是否能作为鉴定种类的标准,还需要做更多的统计来确认。鳍形也是鉴定鳗鲡种的一个重要性状,鳗鲡属鱼类中鳍形中有6种为短鳍形,其余均为长鳍形。而一些基于成鳗的生物学性状在“玻璃鳗”中表现并不显著或者是难以辨认。在生产上还常用生理生态 (活动状态、苗种汛期及对特种药物耐受度) 等其他相关特性加以区分,但很难成为分类的“金标准”。
总之,采用常规形态学方法鉴定鳗鲡幼苗有以下几个缺陷:(1) 外部形态特征存在一定的变异范围, 从而导致鉴定结果不准确; (2) 形态学方法易受发育阶段的限制; (3) 鉴定需要专业人员和繁重的工作量。
研究者用同功酶[10],RAPD[11-12]和微卫星[13]等不同的分子标记对鳗鲡成功进行了种质的鉴定,但这些标记通常需要多个标记位点的组合和较为繁琐的数据分析,因此,较难实现快速准确鉴定物种。
大量的研究表明,利用COI基因序列的DNA条形码技术能够作为物种鉴定的有效手段,在后生生物的不同生长阶段、不同生长发育时期的不同形态条件下都能够很好鉴别物种[14]。因此在鳗鲡幼苗这些形态相似性很高物种的准确鉴别中具有很高的应用价值。FISH-BOL组织建立的鱼类COI基因序列的数据库,提供了一种快速、简便而且可信的分类方法。吴宁等[15]采用DNA测序技术对我国常见6种养殖鳗鲡的COI基因片段进行了PCR扩增和测序,分析了COI基因的碱基组成的种间变异并构建了6种鳗鲡的系统发育树,认为COI基因可以做为鳗鲡种质鉴定的工具。本文通过设计一对能高效扩增鳗鲡线粒体COI基因片段的引物,对一些新购鳗苗样品进行了种类的鉴定,获得了其种类归属,并鉴定了一些在生产中产生的错认或存在混杂的样品,为鳗苗种类的准确鉴定提供了一种可靠的技术手段。所得的COI序列通过聚类分析,与种类鉴定结果一样,获得了7个主要的分支,可分别代表不同的鳗鲡物种,从而与BOLD的鉴定结果相对照,证实COI基因可以作为鳗鲡属鱼类的DNA指纹。基于所得到鳗鲡mtDNA COI基因序列的系统发育分析结果与其他基于不同基因或分子标记的结果进行参照比较,可为鳗鲡属鱼类的系统发育研究提供的更为准确的信息。
同时,通过本研究的调查发现,有养殖业者在引进新的养殖种类时也存在着一定的盲目性,在对养殖种类及其生物学特性并不了解的情况下进行了试养,结果造成了较大的损失,而在鳗苗的引进和交易过程中也存在着一些种类混杂和种类不清的情况,也需要相关单位进行协调和规范。
-
表 1 基于mtDNA COI基因序列的7种鳗鲡种内及种间遗传距离
Table 1 Genetic distances among 7 eel species according to partial mtDNA COI gene sequence
eel A.rostrata A.anguilla A.mossambica A.marmorata A.luzonensis A.bicolor pacifica A.japonica A.rostrata 0.004 A.anguilla 0.033 0.007 A.mossambica 0.075 0.081 0.003 A.marmorata 0.073 0.082 0.070 0.004 A.luzonensis 0.078 0.082 0.076 0.035 0.005 A.bicolor pacifica 0.087 0.090 0.081 0.053 0.061 0.019 A.japonica 0.087 0.094 0.086 0.058 0.062 0.087 0.003 -
[1] HEBERT P D N, RATNASINGHAM S, DEWAARD J R. Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings:Biological Sciences, 2003, 270:S96-S99. DOI: 10.1098/rsbl.2003.0025
[2] WARD R D, HANNER R, HEBERT P D N. The campaign to DNA barcode all fishes, FISH-BOL[J].Journal of Fish Biology, 2009, 74(2):329-356. DOI: 10.1111/jfb.2009.74.issue-2
[3] 林能锋, 苏永全, 丁少雄, 等.大黄鱼微卫星标记引物在石首鱼科几个近缘种中的通用性研究[J].中国水产科学, 2008, 15(2):237-243. http://www.cnki.com.cn/Article/CJFDTOTAL-ZSCK200802007.htm [4] UNTERGASSER A, CUTCUTACHE I, KORESSAAR T, et al.Primer3-new capabilities and interfaces[J]. Nucleic Acids Research, 2012, 40(15):e115. DOI: 10.1093/nar/gks596
[5] KORESSAAR T, REMM M.Enhancements and modifications of primer design program Primer3[J].Bioinformatics, 2007, 23(10):1289-1291. DOI: 10.1093/bioinformatics/btm091
[6] RATNASINGHAM S and HEBERT P D N. BOLD:The barcode of life data system (www.barcodinglife.org)[J]. Molecular Ecology Note, 2007, 7:355-364. DOI: 10.1111/j.1471-8286.2007.01678.x
[7] KUMAR S, STECHER G and TAMURA K. MEGA7:Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7):1870-1874. DOI: 10.1093/molbev/msw054
[8] BASTROP R, STREHLOW B, JURSS K, STURMBAUER C. A new molecular phylogenetic hypothesis for the evolution of freshwater eels[J]. Molecular Phylogenetics and Evolution, 2000, 14(2):250-258. DOI: 10.1006/mpev.1999.0694
[9] AOYAMA J, NISHIDA M, TSUKAMOTO K. Molecular phylogeny and evolution of the freshwater eel, genus Anguilla[J]. Molecular Phylogenetics and Evolution, 2001, 20(3):450-459. DOI: 10.1006/mpev.2001.0959
[10] 罗土炎, 饶小珍, 林岗, 等. 4种鳗鲡同工酶的比较研究[J].福建农业学报, 2009, 24(5):421-428. http://www.fjnyxb.cn/CN/abstract/abstract385.shtml [11] 杨弘, 王希道, 吴婷婷, 等.用RAPD技术研究3种鳗鱼的种质鉴定[J].中国水产科学, 2002, 9(3):269-272. http://www.cnki.com.cn/Article/CJFDTOTAL-ZSCK200203018.htm [12] 张新艳, 樊海平, 范斯敏, 等.应用RAPD技术对6种玻璃鳗的种质鉴定和遗传多样性分析[J].江西农业大学学报, 2010, 32(4):683-688. http://www.cnki.com.cn/Article/CJFDTOTAL-JXND201004013.htm [13] 梁俊, 李道季, 卢莉琼.日本鳗鲡 (Anguilla japonica) 和欧洲鳗鲡 (A.anguilla) 的微卫星差异[J].海洋与湖沼, 2003, 34(4):414-421. DOI: 10.11693/hyhz200304008008 [14] HEBERT P D N, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proc.R. Soc. Lond.B, 2003, 70:313-321.
[15] 吴宁, 黎中宝, 林小云, 等. 6种鳗鲡 (Anguilla) 线粒体DNA COI序列的比较研究[J].海洋与湖沼, 2010, 41(6):930-934. DOI: 10.11693/hyhz201006020020 -
期刊类型引用(1)
1. 付秀兰, 谢斐昂, LUSANA James, 陈永久, 李德伟, 蒋日进. 基于线粒体D-loop DNA序列的瓯江口鳗鲡幼苗遗传鉴定. 浙江海洋大学学报(自然科学版). 2018(06): 489-493 .  百度学术
百度学术
其他类型引用(0)




 下载:
下载: