Rhizosphere Bacterial Community and Diversity at Fields of Wilt Resistant or Susceptible Mulberry Trees
-
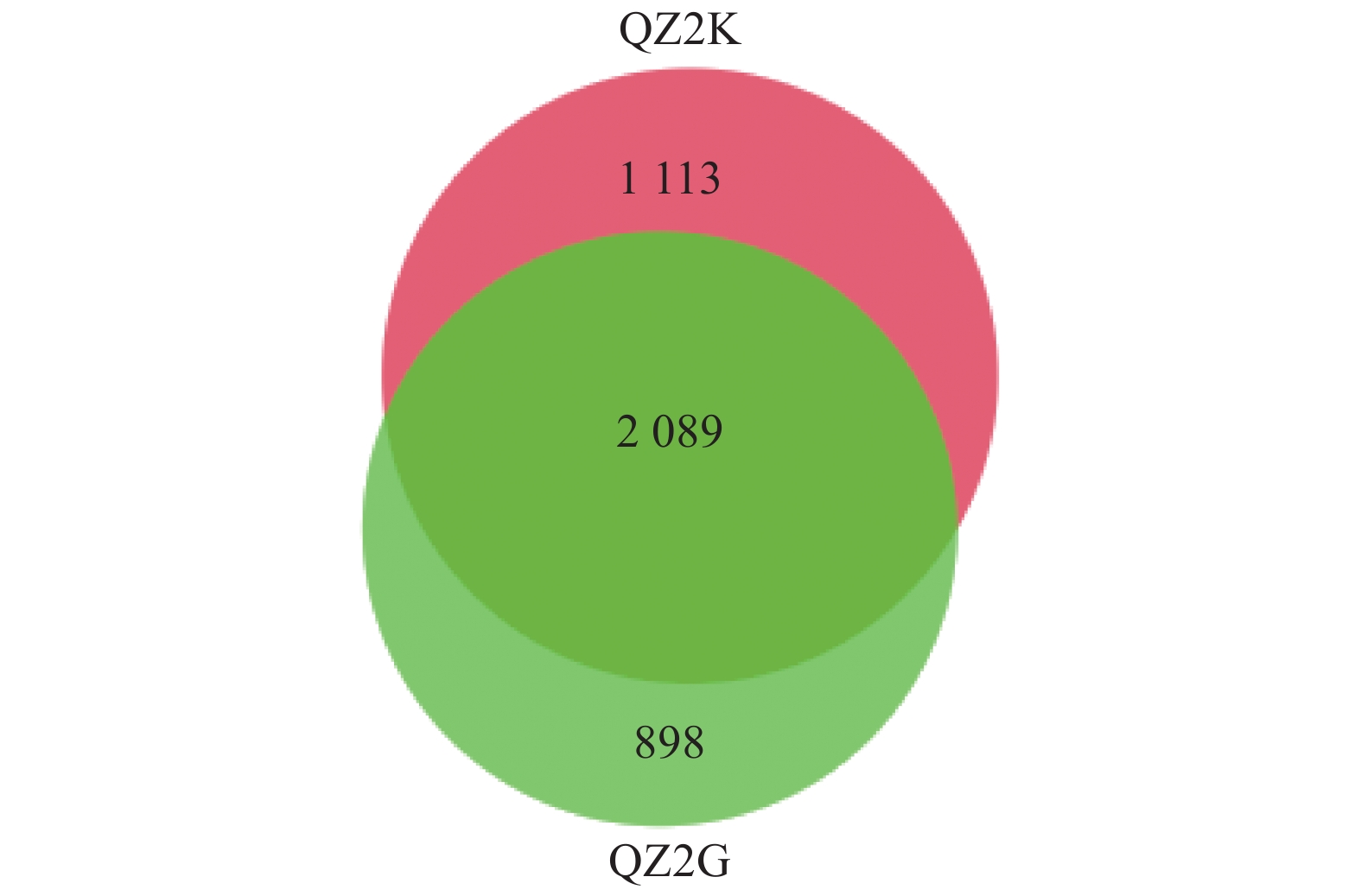
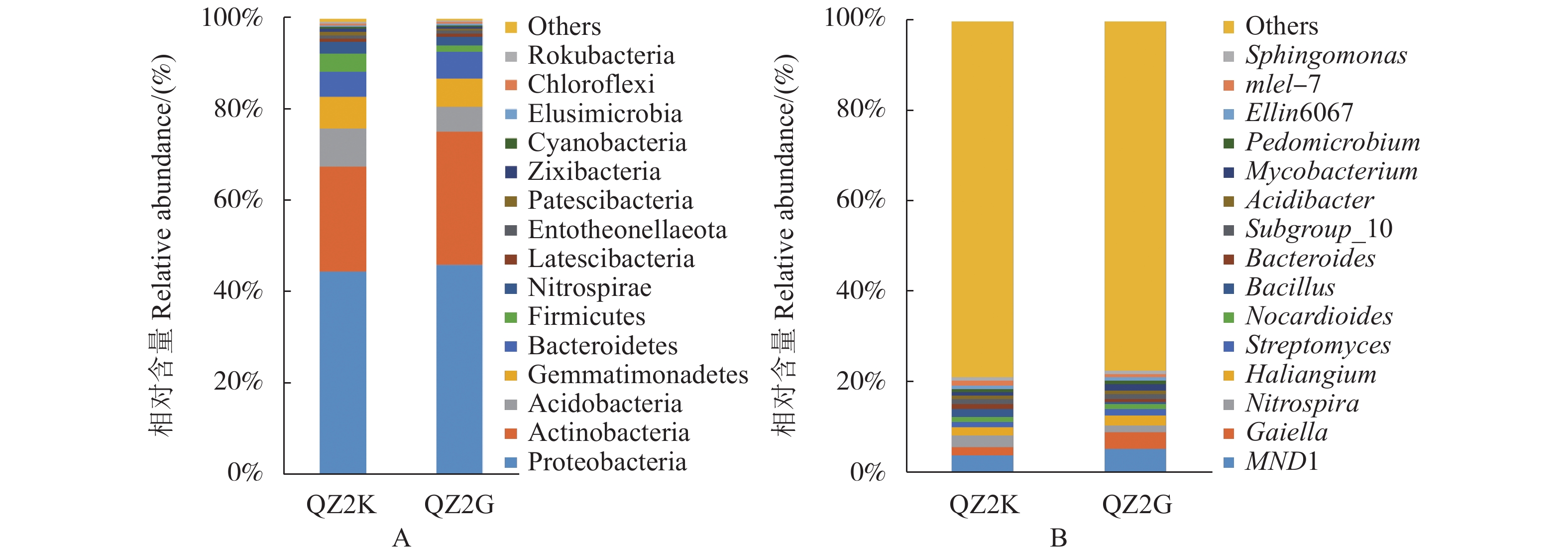
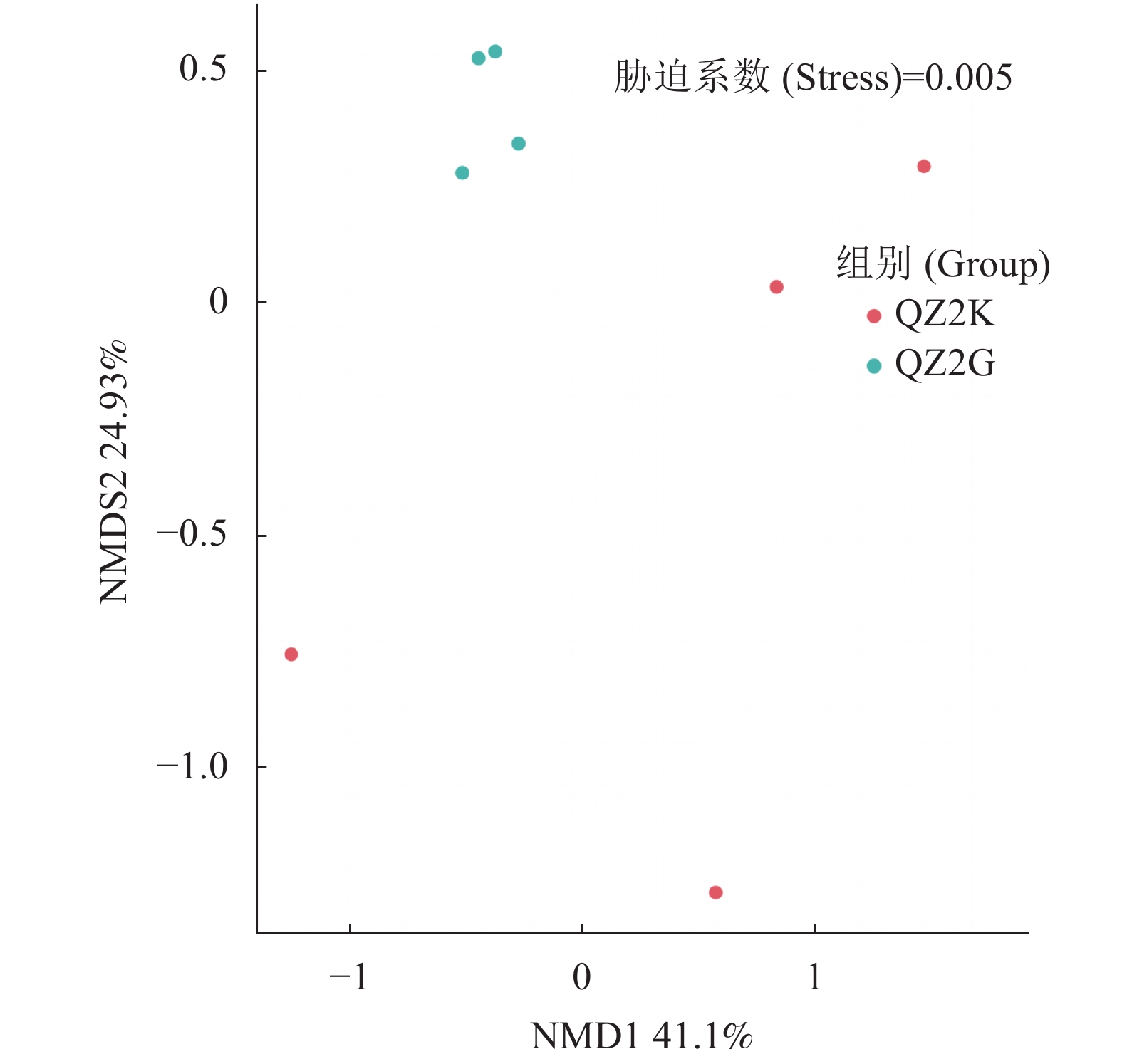
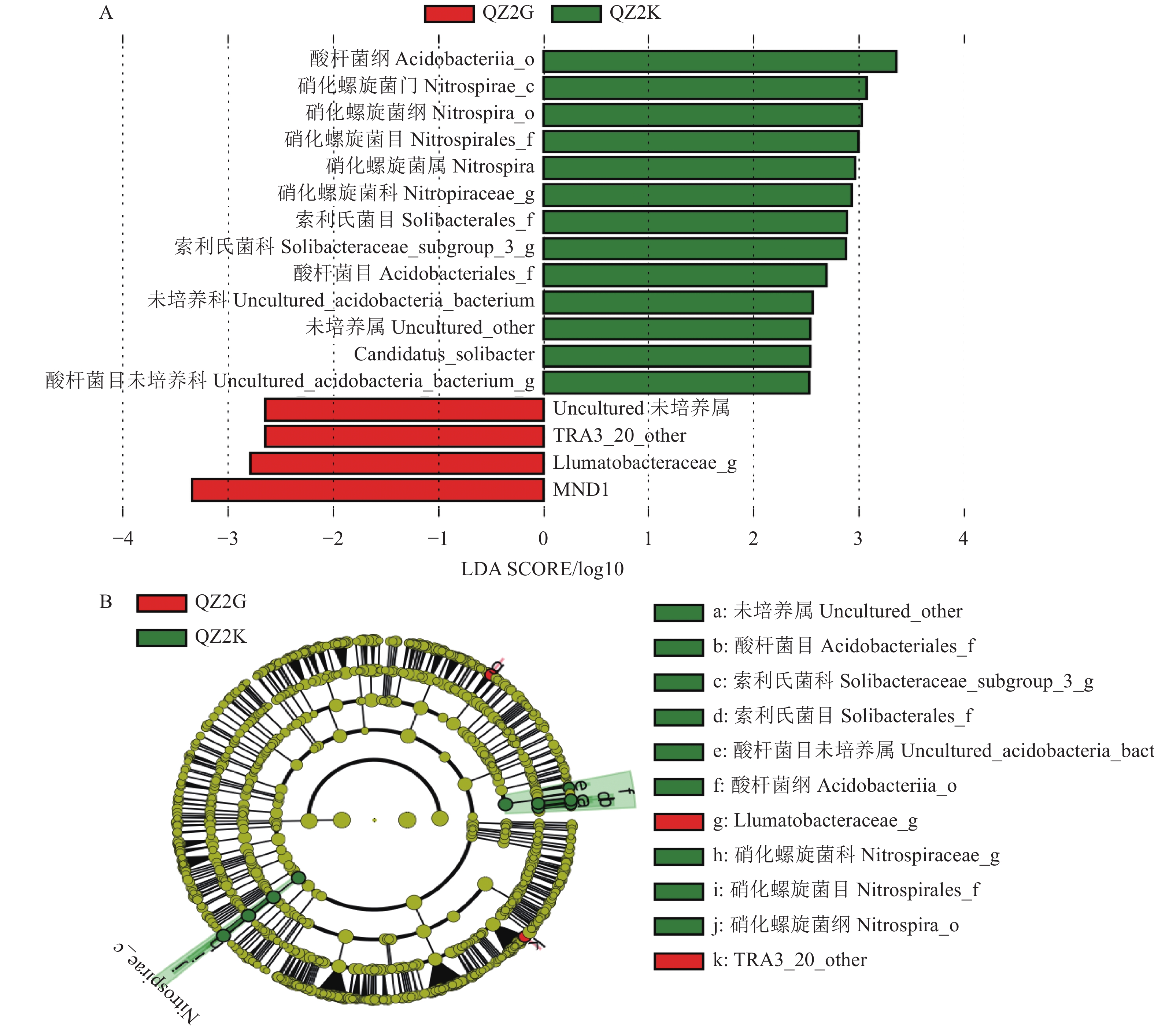
摘要:目的 通过抗、感青枯病桑树根际土壤细菌群落结构与多样性分析,阐明根际细菌群落与桑树抗、感青枯病的相关性。方法 采用Illumina MiSeq测序技术对抗青枯病桑树基因型(抗青283×抗青10)和感病基因型(桂桑优62)根际细菌的16S rRNA基因V3-V4区进行扩增,高通量测序后比较分析了其根际细菌群落结构与多样性。结果 ① 抗、感青枯病桑树根际土壤的优势菌门为变形菌门、放线菌门、酸杆菌门、芽单胞菌门和拟杆菌门,优势菌属为MND1、Gaiella、硝化螺菌属、Haliangium和链霉菌属;② 抗、感青枯病桑树根际土壤细菌群落Alpha多样性无显著差异,但经NMDS排序,则可显著区分(stress=0.005<0.05),其中抗性基因型根际土壤中具有重要作用的细菌类群较多,包括:硝化螺旋菌纲、酸杆菌纲、硝化螺旋菌目、索利氏菌目、酸杆菌目、硝化螺旋菌科、酸杆菌目未培养科、索利氏菌科Subgroup 3和Subgroup 2未培养科,而敏感基因型桂桑优62根际重要作用的细菌类群则主要为Ilumatobacteraceae和TRA3 20 Other两个科。③ 抗、感青枯病桑树根际细菌差异COG类目为763,仅占17.25%。结论 抗、感青枯病桑树根际土壤细菌群落丰富度和多样性虽无显著差异,但其重要作用的类群显著不同,说明桑树根际土壤细菌群落结构可能与种质对青枯病的抗性具有相关性。本结果将为进一步研究桑树根际微生态特征、筛选有益功能菌株并用于桑青枯病的有效防控具有重要意义。
-
关键词:
- 桑树 /
- 青枯病 /
- Illumina高通量测序 /
- 细菌多样性 /
- 群落结构
Abstract:Objective Structure and diversity of rhizosphere bacterial communities at fields of mulberry trees resistant (QZ2K) or susceptible (QZ2G) to wilt disease were studied.Method The V3-V4 regions of 16S rRNA in rhizosphere bacteria were amplified and sequenced using high-throughput sequencing technology on Illumina MiSeq to determine the bacterial community structure, diversity, and functions. Results from the two field samples were compared.Result (1) At phylum and genus levels, the dominant rhizosphere bacteria were similar at QZ2K (Kangqing 283×Kangqing 10 mulberry field) and at QZ2G (Guisangyou 62 mulberry field). The phyla included Proteobacteria, Actinobacteria, Acidobacteria, Gemmatimonadetes, and Bacteroidetes, while the genera consisted of MND1, Gaiella, Nitrospira, Haliangium, and Streptomyces. (2) Although no significant difference in the alpha diversity of the bacteria communities at the two different fields, the NMDS ordination showed significant differences (stress 0.005<0.05). At QZ2K, the bacteria related to significant metabolic functions were Nitrospira, Acidobacteriia, Nitrospirales, Solibacteraies, Acidobacteriales, Nitrospiraceae, and the uncultured Acidobacteria, Solibacteraies Subgroup 3, and Solibacteraies Subgroup 2. At QZ2G, only Ilumatobacteraceae and TRA3-20-other were identified. (3) According to the Wilcoxon signed rank test, the 763 different rhizosphere bacteria orthologs identified were only 17.25% of all COG orthologs on both fields.Conclusion There were no significant differences in the richness and diversity of rhizosphere bacteria community between the two fields. However, the bacteria associated with significant functions differed significantly between them, which could well be the species that made the difference in the occurrence of wilt disease on the mulberry plants. The information obtained in this study was of value for further studies on the microecological characteristics of mulberry rhizosphere as well as selection and application of functional bacteria for wilt control. -
0. 引言
【研究意义】香蕉(Musa acuminat L. AAA group cv. Brazilian)是热带地区的重要经济作物之一,被联合国粮农组织(FAO)定位为发展中国家仅次于水稻、小麦、玉米之后的第四大粮食作物。然而其根系浅生,易受旱,干旱胁迫极大地伤害香蕉正常的生理代谢活动,造成香蕉减产和品质下降,是香蕉产业中亟需解决的关键问题。【前人研究进展】香蕉水通道蛋白(Aquaporin,AQP)是生物体内广泛存在的一类位于各种细胞膜上24~34 kD的小分子跨膜蛋白[1-2],隶属于MIP(Major intrinsic protein, MIP)超家族,能够调节水分以及甘油、硼、二氧化碳等小分子物质的运输[3-4]。AQP在植物的生长发育中起着重要的作用[5-7],如种子萌发、细胞伸长、种子发芽、生殖生长和气孔运动等[8]。越来越多的研究表明,AQP能够维持植物中水的稳态响应各种非生物胁迫[4],主要包括冷害、盐害、旱害等。近年来的研究结果表明AQP能够提高植物的抗旱性,在植物中过表达OsPIP1、OsPIP2、VfPIP1、BnPIP1能够增加植物对渗透胁迫或干旱胁迫的耐受性[4]。过表达TaAQP7提高了转基因烟草的抗旱性[9]。用小麦TaTIP2; 2转化拟南芥提高了转基因植物的抗旱能力[10]。【本研究切入点】前期试验表明了MaAQP1提高了转基因拟南芥的抗旱性[11],而其提高植物抗旱性的作用机制仍未有报道。【拟解决的关键问题】本试验通过对干旱胁迫的香蕉进行cDNA合成及文库构建,克隆MaAQP1启动子并构建其诱饵载体,为进一步进行酵母单杂等研究MaAQP1响应干旱胁迫的作用机制奠定了基础。
1. 材料与方法
1.1 试验材料
1.1.1 植物材料
将5叶1心的巴西蕉(Musa acuminat L. AAA group cv. Brazilian)进行干旱胁迫处理[11],当土壤含水量为45%时取根、茎、叶一起混合冻样。
1.1.2 试验试剂
Plasmid Maxi Kit购自德国Qiagen公司,TRIzol Reagent购自美国Invitrogen公司,1 kb DNA Ladder和超纯琼脂糖购自上海海科生物技术有限公司,Taq DNA聚合酶等购自宝生物工程有限公司。cDNA文库构建试剂盒购自Clontech公司;Adenine Sulfate(Ade)为Amresco公司产品。各种工具酶、Marker购自TaKaRa公司。PCR引物合成和测序由华大完成。
1.2 试验方法
1.2.1 MaAQP1启动子克隆及序列分析
在香蕉全基因组测序[12]的基础上通过香蕉A基因组网址(http://banana-genome.cirad.fr/)获得MaAQP1的ATG起始密码子的2 kb的5′侧翼区域。通过引物从香蕉基因组DNA扩增序列,最后获得MaAQP1的翻译起始位点(ATG)的1 362 bp视为全长启动子,将其构建到pMD19-T载体上并测序确认。利用启动子顺式作用元件预测软件PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对该启动子元件进行预测分析。
1.2.2 pHIS2-pMaAQP1诱饵载体构建
根据MaAQP1启动子序列分别在其两端添加Eco RI和Sac I的酶切位点序列设计引物,引物序列pMaAQP1-FAAGAATTCCTGCGACGGTTCGTAAAGAG;pMaAQP1-R:GGGAGCTCTGAGTGGAGGAATCAGGGTG,从pMD19T-pMaAQP1上进行扩增,将扩增条带回收,和pHIS2表达载体分别用Eco RI和Sac I进行双酶切,酶切产物分别纯化回收后进行连接,连接产物转化到E.coli DH5α感受态细胞中,活化后涂布于LB固体培养基上(内含30 ng·mL−1 Amp),37 ℃过夜培养后挑取单菌落摇菌,提取质粒进行酶切鉴定,然后选取阳性单克隆送华大基因测序,测序正确的即为诱饵载体pHIS2-pMaAQP1。
1.2.3 诱饵载体转化酵母菌株及自激活检测
将诱饵质粒pHIS2-pMaAQP1、阳性和阴性对照分别转化到酵母菌Y187感受态中,并涂布于平板SD-Trp-Leu-His + 50 mmol·L−1 3AT上生长。从SD-T平板中挑取单菌落于50 mL液体培养基中,在温度30 ℃、转速225 r·min−1培养18 h。将菌液转接于500 mL液体YPDA培养基中,使初始OD600 = 0.2,30 ℃,转速824 r·min−1 条件下培养4~5 h,至OD600=0.6。在转速1 737 r·min−1 下离心5 min集菌。依次用30 mL无菌H2O,转速1 000 r·min−1,室温下5 min集菌并弃上清。依次用20 mL 0.1 mol·L−1 LiAc,10 mL 0.1 mol·L−1 LiAc重复上一步骤,离心集菌弃上清。向离心管中依次加入1.44 mL 1 mol·L−1 LiAc,9.6 mL 50%PEG3350,25 μg文库质粒DNA,300 μL ssDNA(10 mg·mL−1)剧烈振荡1 min至完全混匀。在30 ℃水浴中孵育30 min。42 ℃水浴热激25 min。重复30 ℃水浴1 h。在室温下转速1 453 r·min−1 离心5 min离心集菌弃上清。每个转化用200 μL无菌H2O重悬菌体,温和混匀,并涂布于相应的缺陷型平板中,在30 ℃中培养3~4 d。
分别从表1中各转化的平板上各随机挑取3个单菌落,稀释后涂板至对应的不添加histidine、添加不同浓度(25、50 mmol·L−1)3AT的缺陷型平板上(3AT是酵母HIS3蛋白合成的竞争性抑制剂,用于抑制His3基因的泄漏表达),30 ℃恒温培养3 d。记录每块平板上的克隆数,并计算每个转化反应在缺陷平板上的转化子数量和生长率。
表 1 酵母转化反应Table 1. Yeast transformation reaction反应
ReactionAD质粒
AD plasmidBD质粒
BD plasmid转化平板
Conversion plate检测平板
Detection plate检测内容
Test content1 —— pHIS2-pMaAQP1 SD-T SD-TH+3AT 自激活检测 Self-activation detection 2 pGAD53m pHIS2 SD-TL SD-TLH+3AT 阴性对照 Negative control 3 pGAD53m pHIS2-p53 SD-TL SD-TLH+3AT 阳性对照 Positive control 1.2.4 用于构建文库的cDNA合成
用CTAB法提取干旱胁迫处理的香蕉组织,使用Oligotex mRNA Kits(Qiagen)分离纯化样本的mRNA。用Supscript double stand cDNA Kit分别进行cDNA第一链和第二链的合成,加5′接头(3个读码框,每个读码框连接一份,共3份),使用1%浓度的低熔点琼脂糖胶,电泳cDNA产物,切胶回收1 000 bp以上的片段,用14 μL的DEPC水溶解回收产物。1%琼脂糖凝胶电泳检测。
1.2.5 酵母单杂交文库构建
(1)cDNA与载体的连接 使用同源重组的方式,将7 μL cDNA与3 μL的经酶切处理线性化的pGADT7-Rec2(单杂交)载体,5 μL的Infusion重组酶和5 μL的H2O混匀。于50 ℃反应1 h,加入2 μL灭活的Proteinase K重组酶,78 μL无菌H2O至总体积为100 μL,加入1 μl Glycogen(20 μg·μL−1),375 μL 100% ethanol,50 μL 7.5 mol·L−1 NH4OAc,混匀并于−80 ℃ 1 h,在4 ℃下,转速6 948 r·min−1离心30 min。弃上清,加入150 μL 70%乙醇,在4 ℃下,转速6 948 r·min−1离心3 min,重复此步骤一次,去尽上清,在室温下将cDNA晾干5~1 min。用10 μL DEPC H2O重悬cDNA,瞬离2 s收集cDNA,置冰上。
(2)电转化大肠杆菌感受态细胞 每2.5 μL的重组产物转化50 μL感受态细胞,将1 mm电转杯(Bio-Rad)置于−80 ℃预冷30 min。将2.5 μL重组产物和50 μL感受态细胞加入电转杯,置冰上45 min。于电转化仪(Bio-Rad)上电击,电击后迅速向电转杯中加入LB培养基1 mL,然后取入到新的15 mL离心管,补足体积到5 mL,置于37 ℃,824 r·min−1~868 r·min−1离心培养至少1 h。培养结束后,将培养物依次稀释10、100、1 000、10 000倍,分别取10 μL稀释液涂平板,剩余培养物可4 ℃保存过夜,或者加入甘油至终浓度20%存于−80 ℃。
(3)构建文库质量鉴定 库容量的鉴定:取转化后菌原液10 μL稀释1 000倍后,从中取出10 μL涂布LB平板(含氨苄抗性),第2 d计数。库容量(CFU·mL−1)= 平板上的克隆数/10 ×1 000 ×103,文库总库容量(CFU)= 库容量×文库菌液总体积。插入片段大小鉴定:挑取平板上的单克隆,PCR扩增,电泳检测PCR产物大小。
2. 结果与分析
2.1 基因启动子的克隆
如图1所示,根据香蕉A基因组网站(http://banana-genome.cirad.fr)设计启动子的引物进行扩增,扩增长度为1 362 bp。
2.2 基因启动子的序列分析
通过plantcare和PLACE(www.dna.affrc.go.jp)分析其顺式作用元件,结果显示了1 362 bp的启动子序列中共有72个顺式作用元件,包括了TATA-box和CAAT-box核心元件,ABA响应元件ABRE、MYB元件、MYC元件、ERE元件、MeJA响应元件,BOX II、G-box、GT1-motif、I-BOX等光响应元件,以及分生组织响应元件等(表2)。
表 2 MaAQP1启动子元件分析Table 2. Element analysis on MaAQP1 promoter序号
Number结合位点
TFBs元件功能
Function of the motif器官
Organism序列
Sequence位置
Position1 AAGAA-motif LRE 燕麦
Avena sativaGAAAGAA/gGTAAAGAAA −1 172,−1 178 2 ABRE 脱落酸响应
Abscisic acid responsiveness拟南芥
Arabidopsis thalianaACGTG −55,−863 3 AT~TATA-box LRE 拟南芥
Arabidopsis thalianaTATATA −319,−343,−331,−355,−327,−353,−339,−363,
−323,−347,−335,−359,−329,−351,−341,−321,
−345,−333,−325,−337,−357,−349,−3614 Box II 光响应元件
light responsive element马铃薯
Solanum tuberosumTGGTAATAA −32 5 CAAT-box 核心启动子
Core promoter烟草/拟南芥/豌豆
Nicotiana glutinosa/Arabidopsis thaliana/Pisum sativumCAAT −43,−459,−315,−316,−125,−583,−1 197,
−208,−725,−421,−471,−1586 CAT-box 分生组织表达
meristem expression拟南芥
Arabidopsis thalianaGCCACT −254 7 CCAAT-box 结合位点
MYBHv1 binding site MYBHv1大麦
Hordeum vulgareCAACGG −935 8 CGTCA-motif 响应
MeJA-responsiveness MeJA大麦
Hordeum vulgareCGTCA −1 088 9 ERE 烯响应元件
ethylene-responsive element烟草
Nicotiana glutinosATTTTAAA −377,−775 11 GT1-motif 乙烯响应元件
light responsive element燕麦
Avena sativaGGTTAAT −65 12 I-box 光响应元件
light responsive element小麦/棉花
Triticum aestivum/
Gossypium hirsutumAAGATAAGGCT/AGATAAGG −611,−612 13 MYB SRE 拟南芥
Arabidopsis thalianaCAACCA −151 14 MYB recognition site SRE 拟南芥
Arabidopsis thalianaCCGTTG −878 15 MYC SRE 拟南芥
Arabidopsis thalianaCATTTG/CATGTG −111,−264 16 TATA-box 核心启动子元件
Core promoter element甘蓝/拟南芥
Brassica napus/ Arabidopsis thalianaATATAT/TATATA/taTATAAAtc/ATTATA −318,−326,−323,−320,−328,−324,−332,−319,
−327,−322,−321,−325,−330,−329,−333,−334,
−335,−336,−343,−344,−345,−346,−347,−348,
−349,−350,−351,−352,−353,−354,−355,
−356,−357,−358,−359,−363,−364,−365,
−389,−391,−837,−83817 TGACG-motif MeJA响应
MeJA-responsiveness大麦
Hordeum vulgareTGACG −58,−226 18 Unnamed__1 未知
unknown玉米
Zea maysCGTGG −864,−1 274 19 Unnamed__4 未知
unknown欧芹
Petroselinum hortenseCTCC −28,−1 016,−1 009,−1 353,−913,−1 255,
−1 013,−1 261,−92220 as-1 LRE 拟南芥
Arabidopsis thalianaTGACG −58,−226 2.3 诱饵载体的构建
取转化了诱饵载体的大肠杆菌菌液进行PCR扩增,将产物用1%琼脂糖凝胶进行电泳鉴定。并对扩增得到阳性条带的克隆质粒抽提和测序。测序结果正确,同时其菌落PCR结果如图2,条带正确,说明MaAQP1启动子片段连接到了表达载体pHIS2上。
2.4 诱饵载体转化酵母菌株及自激活检测
分别从3个不同转化反应的平板上各随机挑取3个单菌落,稀释后涂板至相应的不添加Histidine(组氨酸),添加不同浓度(25、50 mmol·L−1)的3AT(3-ami,2,4-triazole)缺陷型平板上,在30 ℃下培养3 d,结果如表3和图3所示。阳性对照随着3AT浓度增加,生长率会降低,与阴性对照有明显差别。阴性对照由于HIS3报告基因未被激活,在添加3AT的平板上生长明显减少,随着3AT浓度升高,转化子数量减少。自激活检测结果显示,在不添加3AT时,平板上的生长菌落3 424个,在分别添加了25 mmol·L−1和50 mmol·L−1 3AT的平板上,转化子的生长明显受到抑制,其生长比例与阴性对照的生长比例基本一致,表明其未激活HIS3报告基因。当添加25 mmol·L−1 3AT时,其生长受到抑制,菌落个数为408个,与对照的生长比例为11.9%,而当添加了50 mmol·L−1 3AT时,其菌落个数为68个,与对照的生长比例为1.9%。因此,后续试验可以用50 mmol·L−1 3AT进行。
表 3 自激活检测结果Table 3. Results of self-activation test反应
Reaction转化平板
Conversion plate检测平板
Detection plate检测内容
Test content3AT/(mmol·L−1) 0 25 50 生长数目
Growth number生长数目
Growth number25/0 生长比例
25/0 Growth ratio生长数目
Growth number50/0 生长比例
50/0 Growth ratio1 SD-T SD-TH 自激活检测
Self-activation detection3 424 408 11.9% 68 1.9% 2 SD-TL SD-TLH 阴性对照
Negative control3 968 1 376 34.6% 31 0.7% 3 SD-TL SD-TLH 阳性对照
Positive control5 024 5 136 102.2% 4 160 82.8% 2.5 cDNA文库的构建
将提取的干旱胁迫处理的香蕉总RNA,琼脂糖凝胶电泳检测,结果如图4中显示,总RNA的28S rRNA和18S rRNA条带清晰,条带28S亮度约为18S的2倍,说明总RNA质量较好,并无发生降解和污染。将RNA进行mRNA分离纯化,如图5所示,mRNA条带呈弥散状分布,分布均匀,mRNA总量为4.5 μg,可以满足建库需要。
使用同源重组的方法,将线性化的香蕉cDNA与经酶切处理线性化的pGADT7-Rec2(单杂交)载体混合共转化至大肠杆菌感受态中,取原液稀释后涂布于LB平板上(含氨苄抗性),第2 d计数,如图6所示,插入片段集中在500~2 000 bp,平均插入片段大于1 200 bp,阳性率为100%。在10 μL菌液稀释100倍后,取10 μL涂板,共生长了约250个克隆,其库容量为2.5×106 CFU·mL−1,共计5 mL的转化后原始菌液,则总库容量为1.25×107 CFU。
3. 讨论与结论
香蕉是大水大肥作物,干旱胁迫严重影响其生长发育及品质。目前的研究表明,水通道蛋白基因(AQP)作为膜蛋白能够增加植物的水分运输能力,提高植物的耐旱性。在拟南芥中,将AtPIP1; 2和AtPIP2; 2沉默会降低拟南芥抵御干旱的耐受性[13-14]。在水稻中过表达OsPIP1、OsPIP2和OsPIP1; 3能够增强植物对干旱胁迫的耐受性[15-16]。在烟草中过表达TaAQP7和TaAQP8可以提高植物对干旱的耐受性[3,9]。因此,研究AQP响应干旱胁迫的作用机制,对于进一步通过生物技术提高香蕉的耐旱能力具有重要的理论意义。本研究通过克隆干旱胁迫下的香蕉cDNA,构建用于酵母单杂交的cDNA文库,通过克隆香蕉水通道蛋白基因MaAQP1启动子,构建酵母诱饵表达载体,为下一步利用酵母单杂交筛选与香蕉MaAQP1互作的转录因子奠定了基础。
启动子在时间和空间意义上对于启动基因转录和调节基因表达都很重要。靶向基因及其启动子的遗传修饰可用于增强作物生长性能[17]。顺式元件与转录因子(TFs)之间的相互作用在转录的协调调控中对激活或抑制靶基因的表达起关键作用[18-19]。目前,可用于作物遗传改良的启动子很少。植物中最常见的启动子类型是35 s启动子CaMV35S,它可以在标记植物中驱动高水平的基因表达,而玉米泛素启动子则在单子叶植物中驱动基因表达。这些启动子能够在几乎所有组织和发育阶段的标志物或单子叶植物中驱动高水平的基因表达[20-22]。在特定条件下或某些组织中,诱导型或组织特异性启动子调节靶基因的表达。迄今为止,已经报道了一些植物的组织特异性和胁迫诱导型启动子。例如,拟南芥中的rd29A和rd29B启动子对高盐度和干旱有反应[23]。在小麦中,Dreb2启动子响应干旱胁迫[24]。来自芥菜植物的BjSOS2基因启动子在高盐,干旱和非生物胁迫以及其他形式的胁迫下起作用[25]。硬粒小麦基因TdPIP2; 1水稻及其启动子区域响应非生物胁迫[26]。本研究通过PlantCARE应用程序分析了MaAQP1启动子的顺式作用元件,元件包含了多个TATA-box和CAAT-box核心元件,以及与ABA和MeJA响应,MYB,MYC和光响应相关元件。TATA盒是在转录起始位点表达基因的核心启动子元件。CAAT-box是具有增强子活性的启动子中常见的顺式作用元件。但是,在MaAQP1启动子中未发现与干旱胁迫相关的反应元件。本研究发现ABRE是脱落酸(ABA)的顺式作用元件。研究表明ABA是一种重要的植物激素,参与许多重要的生物过程和反应,包括非生物胁迫。启动子的克隆及元件分析为下一步运用酵母单杂交鉴定直接调节MaAQP1启动子的转录因子,以及靶向转录因子调节MaAQP1表达的分子机制奠定了基础。
酵母单杂交cDNA文库的构建是酵母单杂交试验进行的基础,文库的质量好坏直接影响筛库过程。而cDNA文库的质量又是由转录成cDNA的mRNA决定的,因此必须对其质量进行严格的把关。本研究利用CTAB法提取香蕉组织RNA,RNA条带清晰无降解,进一步利用试剂盒分离纯化mRNA,合成香蕉cDNA,构建的cDNA文库,符合进一步试验的要求。另外,库容量以及插入片段的大小也直接影响到后期利用cDNA文库进行互作基因筛选的效率及结果,相关的研究表明,构建的木薯cDNA文库库容为1.5×106 CFU,插入片段大小为250~3 000 bp,能够为进一步的酵母单杂交筛选奠定良好的基础[27]。在本研究中,构建的文库库容为1.25×107 CFU,插入片段平均在1 200 bp左右,完全满足试验的要求。
-
图 5 抗感青枯病桑树根际细菌预测得到的COG相对丰度
注:A: RNA加工与修饰; B: 染色体结构与动态;C: 能量产生与转化; D: 细胞周期控制,细胞分裂,染色体分裂; E: 氨基酸转运与代谢; F: 核酸转运与代谢;G:碳水化合物的运输与代谢; H: 酶运输与代谢; I: 脂质运输与代谢; J:翻译,核糖体结构和生物发生; K: 转录; L: 复制,重组和修复; M: 细胞壁/膜/包膜生物发生; N: 细胞运动; O: 翻译后修饰,蛋白质更新,伴侣; P: 无机离子的运输与代谢; Q:次生代谢产物的生物合成,转运和分解代谢; R:仅通用功能预测; S:未知功能; T:信号转导机制; U:细胞内运输,分泌和囊泡运输; V: 防御机制; W: 细胞外结构; Z: 细胞骨架。
Figure 5. Relative abundance of COG at QZ2K and QZ2G
Note: A: RNA processing and modification; B: Chromatin structure and dynamics; C: Energy production and conversion; D: Cell cycle control, cell division, and chromosome partitioning; E: Amino acid transport and metabolism; F: Nucleotide transport and metabolism; G: Carbohydrate transport and metabolism; H: Coenzyme transport and metabolism; I: Lipid transport and metabolism; J: Translation, ribosomal structure and biogenesis; K: Transcription; L: Replication, recombination, and repair; M: Cell wall/membrane/envelope biogenesis; N: Cell motility; O: Posttranslational modification, protein turnover, and chaperones; P: Inorganic ion transport and metabolism; Q: Secondary metabolites biosynthesis, transport, and catabolism; R: General function prediction only; S: Function unknown; T: Signal transduction mechanisms; U: Intracellular trafficking, secretion, and vesicular transport; V: Defense mechanisms; W: Extracellular structures; Z: Cytoskeleton.
表 1 高通量测序数据概况
Table 1 Overview of high-throughput sequencing data
样品
Sample高质量序列
Clean tags优质序列
Valid tags平均长度 /bp
Valid mean length测序深度指数/%
Goods coverageQZ2K.1 33 883 28 948 429 94.81 QZ2K.2 38 786 32 371 430 94.69 QZ2K.3 32 328 26 941 432 94.57 QZ2K.4 41 499 34 267 432 94.56 平均值 average 36 624.0 30 631.8 430.8 94.658 QZ2G.1 38 747 32 496 432 94.53 QZ2G.2 37 626 31 688 432 94.43 QZ2G.3 40 749 34 814 432 94.78 QZ2G.4 40 656 34 090 432 95.22 平均值 average 3 944.5 33 272.0 432.0 94.740 表 2 抗感青枯病桑树根际土壤细菌α多样性指数(平均值±标准误,n=4)
Table 2 Alpha diversity indices of bacteria in rhizosphere soils at QZ2K and QZ2G(Mean±SE,n=4)
样品
Samples谱系多样性指数
PD whole TreeChao1 文库覆盖率(%)
Goods coverage观测物种数
Observed Species香浓-威纳指数
Shannon-Wiener辛普森指数
SimpsonQZ2K 84.97±0.77 3 190.57±22.48 0.95±0.00 2 333.88±40.49 9.51±0.09 0.995±0.001 QZ2G 82.86±1.44 3 178.61±87.45 0.95±0.00 2 304.30±49.58 9.65±0.03 0.997±0.001 -
[1] XIONG W, SONG Y Q, YANG K M, et al. Rhizosphere protists are key determinants of plant health [J]. Microbiome, 2020, 8: 27. DOI: 10.1186/s40168-020-00799-9
[2] RODRIGUEZ P A, ROTHBALLER M, CHOWDHURY S P, et al. Systems biology of plant-microbiome interactions [J]. Molecular Plant, 2019, 12(6): 804−821. DOI: 10.1016/j.molp.2019.05.006
[3] BERENDSEN R L, VISMANS G, YU K, et al. Disease-induced assemblage of a plant-beneficial bacterial consortium [J]. The ISME Journal, 2018, 12(6): 1496−1507. DOI: 10.1038/s41396-018-0093-1
[4] ROUT M E, SOUTHWORTH D. The root microbiome influences scales from molecules to ecosystems: The unseen majority1 [J]. American Journal of Botany, 2013, 100(9): 1689−1691. DOI: 10.3732/ajb.1300291
[5] KWAK M J, KONG H G, CHOI K, et al. Rhizosphere microbiome structure alters to enable wilt resistance in tomato [J]. Nature Biotechnology, 2018, 36(11): 1100−1109. DOI: 10.1038/nbt.4232
[6] ROLFE S A, GRIFFITHS J, TON J. Crying out for help with root exudates: adaptive mechanisms by which stressed plants assemble health-promoting soil microbiomes [J]. Current Opinion in Microbiology, 2019, 49: 73−82. DOI: 10.1016/j.mib.2019.10.003
[7] WEI Z, GU Y A, FRIMAN V P, et al. Initial soil microbiome composition and functioning predetermine future plant health [J]. Science Advances, 2019, 5(9): eaaw0759. DOI: 10.1126/sciadv.aaw0759
[8] 颜朗, 张义正, 方志荣, 等. 不同马铃薯基因型对根际细菌群落结构的影响 [J]. 四川大学学报(自然科学版), 2020, 57(2):383−390. YAN L, ZHANG Y Z, FANG Z R, et al. Effects of potato genotype on rhizososphere bacterial community structure [J]. Journal of Sichuan University (Natural Science Edition), 2020, 57(2): 383−390.(in Chinese
[9] LAREEN A, BURTON F, SCHÄFER P. Plant root-microbe communication in shaping root microbiomes [J]. Plant Molecular Biology, 2016, 90(6): 575−587. DOI: 10.1007/s11103-015-0417-8
[10] 蔡秋华, 左进香, 李忠环, 等. 抗性烤烟品种根际微生物数量及功能多样性差异 [J]. 应用生态学报, 2015, 26(12):3766−3772. CAI Q H, ZUO J, LI Z H, et al. Difference of rhizosphere microbe quantity and functional diversity among three flue-cured tobacco cultivars with different resistance [J]. Chinese Journal of Applied Ecology, 2015, 26(12): 3766−3772.(in Chinese
[11] MENDES L W, RAAIJMAKERS J M, DE HOLLANDER M, et al. Influence of resistance breeding in common bean on rhizosphere microbiome composition and function [J]. The ISME Journal, 2018, 12(1): 212−224. DOI: 10.1038/ismej.2017.158
[12] 曹梦琪. 桑树青枯病病原菌的分离鉴定及其活体检测方法的建立 [D]. 镇江: 江苏科技大学, 2017. CAO M Q. Isolation, Identification and Establishment of Viable Cells Detection of the Pathogen of Mulberry Bacterial Wilt [D]. Zhenjiang: Jiangsu University of Science and Technology, 2017. (in Chinese).
[13] 李磊. 华南地区桑树青枯病原菌的收集鉴定及侵染机制的初步研究 [D]. 镇江: 江苏科技大学, 2017. LI L. Collecting for identification of the pathogen of Mulberry Wilt Disease in South China and a preliminary study on the mechanism of infection [D]. Guangzhou: Jiangsu University of Science and Technology, 2017. (in Chinese).
[14] 王树昌, 耿涛, 黄华平. 海南蚕桑 [M]. 海口: 南海出版公司, 2017: 68-80. [15] 黄富, 张伟国, 黄胜, 等. 桑树抗青283×抗青10杂交组合的育成 [J]. 广东蚕业, 2006, 4(4):24−28. HUANG F, ZHANG WG, HUANG S, et al. Breeding of mulberry Kangqing 283 × Kangqing 10 [J]. Guangdong Sericulture, 2006, 4(4): 24−28.(in Chinese
[16] 朱方荣. 桂桑优62和桂桑优12的特点及栽培 [J]. 广西农业科学, 2001(4):199. ZHU F R. Characteristics and cultivation of Guisangyou 62 and Guisangyou 12 [J]. Guangxi Agricultural Sciences, 2001(4): 199.(in Chinese
[17] NOSSA C W. Design of 16S rRNA gene primers for 454 pyrosequencing of the human foregut microbiome [J]. World Journal of Gastroenterology, 2010, 16(33): 4135. DOI: 10.3748/wjg.v16.i33.4135
[18] 邱洁, 徐丽丽, 钱叶, 等. 不同品种桑树根际土壤细菌群落及土壤理化性质的研究 [J]. 蚕业科学, 2017, 43(4):568−576. QIU J, XU L L, QIAN Y, et al. An investigation on rhizospheric bacterial community and soil physical and chemical properties of different mulberry varieties [J]. Acta Sericologica Sinica, 2017, 43(4): 568−576.(in Chinese
[19] BHATTI A A, HAQ S, BHAT R A. Actinomycetes benefaction role in soil and plant health [J]. Microbial Pathogenesis, 2017, 111: 458−467. DOI: 10.1016/j.micpath.2017.09.036
[20] 向立刚, 周浩, 汪汉成. 健康与感染青枯病烟株根际土壤与茎秆细菌群落结构与多样性 [J]. 微生物学报, 2019, 59(10):1984−1999. XIANG LG, ZHOU H, WANG H C. Bacterial community structure and diversity of rhizosphere soil and stem of healthy and bacterial wilt tobacco plants [J]. Acta Microbiologica Sinica, 2019, 59(10): 1984−1999.(in Chinese
[21] KYSELKOVÁ M, KOPECKÝ J, FRAPOLLI M, et al. Comparison of rhizobacterial community composition in soil suppressive or conducive to tobacco black root rot disease [J]. The ISME Journal, 2009, 3(10): 1127−1138. DOI: 10.1038/ismej.2009.61
[22] ADHIKARI T B, JOSEPH C M, YANG G P, et al. Evaluation of bacteria isolated from rice for plant growth promotion and biological control of seedling disease of rice [J]. Canadian Journal of Microbiology, 2001, 47(10): 916−924. DOI: 10.1139/w01-097
[23] HUANG A C, JIANG T, LIU Y X, et al. A specialized metabolic network selectively modulates Arabidopsis root microbiota [J]. Science, 2019, 364(6440): 1−9.
[24] COTTON T E A, PÉTRIACQ P, CAMERON D D, et al. Metabolic regulation of the maize rhizobiome by benzoxazinoids [J]. The ISME Journal, 2019, 13(7): 1647−1658. DOI: 10.1038/s41396-019-0375-2
[25] BULGARELLI D, ROTT M, SCHLAEPPI K, et al. Revealing structure and assembly cues for Arabidopsis root-inhabiting bacterial microbiota [J]. Nature, 2012, 488(7409): 91−95. DOI: 10.1038/nature11336
[26] VÍCTOR J C, JUAN P J, VIVIANE C, et al. Pathogen-induced activation of disease - suppressive functions in the endophytic root microbiome [J]. Science, 2019, 366(6465): 606−612. DOI: 10.1126/science.aaw9285
-
期刊类型引用(2)
1. 李佳思,刘迎庆,张永恒,张迎澳,肖烨子,刘露,余有本. 茶树CsNCED2启动子互作转录因子筛选及在非生物胁迫中的响应. 茶叶科学. 2023(03): 325-334 .  百度学术
百度学术
2. 赵彩良,张洁,唐锐敏,贾小云. 甘薯块根cDNA酵母文库的构建及IbNCED3启动子互作蛋白的筛选鉴定. 山西农业大学学报(自然科学版). 2022(04): 19-27 .  百度学术
百度学术
其他类型引用(2)




 下载:
下载: