Genome-wide Identification and Expression Analysis of Metacaspase Gene Family in strawberry
-
摘要:目的 精氨酸/赖氨酸特异性半胱氨酸酶(metacaspases,MCs)在植物生长发育的细胞程序性死亡(programmed cell death,PCD)中发挥重要的调控作用,为系统鉴定草莓FaMC基因家族,进行草莓FaMC基因家族全基因组鉴定及表达分析。方法 通过草莓全基因组数据分析FaMC蛋白的理化性质、结构域、系统发育关系和保守基序,采用qRT-PCR技术对草莓6个组织器官和6个果实发育时期中的FaMC基因进行表达分析。结果 蛋白序列分析表明,草莓基因组中21个FaMC成员分为3种类型:I型、I*型和II型。系统发育树和保守结构域分析表明:I型FaMC含N端前结构域且包含1个锌指结构;I*型FaMC含N端前结构域,但其中没有锌指结构;II型FaMC不含N端前结构域。基因表达分析发现,FaMC基因在不同组织中存在表达差异性,I型、I*型和II型FaMC基因分别在叶片、根和花中高表达。在果实发育过程中,多数I*型FaMC基因的表达水平呈现出高-低-高的动态变化趋势;部分I型、II型FaMC在果实发育过程中呈现相反的表达模式。结论 FaMC基因可能在草莓生长过程中发挥重要作用。
-
关键词:
- 草莓 /
- metacaspase /
- 细胞程序性死亡(PCD) /
- 基因组 /
- 表达分析
Abstract:Objective To identify and study the genes in strawberry (Fragaria ananassa) which related to the synthesis of metacaspases (MCs), the enzymes that played vital roles in regulating the programmed cell death (PCD) in plants.Method The physiochemical properties, conserved domains, phylogenetic relationships and conserved motifs of FaMC proteins were analyzed based on strawberry whole genome data. The expressions of FaMC genes in 6 tissues and 6 fruit development stages of strawberry were carried out by qRT-PCR.Result The 21 FaMC proteins identified in this study could be divided into Type-I, Type-I*, and Type-II based on the differences on their sequences. The phylogenetic trees and conserved domains of the 3 different types showed the differentiations between them. Type-I FaMC protein consisted of N-terminal domain (NTD) with one zinc finger motif, Type-I* consisted of NTD but no zinc finger, and Type-II had not NTD. The FaMC genes were distinctively differently expressed in different tissues and developmental stages. Type-I expressed in the leaves, Type-I* in the roots, and Type-II in the flowers. And, during the berry development stage, most Type-I* exhibited a high-low-high U-pattern of changes, whereas, part of Type-I and Type-II had a reversed U-pattern.Conclusion FaMC genes were identified to be closely related to the synthesis of metacaspases that played important roles in the development and growth of strawberry plants.-
Keywords:
- Strawberry /
- metacaspase /
- programmed cell death /
- genome /
- expression analysis
-
0. 引言
【研究意义】鹿茸菇,学名荷叶离褶伞[Lyophyllum decastes (Fr.) Singer][1],是一种珍稀的药食两用型高等真菌。鹿茸菇口感爽脆、味道清淡,备受老百姓的喜爱,成为“网红菇”[2]。国内市场对于鹿茸菇的巨大需求,推动了鹿茸菇产业的快速发展,使其成为继金针菇、杏鲍菇、真姬菇、双孢蘑菇之后的新型食用菌工厂化栽培品种,生产基地主要聚集于上海、江苏、浙江、福建等地相关食用菌企业。菌丝培养阶段在整个食用菌栽培过程中十分重要。鹿茸菇菌丝在栽培基质中萌发时间长,抗杂菌能力弱[3],易发生有害菌侵害,污染率高,后期出菇产量低甚至不出菇[4]。目前鹿茸菇工厂化栽培多以固体菌种为主,传统固体菌种存在萌发时间长、制种周期长、菌龄不一致[5]、污染率高等弊端,而液体菌种培养工序简单[6],扩繁迅速,短时间内可达到一定的生物量[7-9],菌球更易分散,菌丝萌发快[10]。液体菌种较固体菌种更适用于鹿茸菇工厂化生产,因此急需开展鹿茸菇液体菌种配方优化研究。【前人研究进展】第二次世界大战期间,为了满足对抗生素的大量需求,发酵罐制造业开始兴起,同时解决了液体深层发酵的供氧问题,奠定了现代发酵工业的基础。Humfeld [11]首次使用液体深层发酵技术成功制备Agaricu campestris菌丝体。随着发酵工程理论知识与实践经验的不断积累,液体深层发酵技术现已广泛应用于大多数食用菌品种[5](除混合种出菇类型,如银耳)生产用种的制备。与固体菌种相比,液体菌种优势更明显,应用前景更广阔[12],制备工艺具备完全替代固体菌种的潜能。关于鹿茸菇方面的研究主要聚焦于鹿茸菇菌丝在平板培养基上的生物学特性,包括不同碳源、氮源、金属离子、生长素对鹿茸菇平板菌丝生长的影响[13-14],摇瓶试验分析不同培养基成分对鹿茸菇液体菌丝生长的影响[15],通气搅拌式发酵罐中优化鹿茸菇液体菌丝发酵条件,液体菌种配方优化等[16]。张汉燚等[17]对鹿茸菇中试发酵培养基进行优化,最佳培养基配方为玉米面5%、大豆0.5%、ZnSO4 0.025%、MgSO4 0.05%、KH2PO4 0.05%。发酵至第8 天时,菌丝体生物量达到最大值(10.578 g·L−1)。席亚丽等[18]对鹿茸菇摇瓶发酵条件进行探索,获得最佳培养基配方为玉米面200 g·L−1、蔗糖 15 g·L−1、麸皮35 g·L−1、酵母膏1.5 g·L−1;温度26℃,接种量10%,培养时间为6~8 d。【本研究切入点】鹿茸菇液体菌种培养优化的研究多采用单因素和正交试验结合的方式,试验设计考虑多种因素的影响,但无法明确因素之间交互作用对响应值的影响,缺少各因素与响应值之间的函数关系,无法确定最大响应值所对应的因素组合方式。目前,单因素试验和Box-Behnken响应面法结合优化鹿茸菇液体菌种发酵配方的研究较少,鹿茸菇液体发酵培养工厂化生产效率有待提高。【拟解决的关键问题】本研究以鹿茸菇为研究材料,采用摇瓶培养方式,以提高菌丝生物量为目的,采用单因素试验和Box-Behnken响应面法结合的方式,对培养基碳源、氮源和无机盐的添加量进行优化,以期获得适合鹿茸菇菌丝生长的液体发酵培养基配方,为推动鹿茸菇液体菌种生产提供理论依据。
1. 材料与方法
1.1 试验材料
1.1.1 供试菌株
鹿茸菇菌种(编号:LDS-1-YJY-2018)保存于福建农林大学(古田)菌业研究院,为福建珍菌子生物科技有限公司生产菌种。
1.1.2 试剂
酵母粉、蛋白胨均购于Oxoid;琼脂粉购于Chembase;葡萄糖、蔗糖、鱼粉、维生素B1、K2HPO4、MgSO4·7H2O、ZnSO4、CaSO4等无机盐均购于国药集团化学试剂有限公司;供试碳源玉米粉、小麦粉、全麦粉均购于陇龙之家,玉米芯购于联丰农产品深加工;供试氮源豆粕粉、牛肉膏、黄豆饼粉、花生饼粉、棉籽饼粉、玉米浆均购于鸿润宝顺培养基原料厂家。
1.1.3 培养基配方
PDA加富液体培养基(1 L):土豆200 g,葡萄糖20 g,蛋白胨2 g,酵母粉2 g,K2HPO4 2.5 g,MgSO4·7H2O 1 g,Vitamin B1 0.1 g,pH自然。PDA加富固体培养基在液体培养基的基础之上加入琼脂20 g。
摇瓶一级种子液培养基(1 L):葡萄糖20 g,蛋白胨2 g,酵母粉2 g,K2HPO4 2.5 g,MgSO4·7H2O 1 g,pH自然。
摇瓶基础发酵培养基(1 L):葡萄糖20 g,蛋白胨2 g,K2HPO4 2.5 g,MgSO4·7H2O 1 g,pH自然。
固体菌种培养基:木屑78%,麸皮20%,葡萄糖1%,石灰1%,含水率65%左右。
1.2 试验方法
1.2.1 摇瓶一级种子液的制备
将保藏菌种接种于PDA加富固体培养基平板,25℃,培养20 d左右直至菌丝长满平板。应用打孔器(0.5 mm直径)在平板上面打30孔,应用接种器将菌块挑入摇瓶一级种子液培养基(含磁力搅拌子),25℃,150 r·min−1培养4 d,第4 天取出摇瓶置于磁力搅拌器上以800 r·min−1、30 min打散菌球,继续培养4 d,即制成一级种子液。
1.2.2 液体菌种生长动力学曲线的测定
按10%接种量将一级种子液接种于100 mL基础发酵培养基,25℃、150 r·min−1振荡培养。每隔1 d分别取出发酵液,100目纱布过滤收集菌球,RO水清洗3遍,60℃烘干至恒重。每个处理设置3个重复。
1.2.3 最适液体发酵培养基成分筛选
基础发酵培养基121℃,灭菌20 min。其中玉米芯、玉米粉、小麦粉、全麦粉、豆粕粉、黄豆饼粉、花生饼粉、棉籽饼粉、鱼粉需115℃灭菌40 min,冷却后均要求过100目筛网,滤液加入基础发酵培养基。按10%接种量接入相应培养基,25℃,150 r·min−1,培养8 d,100目纱布过滤收集菌球,RO水清洗3遍,60℃烘干至恒重。每个处理设置3个重复。该操作方法适用于1.2.3、1.2.4与1.2.5。
以摇瓶基础发酵培养基为基础,分别用不同种类的碳源、氮源、无机盐替代摇瓶基础发酵培养基相对应的培养基成分,以筛选最适合鹿茸菇液体菌球生长的碳源、氮源与无机盐。供试碳源(20 g·L−1):葡萄糖、蔗糖、玉米芯、玉米粉、小麦粉、全麦粉,CK为不加碳源;供试氮源(2 g·L−1):蛋白胨、豆粕粉、牛肉膏、黄豆饼粉、花生饼粉、棉籽饼粉、鱼粉、玉米浆,CK为不加氮源;供试无机盐(1 g·L−1):CoCl2、CuSO4、ZnSO4、CaSO4、MnSO4、FeSO4、NaCl、KCl、MgSO4·7H2O、K2HPO4,CK为不加无机盐。
1.2.4 单因素设计
以摇瓶基础发酵培养基为基础,分别替换其中碳源、氮源、无机盐成分进行单因素试验。各因素质量浓度梯度如下:全麦粉为10、20、30、40、50、60、70 g·L−1;花生饼粉为2、6、10、14、18、22、26 g·L−1;K2HPO4为 0.5、1.0、1.5、2.0、2.5、3.0 g·L−1;MgSO4·7H2O为0.5、1.0、1.5、2.0、2.5、3.0 g·L−1。
1.2.5 响应面设计试验
基于单因素设计结果,以菌丝生物量为响应值评价指标,采用Box-Behnken响应面法对全麦粉(A)、花生饼粉(B)、磷酸氢二钾(K2HPO4,C)、七水硫酸镁(MgSO4·7H2O,D)进行4因素3水平优化设计试验(表1)。
表 1 Box-Behnken设计因素水平及编码Table 1. Codes and levels of factors in Box-Behnken experiment水平
Level因素 Factors/(g·L−1) A 全麦粉
Whole wheat flourB 花生饼粉
Peanut mealC 磷酸氢二钾
K2HPO4/D七水硫酸镁
MgSO4·7H2O/+1 55 24 2.25 2.25 0 50 22 2.00 2.00 −1 45 20 1.75 1.75 1.2.6 液体菌种和固体菌种出菇试验
按照最适发酵培养基配方制备液体菌种,以10%的接种量接种于250 mL摇瓶内,24℃ 130 r·min−1培养8 d,获得液体菌种。采用袋口接种的方式将液体栽培种接种至菌袋内,每袋接种25~30 mL,接种固体菌种作为对照组。养菌与出菇管理工艺均由福建珍菌子生物科技有限公司完成。
1.3 统计分析
培养基成分筛选试验和单因素试验使用Graphpad Prism 9软件作图。响应面试验使用Design Expert 8.0.6软件进行。试验数据均使用SPSS Statistics 26软件进行显著性差异分析。
2. 结果与分析
2.1 鹿茸菇液体菌种生长动力学曲线
由图1可知,鹿茸菇菌丝生物量随培养时间的延长呈先上升后下降的趋势。在发酵过程中,0~2 d为延滞期,2~8 d为快速生长期,8~10 d为稳定期,10 d后进入衰亡期。当鹿茸菇菌丝培养至第8 天时,菌丝生物量达到最大值(4.47 g·L−1),因此确定鹿茸菇液体菌种培养时间为8 d。
2.2 培养基成分的筛选
由图2-A可知,鹿茸菇菌丝在6种供试碳源中均可生长,且菌丝生物量均高于不加碳源的对照组。不同碳源对菌丝生物量的影响具有显著差异性,从高到低的顺序为:全麦粉>葡萄糖>蔗糖>小麦粉>玉米粉>玉米芯,以全麦粉作为碳源时,菌丝生物量达到最大值(8.50 g·L−1),显著高于其他碳源。由图2-B可知,鹿茸菇菌丝在8种供试氮源中均可生长,其中花生饼粉是供试氮源中最适宜鹿茸菇生长的复合氮源,以花生饼粉作为氮源时,菌丝生物量可达11.12 g·L−1。由图2-C可知,当Co2+与Cu2+质量浓度为1 g·L−1时,菌丝生物量均低于不加无机盐的对照组,抑制鹿茸菇菌丝的正常生长,而其余离子均不同程度地促进菌丝的生长发育,其中尤以K+与Mg2+最为显著。以上数据表明,较适合于鹿茸菇菌丝发酵的碳源、氮源、无机盐分别为全麦粉、花生饼粉、K2HPO4与MgSO4·7H2O。
2.3 单因素试验
为了探究培养基单一成分如何影响鹿茸菇液体菌丝的生长,分别开展了全麦粉、花生饼粉、K2HPO4与MgSO4·7H2O单因素质量浓度梯度试验。以全麦粉作为培养基的碳源,质量浓度为40~60 g·L−1时,菌丝生物量达到最大值(13.10 g·L−1)(图3-A)。以花生饼粉作为培养基的氮源时,质量浓度为18~26 g·L−1时,菌丝生物量达到最大值(24.65 g·L−1)(图3-B)。以K2HPO4与MgSO4作为培养基的无机盐时,K2HPO4与MgSO4·7H2O的质量浓度均为1.5~2.5 g·L−1,菌丝生物量分别达到其最大值(图3-C、D)。在不考虑培养基成分之间是否存在交互作用的影响下,单因素设计已成功筛选到各培养基成分较适合鹿茸菇菌丝生长的质量浓度范围。
2.4 响应面优化试验
在单因素试验结果的基础上,采用Box-Behnken设计对碳源(全麦粉)、氮源(花生饼粉)、无机盐(K2HPO4与MgSO4·7H2O)进行4因素3水平响应面优化试验,结果见表2。以因素为自变量,菌丝生物量为响应值,使用Design Expert 8.0.6软件进行多元线性回归和二次项拟合,可得到二次多项回归方程式(1),方差分析见表3。
表 2 Box-Behnken试验设计及试验结果Table 2. Design and results of Box-Behnken experiment试验号
NumberA全麦粉
Whole wheat
flourB花生饼粉
Peanut
mealC磷酸氢
二钾
K2HPO4D七水
硫酸镁
MgSO4·7H2O菌丝生物量
Mycelium
biomass/
(g·L−1)1 −1 −1 0 0 29.61±1.18 2 +1 −1 0 0 29.90±1.45 3 −1 +1 0 0 32.27±1.11 4 +1 +1 0 0 28.77±2.01 5 0 0 −1 −1 30.79±1.28 6 0 0 +1 −1 29.41±1.88 7 0 0 −1 +1 28.00±0.30 8 0 0 +1 +1 30.21±1.09 9 −1 0 0 −1 31.72±2.12 10 +1 0 0 −1 29.37±1.63 11 −1 0 0 +1 30.20±2.00 12 +1 0 0 +1 29.11±2.51 13 0 −1 −1 0 29.29±4.29 14 0 +1 −1 0 30.00±0.68 15 0 −1 +1 0 30.20±1.45 16 0 +1 +1 0 30.11±4.80 17 −1 0 −1 0 30.00±2.97 18 +1 0 −1 0 30.17±4.12 19 −1 0 +1 0 32.48±0.21 20 +1 0 +1 0 28.73±4.01 21 0 −1 0 −1 30.09±4.38 表 3 二次回归方程方差分析Table 3. ANOVA on quadratic regression equation来源
Source平方和
SS自由度
DS均方
MSF P 显著性
Significance模型
Model0.7362 14 0.0526 98.95 <0.0001 ** A 0.0878 1 0.0878 165.28 <0.0001 ** B 0.0127 1 0.0127 23.85 0.0002 ** C 0.0074 1 0.0074 13.96 0.0022 ** D 0.0248 1 0.0248 0.31 <0.0001 ** AB 0.0358 1 0.0358 0.02 <0.0001 ** AC 0.0371 01 0.0371 6.12 <0.0001 * AD 0.0044 1 0.0044 0.32 0.0118 * BC 0.0015 1 0.0015 0.08 0.1114 BD 0.0011 1 0.0011 0.02 0.1702 CD 0.0327 1 0.0327 0.03 <0.0001 ** A2 0.1299 1 0.1299 169.26 <0.0001 ** B2 0.1974 1 0.1974 9.19 <0.0001 ** C2 0.2045 1 0.2045 127.92 <0.0001 ** D2 0.2302 1 0.2302 37.54 <0.0001 ** 残差
Residual0.0074 14 0.0005 失拟项
Lack of fit0.0047 10 0.0005 0.69 0.7126 纯误差
Pure error0.0027 4 0.0007 总和
Cor total0.7436 28 R2=0.9900 R2adj=0.9800 *表示差异显著(P<0.05),**表示差异极显著(P<0.01)。
* indicates significant difference at P<0.05; ** extremely significant at P<0.01.Y=3.34−0.086A+0.21B−0.098C+0.020D+0.0092AB−0.16AC−0.036AD+0.018BC+0.01BD−0.011CD+0.65A2+0.15 B2+0.56C2+0.31D2。 通过表3方差分析表可知,模型P<0.01,说明回归方程在0.01水平极显著,表明试验设计可靠;失拟项P>0.05,说明所得方程与实际拟合非正常误差所占比例较小,可用该模型和回归方程来预测试验数据。R2=0.9900,R2adj=0.9800,进一步说明该模型拟合度较好,可信度高,约98.00%的鹿茸菇液体菌种生物量可用该模型进行预测[19]。A、B、C、D的P值小于0.01,说明其对菌丝生物量有极显著影响。AB、CD之间交互作用对菌丝生物量有极显著影响,AC、AD有显著影响,BD、BC无显著影响。比较F值大小可知,影响菌丝生物量的因素主次顺序为A全麦粉>B花生饼粉>C磷酸氢二钾>D硫酸镁,交互项影响顺序为AC>AD>BC>CD>AB=BD。
响应曲面图可更加直观地反映两两因素之间交互作用情况,等高线的形状为椭圆形表示交互作用明显,圆形则表示交互作用不明显[20]。在全麦粉质量浓度为45~52 g·L−1与花生饼粉质量浓度为21.2~23.5 g·L−1、全麦粉质量浓度为45.0~51.5 g·L−1与K2HPO4 质量浓度为1.9~2.2 g·L−1、全麦粉质量浓度为45.2~51.5 g·L−1与MgSO4·7H2O 质量浓度为1.8~2.1 g·L−1,菌丝生物量均出现极大值(图4-A~C);全麦粉与花生饼粉、碳酸氢二钾均存在显著的交互作用(表3)。其余因素之间几乎不存在交互作用,主要由单因素自身决定菌丝生物量(表3、图4)。
2.5 响应面验证试验
鹿茸菇液体菌种发酵培养基响应面优化试验中,最佳组合为全麦粉47.84 g·L−1、花生饼粉22.39 g·L−1、K2HPO4 2.04 g·L−1、MgSO4·7H2O 1.97 g·L−1,预测菌丝生物量为33.60 g·L−1。考虑实际情况,将各组用量简化为全麦粉47 g·L−1、花生饼粉22 g·L−1、K2HPO4 2.00 g·L−1、MgSO4·7H2O 2.00 g·L−1,此条件下的菌丝生物量为(32.81±1.10) g·L−1。实际值与预测值相差较小,说明本研究建立的模型可对菌丝生物量进行预测,试验设计有效可靠。
2.6 液体菌种与固体菌种出菇试验
如表4所示,液体菌种制种周期仅需16 d,较固体菌种缩短了48 d。液体菌种接种后存在多个萌发位点,2 d后观察到菌丝萌发;固体菌种是从接种块向四周生长,萌发点较少,需4 d才能萌发。鹿茸菇菌丝在栽培基质中萌发时间长,发菌较慢,较其他食用菌更易被杂菌污染,抗杂菌能力更弱,污染率高,后期出菇产量低甚至不出菇。固体菌种萌发时间长、菌丝生长速率低,污染率达29.17%,远高于液体菌种。液体菌种在菌种制备和栽培周期方面均具有优势。
表 4 液体菌种和固体菌种栽培过程及出菇比较Table 4. Comparison of fermentation processes andmushroom fruiting using liquid and solid media指标
Index液体菌种
Liquid strain固体菌种
Solid strain原种培养时间 Original culture time/d 8 24 栽培种培养时间 Culture spawn incubation time/d 8 40 制种周期 Seed production cycle/d 16 64 菌丝萌发时间 Time of mycelium germination/d 2 4 满袋时间 Bags full time/d 42 45 污染率 Pollution rate/% 8.33 29.17 现蕾时间 Budding time/d 7 9 采收时间 Harvest time/d 25 27 单包产量 Single package output/g 500.89±20.12 432.00±25.36 子实体整齐度 Fruiting body uniformity +++ ++ 菇长 Length/cm 12.82±1.19 11.76±0.83 菇径 Size/mm 12.37±1.66 17.36±1.71 盖高 Height/mm 6.86±1.35 10.72±1.85 盖径 Diameter/mm 20.06±3.77 26.10±3.94 +++表示子实体整齐度较一致; ++表示子实体整齐度一致。
+++shows uniform fruiting body formation; ++acceptable fruiting body uniformity.如表4与图5所示,接种液体菌种的子实体整齐度较一致,单包产量和成品菇数量更高,产量较固体菌种提高15.74%。子实体农艺性状方面,接种液体菌种的子实体菇长略高于固体,而固体菌种的菇径、盖高和盖径均高于液体菌种,菌柄粗、菌盖大会影响荷叶离褶伞的口感,降低商品价值。
综合分析液体菌种和固体菌种的培养周期、出菇产量、子实体农艺性状等方面,液体菌种表现优异。因此采用本研究的培养基配方所制备的液体菌种可用于鹿茸菇工厂化栽培。
3. 讨论与结论
本研究经过分析4种廉价的复合碳源与2种昂贵的单一碳源、7种廉价的复合氮源与1种昂贵的复合氮源,发现廉价型的全麦粉与花生饼粉具有作为鹿茸菇液体菌种发酵主料的潜质。此外,本项研究发现鹿茸菇菌丝可利用的碳源、氮源种类极广,但更倾向于利用复合型碳氮源,与魏生龙等[14]研究结果较一致 。在本研究中,玉米粉与小麦粉作为碳源时,鹿茸菇菌丝生物量无显著性差异,该项结果与席亚丽等[18]的研究结果较一致 ,但本研究结果表明全麦粉作为碳源培养鹿茸菇液体菌丝的效果是小麦粉的2.2倍,说明麦壳和麸皮也具有促进液体菌丝生长发育的功能[17]。分析无机盐对鹿茸菇菌丝生长的影响,发现除Co2+与Cu2+显著抑制菌球生长之外,其他供试离子均具有正效应。该研究结果与魏生龙等[14]的研究结果存在一定差异,原因可能在于鹿茸菇平板菌丝与液体菌丝生长发育对离子的吸收利用情况不同[13]。在栽培过程中,鹿茸菇菌丝在栽培料中的快速定植是极为重要的一个环节,菌丝定植时间过长,容易出现高污染,这与程继红[21]所描述的情况极为相似。本研究利用单因素-响应面优化的培养基配方,在摇瓶培养时收获菌丝生物量可达32.81±1.10 g·L−1,该值是张凌珊[16]研究结果的40余倍,应用该配方制备的液体菌种与传统固体菌种相比,制种周期、污染率均大幅下降,单包产量提高15.74%,具有较大的生产应用潜能。
本研究结果最优配方为全麦粉47 g·L−1、花生饼粉22 g·L−1、KH2PO4 2.00 g·L−1、MgSO4·7H2O 2.00 g·L−1,当发酵培养至第8 d时,菌丝生物量达到最大值(32.81±1.10g·L−1),该配方可获得高质量的生产用种。尽管如此,该液体菌种配方优化尚处于发酵优化的小试阶段,后续关于接种量、发酵罐培养方式等技术参数有待进一步优化。此外,本研究结果表明以液体发酵方式制备鹿茸菇生产用种适合鹿茸菇工厂化生产。
-
图 1 草莓FaMC家族氨基酸序列比对分析
注:矩形:LSD1型锌指结构域;大粗实线:脯氨酸和谷氨酰胺的N端前结构域;中粗实线:p10亚基;细实线:p20亚基;虚线:连接区
Figure 1. Multiple sequence alignments of FaMC proteins in strawberry
Note: Rectangle: LSD1-type zinc finger motif; thickest solid line: Pro/Gln-rich NTD; thicker solid line: p10 subunit; solid line: p20 subunit; dotted line: linker.
图 2 草莓与拟南芥MC家族蛋白质系统进化分析
注:拟南芥MC蛋白登录号:AtMC1(NP_171719.2);AtMC2(NP_001031711.1);AtMC3(NP_201229.1);AtMC4(NP_178052.1);AtMC5(NP_178051.1);AtMC6(NP_178050.1);AtMC7(NP_178049.2);AtMC8(NP_173092.1);AtMC9(NP_196040.1)
Figure 2. Phylogenetic tree of MC gene family in strawberry and Arabidopsis
Note: Accessions of AtMC proteins: AtMC1 (NP_171719.2); AtMC2 (NP_001031711.1); AtMC3 (NP_201229.1); AtMC4 (NP_178052.1); AtMC5 (NP_178051.1); AtMC6 (NP_178050.1); AtMC7 (NP_178049.2); AtMC8 (NP_173092.1); AtMC9 (NP_196040.1).
图 4 草莓FaMC基因的表达谱
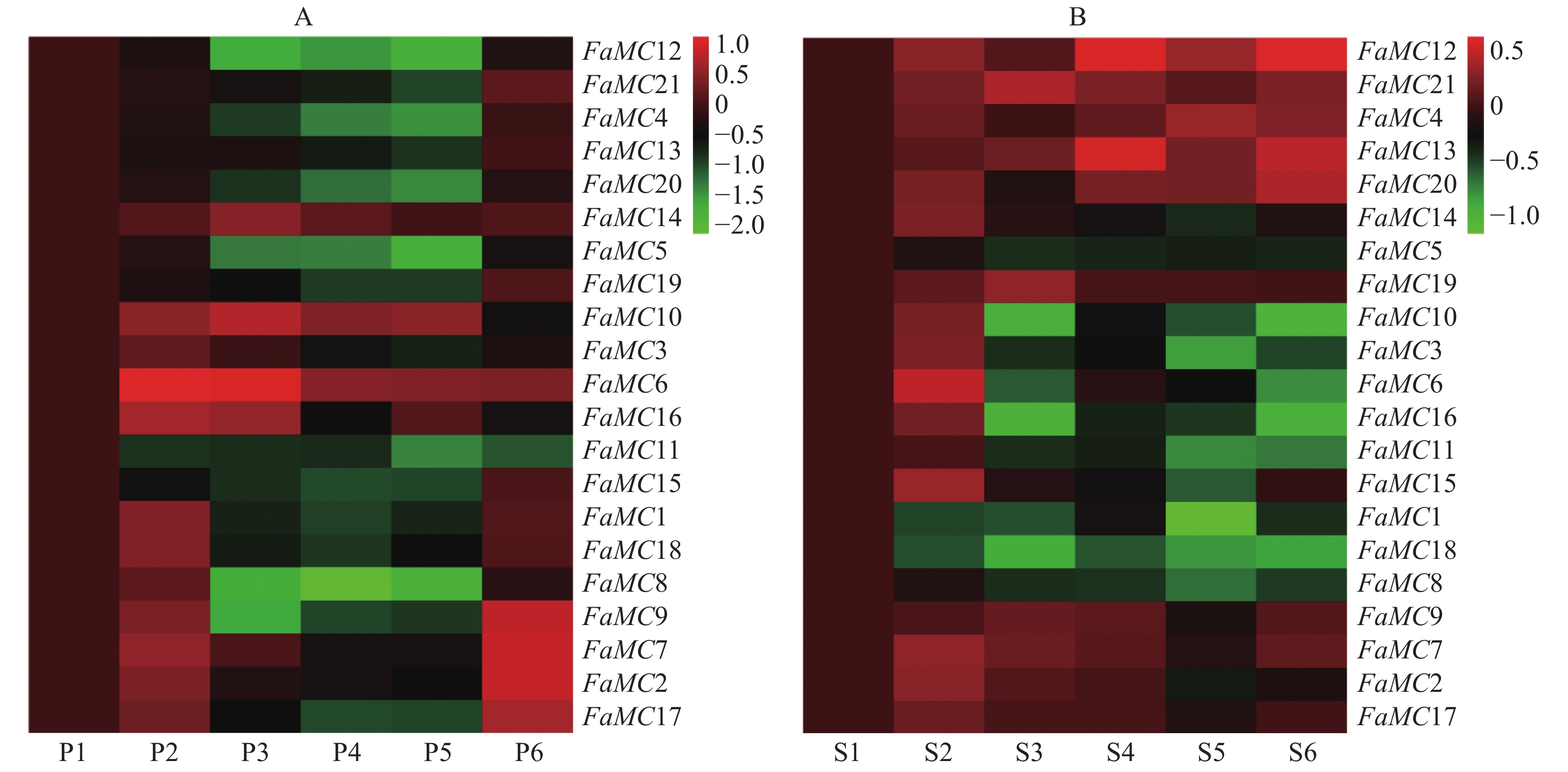
注:A:FaMC基因在不同组织部位的表达谱;P1:叶片;P2:花;P3:根;P4:茎;P5:匍匐茎;P6:幼果。B:FaMC基因在果实不同发育时期的表达谱;S1:小绿期;S2:大绿期;S3:白果期;S4:始红期;S5:片红期;S6:全红期。红色代表高表达,绿色代表低表达。
Figure 4. Expression patterns of FaMC genes in strawberry
Note: a: Expression patterns of FaMC in different tissues; P1: leaf; P2: flower; P3: root; P4: stem; P5: stolon; P6: young fruit. b: expression patterns of FaMC in different fruit developmental stages; S1: small and green; S2: large and green; S3: white; S4: turning red; S5: partial red; S6: fully red. Red color represents high expression, green low expression.
表 1 引物序列
Table 1 Primer sequences
基因
Gene上游引物(5′→3′)
Forward primer下游引物(5′→3′)
Reverse primerFaActin TGGGTTTGCTGGAGATGAT CAGTTAGGAGAACTGGGTGC FaMC1 ATGTTGGAGCACCTCACCTC GCTTGACACCCACTCAGAAG FaMC2 CAAAAACATCCGAAGGGCTA CGGCAACCTTTCTTTAGCTG FaMC3 CTCTCATCTTCCTCCTACAACG GCATCGAGCTCCATATTCTTCC FaMC4 TCTGGCAGTGCTGTTACGTC CGCAGGCAGTTAATTGAGGC FaMC5 CTTCCGTTCTCATGCTCACA CAAAATCCGAGGGACACAGT FaMC6 CGGTGTCAAGCTGCATTCTA CCACCTTTTGTTCCTTTCCA FaMC7 TCCCACTGACATGAACCTCA GCTAGACTCACGCTGGTTCC FaMC8 CGAGAAAGAGCAGATTGGACC GCAAGTTGAGATCGAGTAGAGG FaMC9 TGACCAGTATGTAAAGCCCG ATCTCCTGAAGGTGTGGCAT FaMC10 AATACGGAGCTCGATGGCTG CGTTATGTTTGTCTTGGTAGGG FaMC11 GAAATCCCTGAAATGGCTTG GTGGGTGAACGATGGTTGA FaMC12 ATTATCGTGGGGATGAAGTCG CCATACATATTTTCCACCCCTG FaMC13 GCCGCAATTGTTATGCCCAT CACCCCTGTCCATTCTGCAA FaMC14 CTTCACGATCTGCAACTCCA GGCAGTTGGAGCAGTCTACC FaMC15 CGAGTGGTCAGACAGCATCA ACCAGCCGGAACTTTCTCGT FaMC16 GCCCTTTCAATGCAGACCT ACACCAACGAATCTCCAGCTT FaMC17 ACAAACTCCAGAGCCACCAG TGACAGCCACTGATGAGAATG FaMC18 CGAGTTGTTGAAGCCGGA TCTATTGCTTAGCGGAGCC FaMC19 TACGGGAACATGATGACTGC AGCTTTCCAAGACTCCCTCC FaMC20 GCAGCAATTGTTAGTCCCAT CACTTGTTCCTTTCCATACGC FaMC21 GAAATACGGGTTCTGGTGGA CTCGCAGGCAGTTAATTGGG 表 2 草莓FaMC蛋白理化性质
Table 2 Physiochemical properties and subcellular localization of FaMC proteins in strawberry
基因
Gene基因组编号
Genome ID氨基酸数
Amino
acid分子量
Molecular
weight等电点
pI不稳定指数
Instability
index亲水性
Hydropathicity亚细胞定位
Subcellular
localizationFaMC1 augustus_masked-Fvb3-1-processed-gene-103.9 324 35555.30 5.43 37.04 −0.219 叶绿体 Chloroplast FaMC2 maker-Fvb3-1-snap-gene-242.61 402 44269.43 5.06 42.48 −0.582 细胞质 Cytoplasmic FaMC3 augustus_masked-Fvb3-1-processed-gene-281.5 326 36519.19 7.58 45.73 −0.110 叶绿体 Chloroplast FaMC4 maker-Fvb7-2-augustus-gene-79.38 381 40904.39 6.42 41.60 −0.302 叶绿体 Chloroplast FaMC5 maker-Fvb7-2-augustus-gene-253.66 358 38720.78 6.85 50.79 −0.327 细胞核 Nuclear FaMC6 snap_masked-Fvb3-3-processed-gene-22.24 347 38805.38 8.14 47.98 −0.354 叶绿体 Chloroplast FaMC7 maker-Fvb3-3-augustus-gene-63.33 413 45221.46 5.12 40.94 −0.616 细胞质 Cytoplasmic FaMC8 maker-Fvb3-4-augustus-gene-109.35 323 35386.06 5.19 37.43 −0.207 叶绿体 Chloroplast FaMC9 maker-Fvb3-4-augustus-gene-226.44 413 45224.44 5.06 40.97 −0.579 细胞质 Cytoplasmic FaMC10 augustus_masked-Fvb3-4-processed-gene-255.9 290 31963.38 6.09 39.67 −0.362 叶绿体 Chloroplast FaMC11 maker-Fvb5-1-augustus-gene-207.38 406 45451.82 8.95 52.83 −0.491 线粒体 Mitochondrial FaMC12 maker-Fvb7-1-augustus-gene-86.37 381 40803.28 6.37 44.55 −0.282 叶绿体 Chloroplast FaMC13 maker-Fvb7-1-augustus-gene-129.25 381 40807.37 6.48 45.56 −0.272 叶绿体 Chloroplast FaMC14 maker-Fvb7-1-snap-gene-270.81 338 37729.50 9.10 62.14 −0.194 叶绿体 Chloroplast FaMC15 maker-Fvb5-4-snap-gene-163.56 413 46277.63 8.88 54.06 −0.532 线粒体 Mitochondrial FaMC16 snap_masked-Fvb3-2-processed-gene-39.23 350 39121.78 7.47 49.27 −0.334 叶绿体 Chloroplast FaMC17 maker-Fvb3-2-augustus-gene-75.24 413 45214.42 5.06 41.30 −0.596 细胞质 Cytoplasmic FaMC18 augustus_masked-Fvb3-2-processed-gene-201.8 323 35426.17 5.27 36.96 −0.203 线粒体 Mitochondrial FaMC19 maker-Fvb7-3-augustus-gene-48.64 350 38180.67 7.59 39.34 −0.154 叶绿体 Chloroplast FaMC20 maker-Fvb7-3-augustus-gene-157.36 371 39964.41 6.93 39.93 −0.267 线粒体 Mitochondrial FaMC21 maker-Fvb7-4-augustus-gene-151.37 371 39772.15 6.34 40.74 −0.256 叶绿体 Chloroplast 表 3 草莓FaMC蛋白氨基酸保守序列
Table 3 Conserved motifs of FaMC proteins in strawberry
Motif 长度/aa
Length氨基酸保守序列
Amino acid conserved sequencemotif1 50 PTKKNIRMALHWLVQGCQAGDSLVFHYSGHGTRQPNYTGDEVDGYDETLC motif2 34 ELKGCINDAKCMKYLlinkerFKFPESSIRMLTEEE motif3 50 PLDFETQGMIVDDEINAAIVRPJPAGVKLHAIVDACHSGTVLDLPFLCRM motif4 29 PRSGVWKGTSGGEVISISGCDDBQTSADT motif5 41 ITSTGAMTFAFIQAIERGHAATYGNJLNAMRSTIRNTGSGA motif6 29 NHAPPPPPPNVHGRKRAVICGISYKYSRH motif7 21 GLRQEPQLTASEEFDVYTKPF motif8 50 AIQTILAETDGEITNQELVLRARKILKEQGYTQRPGLYCSDHHTDAPFVC motif9 11 MLVGCSNCRTP motif10 50 PTDMNLITDDDFRZFVDQLKKGCRLTIVSDSCHSGGLIDESVEQIGESHK motif11 50 KTDIDVGKLRPTLFDVFGDDASPKVKKFMKVILNKLQSHEGEGSGGLMGK motif12 50 IKNFLKQSAGDALKSRGIHVPSAFRRHGGDEEESEDREIDMGDGERGYMK motif13 20 QLPPGAZSIRCALCQAVTLI motif14 29 SAVASLLGGSSGAVTSLVGMLLTGGSVGG motif15 11 RGGRYVWEDHR motif16 50 DTLSGSCNKTKAIPFESILEHLTSLTGISTSDIATHFLELFAADASLKFR motif17 21 GDPRALHSAPSSHEPPPPAPY motif18 15 TPPGPPSYAYGALSY motif19 11 GHPPPPPPRPY motif20 15 NRELPLLALIFLLEQ -
[1] PATRICK E, THOMAS P, ROBERT V, et al. Origin and evolution of the octoploid strawberry [J]. Nature genetics, 2019, 51(3): 541−547. DOI: 10.1038/s41588-019-0356-4
[2] 苏代发, 童江云, 杨俊誉, 等. 中国草莓属植物种质资源的研究、开发与利用进展 [J]. 云南大学学报(自然科学版), 2018(6):1261−1276. DOI: 10.7540/j.ynu.20180613 SU D F, TONG J Y, YANG J Y, et al. Advances in research, exploitation and utilization of Fragaria spp. germplasm resources [J]. Journal of Yunnan University(Natural Sciences Edition), 2018(6): 1261−1276.(in Chinese) DOI: 10.7540/j.ynu.20180613
[3] 雷家军, 代汉萍, 谭昌华, 等. 中国草莓属植物的分类研究 [J]. 园艺学报, 2006, 33(1):1−5. DOI: 10.3321/j.issn:0513-353X.2006.01.001 LEI J J, DAI H P, TAN C H, et al. Studies on the taxonomy of the strawberry (Fragaria) species distributed [J]. Acta Horticulturae Sinica, 2006, 33(1): 1−5.(in Chinese) DOI: 10.3321/j.issn:0513-353X.2006.01.001
[4] LUCAS W, GROOVER A, LICHTENBERGER R, et al. The plant vascular system: evolution, development and functions [J]. Journal of Integrative Plant Biology, 2013, 55(4): 294−388. DOI: 10.1111/jipb.12041
[5] AHARONI A, KEIZER L, BROECK H, et al. Novel insight into vascular, stress, and auxin-dependent and -independent gene expression programs in strawberry, a non-climacteric fruit [J]. Plant Physiology, 2002, 129(3): 1019−1031. DOI: 10.1104/pp.003558
[6] FAIT A, HANHINEVA K, BELEGGIA R, et al. Reconfiguration of the achene and receptacle metabolic networks during strawberry fruit development [J]. Plant Physiology, 2008, 148(2): 730−750. DOI: 10.1104/pp.108.120691
[7] CAI J T, ZHANG Z H, ZHOU A Q, et al. Localization of BEN1-LIKE protein and nuclear degradation during development of metaphloem sieve elements in Triticum aestivum L [J]. Acta Biologica Hungarica, 2015, 66(1): 66−79. DOI: 10.1556/ABiol.66.2015.1.6
[8] LUIS C, ARMANDO B, JULIO M, et al. AtMCP1b, a chloroplast-localised metacaspase, is induced in vascular tissue after wounding or pathogen infection [J]. Functional Plant Biology, 2008, 34(12): 1061−1071. DOI: 10.1071/FP07153
[9] DANEVA A, GAO Z, VAN M, et al. Functions and regulation of programmed cell death in plant development [J]. Annual Review of Celland Developmental Biology, 2016, 32: 441−468. DOI: 10.1146/annurev-cellbio-111315-124915
[10] 冉昆, 马怀宇, 杨洪强. 植物细胞程序性死亡中的类胱天蛋白酶研究进展 [J]. 西北植物学报, 2008, 28(12):2564−2570. DOI: 10.3321/j.issn:1000-4025.2008.12.033 RAN K, MA H Y, YANG H Q. Recent advance in the study of caspase-like proteases involved in plant programmed cell death [J]. Acta Botanica Boreali-occidentalia Sinica, 2008, 28(12): 2564−2570.(in Chinese) DOI: 10.3321/j.issn:1000-4025.2008.12.033
[11] SANMARTIN M, JAROSZEWSKI L, RAIKHEL N, et al. Caspases. Regulating death since origin of life [J]. Plant Physiology, 2005, 137(3): 841−847. DOI: 10.1104/pp.104.058552
[12] LAM E, ZHANG Y. Regulating the reapers: activating metacaspases for programmed cell death [J]. Trends in Plant Science, 2012, 17(8): 487−494. DOI: 10.1016/j.tplants.2012.05.003
[13] TSIATSIAN L, BREUSEGEM F, GALLOIS P, et al. Metacaspases [J]. Cell Death and Differentiation, 2011, 18(8): 1279−1288. DOI: 10.1038/cdd.2011.66
[14] FAGUNDES D, BOHN B, CABREIRA C, et al. Caspases in plants: Metacaspase gene family in plant stress responses [J]. Functional & Integrative Genomics, 2015, 15(6): 639−649. DOI: 10.1007/s10142-015-0459-7
[15] MININA EA, STAEL S, VAN F, et al. Plant metacaspase activation and activity [J]. Methods in Molecular Biology, 2014, 1133: 237−253. DOI: 10.1007/978-1-4939-0357-3_15
[16] KLEMENCIC M, FUNK C. Evolution and structural diversity of MCs. [J]. Journal of Experimental Botany, 2019, 70(7): 2039−2047. DOI: 10.1093/jxb/erz082
[17] ALEXIS A, EDGAR S, LAURA S, et al. Two aspartate residues at the putative p10 subunit of a type II metacaspase from Nicotiana tabacum L. may contribute to the substrate-binding pocket [J]. Planta, 2014, 239(1): 147−160. DOI: 10.1007/s00425-013-1975-0
[18] DIETRICH R A, RICHBERG M H, SCHMIDT R, et al. A novel zinc finger protein is encoded by the Arabidopsis LSD1 gene and functions as a negative regulator of plant cell death [J]. Cell, 1997, 88(5): 685−694. DOI: 10.1016/s0092-8674(00)81911-x
[19] KWON S, HWANG D. Expression analysis of the metacaspase gene familly in Arabidopsis [J]. Journal of Plant Biology, 2013, 56: 391−398. DOI: 10.1007/s12374-013-0290-4
[20] WANG L, ZHANG H. Genomewide survey and characterization of metacaspase gene family in rice(Oryza sativa) [J]. Joural of Genetics, 2014, 93(1): 93−102. DOI: 10.1007/s12041-014-0343-6
[21] ZHANG C H, GONG P J, WEI R, et al. The metacaspase gene family of Vitis Vinifera L. : characterization and differential expression during ovule abortion in stenospermocarpic seedless grapes [J]. Gene, 2013, 528(2): 267−276. DOI: 10.1016/j.gene.2013.06.062
[22] AHMAD R, ZUILY Y, PASSAQUET C, et al. Ozone and aging up-regulate type II metacaspase gene expression and global metacaspase activity in the leaves of field-grown maize (Zea mays L.) plants [J]. Chemosphere, 2012, 87(7): 789−795. DOI: 10.1016/j.chemosphere
[23] CAO Y P, MENG D D, CHEN T, et al. Metacaspase gene family in Rosaceae genomes: comparative genomic analysis and their expression during pear pollen tube and fruit development [J]. PLos One, 2019, 14(2): 1−18. DOI: 10.1371/journal.pone.0211635
[24] BOLLHONER B, ZHANG B, STAEL S, et al. Post mortem function of AtMC9 in xylem vessel elements [J]. New Phytologist, 2013, 200(2): 498−510. DOI: 10.1111/nph.12387
[25] HE R, DRURY G, ROTARI V, et al. Metacaspase-8 modulates programmed cell death induced by ultraviolet light and H2O2 in Arabidopsis [J]. The Journal of Biological Chemistry, 2018, 283(2): 774−783. DOI: 10.1074/jbc.M704185200
[26] MARIA F, LADA H, ANDREI S, et al. Metacaspase-dependent programmed cell death is essential for plant embroyo genesis [J]. Current Biology, 2004, 14(9): 339−340. DOI: 10.1016/j.cub.2004.04.019
[27] ZHOU Y, HU L F, JIANG L, et al. Genome-wide identification, characterization, and transcriptional analysis of the metacaspase gene family in cucumber (Cucumis sativus) [J]. Genome, 2018, 61(3): 187−194. DOI: 10.1139/gen-2017-0174
[28] BOSTANCIOGLU S M, TOMBULOGLU G, TOMBULOGLU H. Genome-wide identification of barley MCs (metacaspase) and their possible roles in boron-induced programmed cell death [J]. Molecular Biology Reports, 2018, 45(3): 211−225. DOI: 10.1007/s11033-018-4154-3
[29] BOLLHONER B, LUKKARI S, BYGDELL J, et al. The function of two type II metacaspases in woody tissues of Populus trees [J]. New Phytologist, 2018, 217(4): 1551−1565. DOI: 10.1111/nph.14945
[30] 张智慧. 小麦颖果筛分子发育中II型metacaspase蛋白(TaeMCA II)的定位及蛋白质组学分析[D]. 武汉: 华中农业大学, 2015. ZHANG Z H. Localizrtion of the type II metacaspase protein (TaeMCA II) and proteomic analysis in sieve elements development of Triticum Aestivuml[D]. Wuhan: Huazhong Agricultural University, 2015. (in Chinese)
-
期刊类型引用(5)
1. 周静,张馨,赵兴秀,何义国,陈雨,张靖. 长裙竹荪液体培养基优化. 食用菌. 2024(03): 9-13+16 .  百度学术
百度学术
2. 金文松,李建华,单灿灿,吴涛锐,程泳春,程利纹,李佳欢,孙淑静,胡开辉. 鹿茸菇菌包后熟指标与出菇产量间的相关性. 福建农林大学学报(自然科学版). 2024(04): 563-569 .  百度学术
百度学术
3. 彭汇涵,于海玲,王明元,杨苗,万雨欣. 鹿茸菇液体菌种培养过程中生理指标的变化规律. 华侨大学学报(自然科学版). 2024(05): 667-672 .  百度学术
百度学术
4. 龚娜,刘国丽,陈珣,马晓颖,肇莹,肖军. 一株野生肺形侧耳的鉴定及其液体发酵培养基的优化. 浙江农业学报. 2024(11): 2535-2545 .  百度学术
百度学术
5. 陈珣,肇莹,龚娜,刘国丽,马晓颖,张敏,肖军. 基于响应面法优化滑菇液体菌种发酵培养基配方. 南方农业学报. 2024(12): 3625-3635 .  百度学术
百度学术
其他类型引用(0)




 下载:
下载: