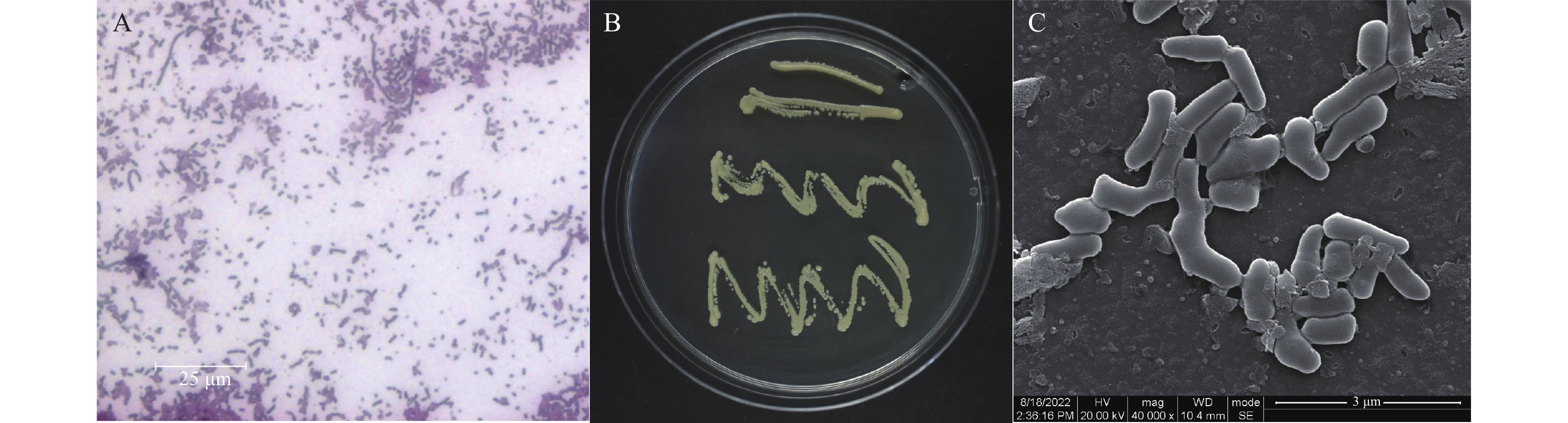
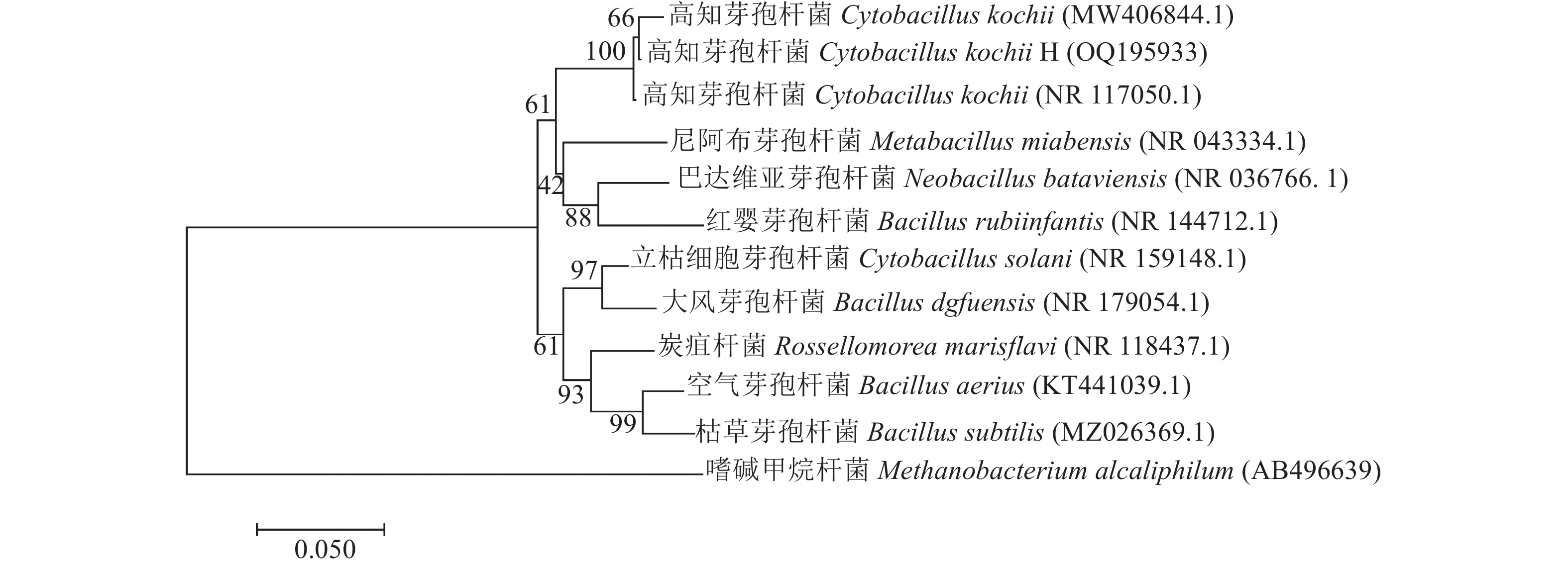
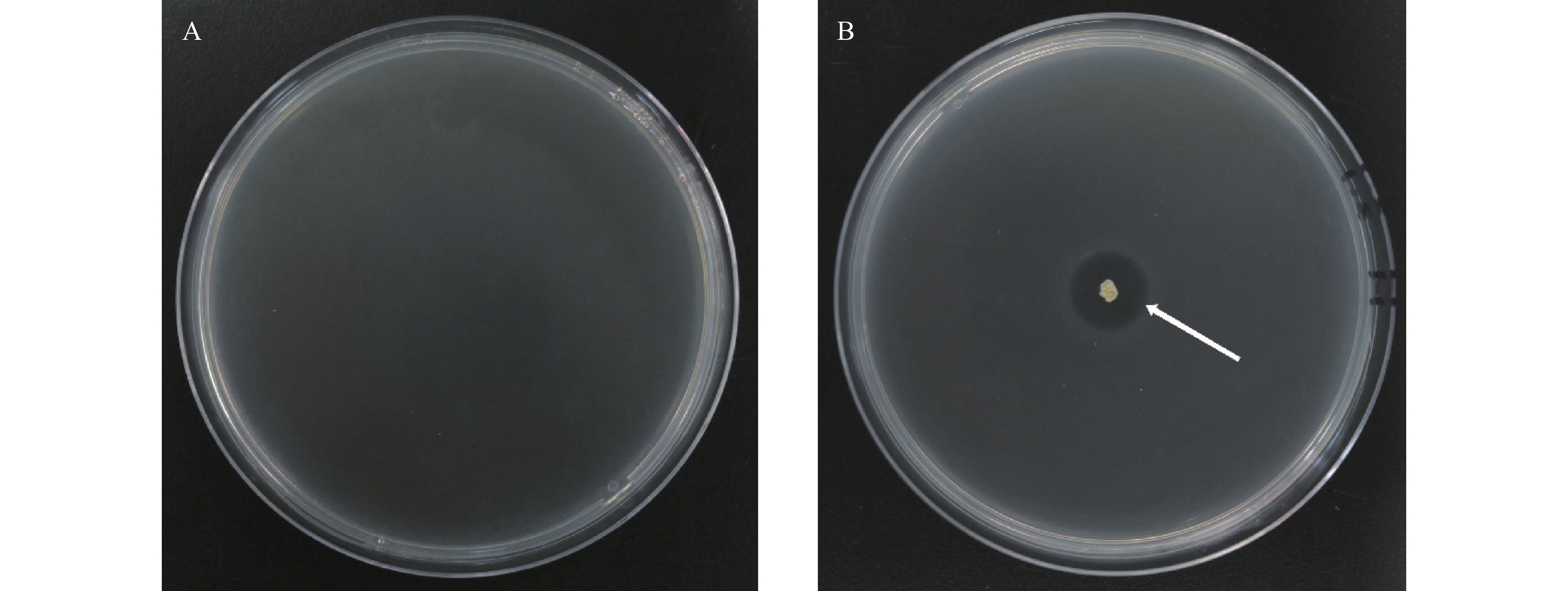
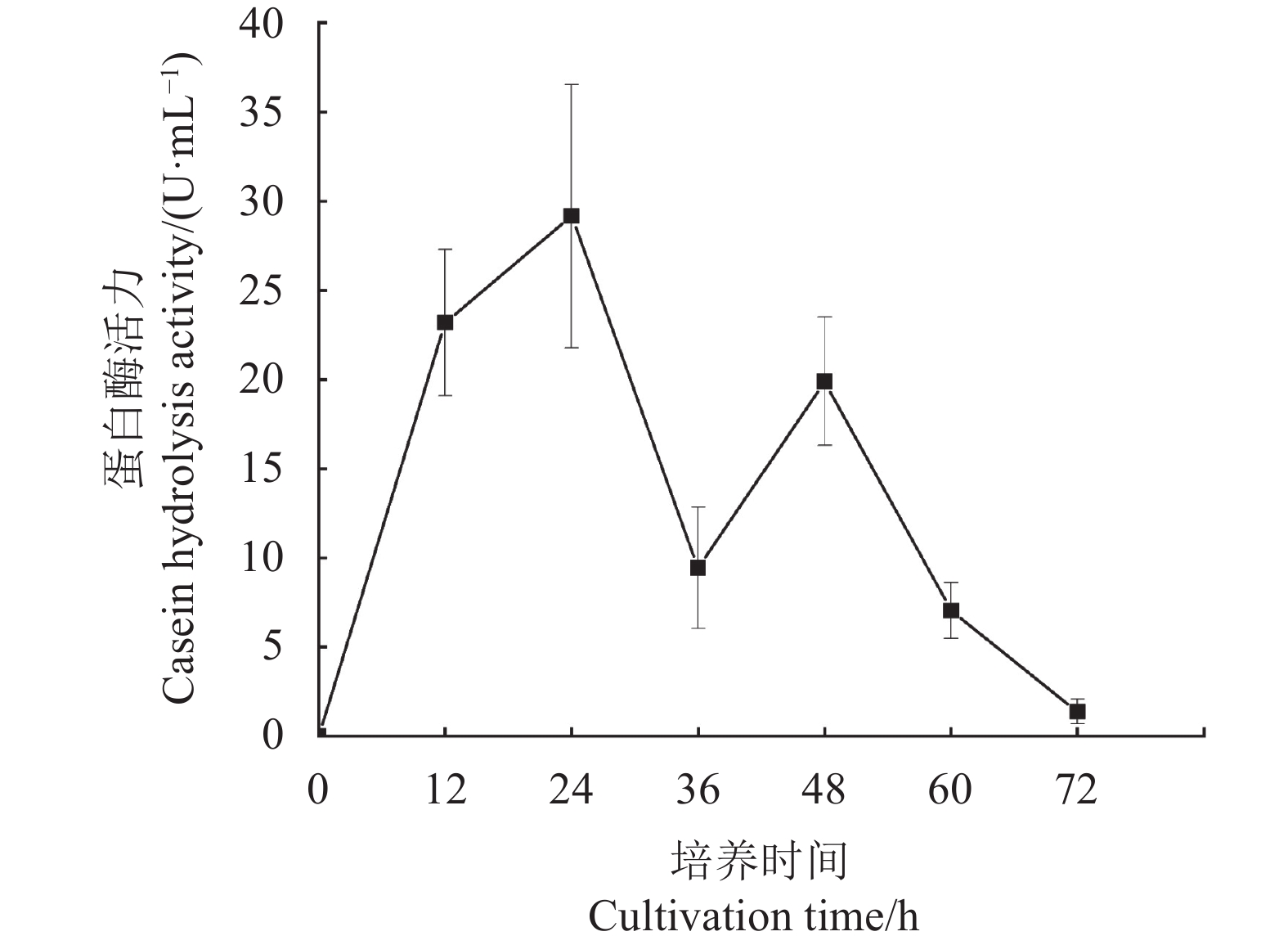
Process Optimization and Antimicrobial Effect of Cytobacillus kochii H Protease
-
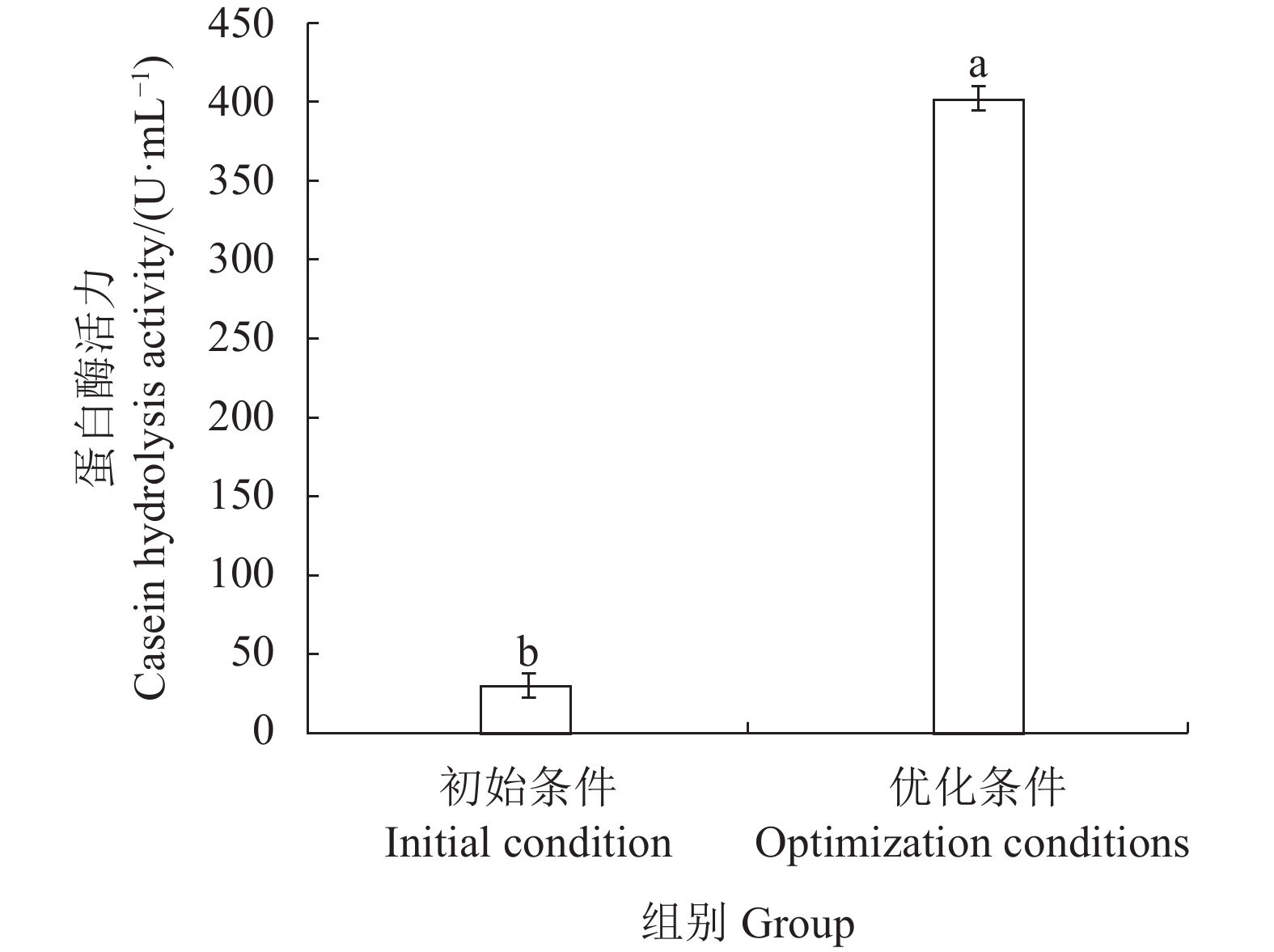
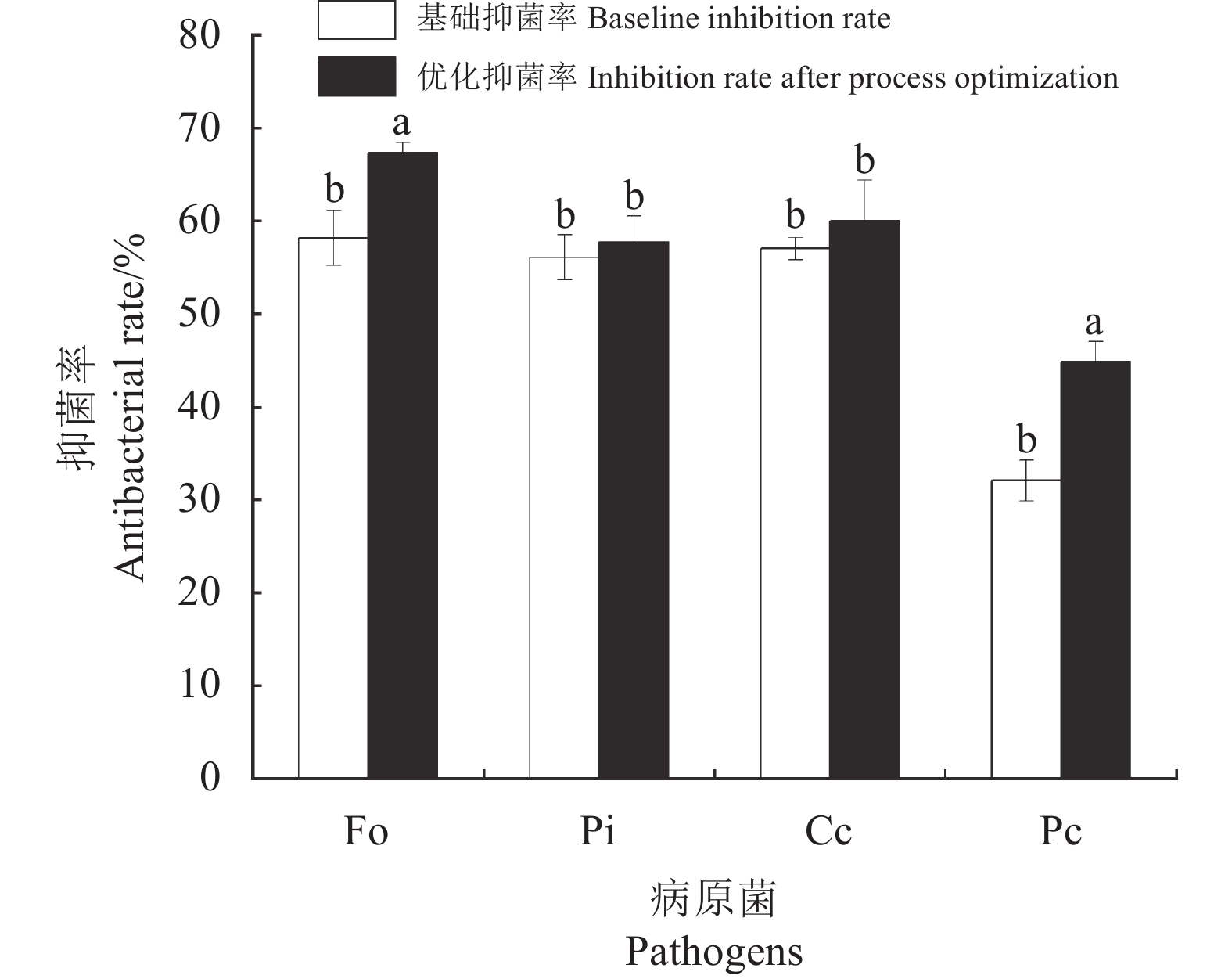
摘要:目的 对筛选到具有抑菌效果且产蛋白酶的菌株H进行分类地位的确定,同时对其产蛋白酶的条件进行优化,以期为其在植物病害防治上的应用奠定基础。方法 通过生理生化、电子显微镜扫描及16sRNA测序相结合的方法明确菌株H的分类地位;通过单因素和正交试验对菌株H产蛋白酶条件进行优化,并检测其优化前后对病原真菌的抑菌效果。结果 菌株H鉴定为高知芽孢杆菌(Cytobacillus kochii);菌株H产蛋白酶的优化条件为:pH8.0、葡萄糖20.0 g·L−1、蛋白胨8.0 g·L−1、MgSO4 1.0 g·L−1、CaSO4·2H2O 0.1 g·L−1,在装有50 mL发酵培养基的250 mL三角瓶中接种2.5 mL108 CFU·mL−1的种子液,培养24 h后蛋白酶活力达到402.2 U·mL−1,较初始培养条件下的蛋白酶活力提高13.92倍;优化后菌株H对尖孢镰刀菌(Fusarium oxysporum)和辣椒疫霉病菌(Phytophthora capsici)的抑菌率分别为67.32%和44.87%,较优化前抑菌率提升值分别为9.15%和12.82%。结论 明确菌株H为高知芽孢杆菌(C. kochii),优化了高知芽孢杆菌产蛋白酶条件及抑菌效果。Abstract:Objective Taxonomy of antimicrobial protease-producing Cytobacillus kochii H was studied for the development of a natural disease control agent on plants.Method Taxonomy of C. kochii H was determined by physiological, biochemical, electron microscopy scanning, and 16sRNA sequencing methods. A process of producing the antimicrobial protease secreted by the bacterium was optimized in single-factor and orthogonal experiments. The efficacy of the culture broth on 4 selected pathogenic fungi was applied in an in vitro test as the evaluation criterium.Result The optimized C. kochii H protease-producing process was conducted in a 250 mL triangular flask using 50 mL of pH 8.0 medium, which contained 20.0 g·L−1 of glucose, 8.0 g·L−1 of peptone, 1.0 g·L−1 of MgSO4, and 0.1g·L−1 of CaSO4-2H2O, inoculated with 2.5 mL of 108 CFU·mL−1 C. kochii H fermentation liquid. After 24 h of incubation, the protease activity reached 402.2 U·mL−1, which was 13.92 times greater than it was prior to the optimization. The antimicrobial rates of the broth against Fusarium oxysporum and Phytophthora capsici were 67.32% and 44.87%, respectively, i.e., 9.15% and 12.82%, respectively, increases over those before the optimization.Conclusion The antimicrobial protease-producing C. kochii H was identified taxonomically. The culture process of the enzyme production was optimized to deliver significant in vitro inhibition rates on F. oxysporum and P. capsici.
-
0. 引言
【研究意义】骨形态发生蛋白(Bone morphogenetic proteins,BMPs)家族除BMP1属于金属肽链内切酶家族外,其他均属于转化生长因子超家族。它们广泛存在,与两类特异受体结合组成信号传导通路诱导细胞内的一系列生理活动并参与生物体内多种功能,包括骨和喙的形成、羽毛发育、卵泡发育和癌症发生等[1-2]。检测骨形态发生蛋白在宿主细胞的表达水平能反应宿主的发育生长状态,在临床应用中有着重要的价值。【前人研究进展】研究发现,BMP家族蛋白除了均对骨骼发育起作用外,BMP1、4、6、8a和15还在哺乳动物卵巢发育及卵泡发生过程的阶段中起作用[3-7];BMP家族蛋白基因还对动物体的发育有不同程度的调控作用,BMP2对胚胎心脏和羊膜发育起重要作用[8],BMP5可在动物急性损伤中被激活,从而促进动物不同器官和组织的修复 [9],BMP7和BMP8a在肾的发育[10]以及精子形成[6]的过程中有重要作用;BMP3在成骨组织的形成,对骨组织中的其他BMP具有拮抗作用[11];BMP10主要表达于胚胎心脏发育,已证明可抑制多种癌症的发展[12-14]。目前,在人(Homo)[15]、大鼠(Rattus)[16]、麻羊(Capra hircus)[17]、军曹鱼(Rachycentron canadum)[18]、兔(Oryctolagus cuniculus)[19]等动物中均有构建部分BMP家族蛋白的SYBR Green Ⅰ 荧光定量RT-PCR的检测方法。基因的mRNA表达水平的检测方法包括实时荧光定量RT-PCR技术和转录组测序(RNA-seq),但目前最常用的方法是实时荧光定量RT-PCR,其优势在于可以更加简便、快速、准确地进行定量表达。【本研究切入点】而目前用SYBR Green Ⅰ 荧光定量RT-PCR方法测定鸭BMP家族蛋白mRNA表达水平还未见报道。【拟解决的关键问题】本研究旨在建立一种用于检测鸭BMPs蛋白mRNA表达水平的SYBR Green Ⅰ实时荧光定量RT-PCR方法,从而快速、精确检测鸭细胞和体内的BMPs蛋白mRNA转录水平,为BMPs蛋白的功能研究奠定基础。
1. 材料与方法
1.1 样本与细胞
无菌采集7日龄健康半番鸭心脏、肝脏、脾脏、肾脏、胸腺、法氏囊、上喙、肌肉组织,分别称量100 mg,加入1 mL Trizol后用高通量组织研磨仪研磨捣碎,提取RNA,−70 ℃保存备用;大肠杆菌DH5α感受态细胞购自北京全式金生物技术有限公司;鸭胚成纤维细胞传代细胞系(DEFs)购自美国ATCC细胞库。
1.2 主要试剂
SYBR Green Ⅰ qPCR MIX和RNA提取试剂盒及RNA纯化试剂盒购自北京全式金生物技术有限公司,RT-PCR反转录试剂盒、胶回收试剂盒和Phanta Max Super-Fidelity DNA Polymerase均购自南京诺唯赞生物科技有限公司,DNase购自天根生化科技(北京)有限公司,质粒小提试剂盒购自QIAGEN公司,pMD-18-T载体购自宝生物工程(大连)有限公司,2×Dream Taq Green PCR Master Mix购自赛默飞世尔科技(中国)有限公司。
1.3 引物设计与合成
根据GenBank登录的鸭BMP1、BMP2、BMP3、BMP4、BMP5、BMP6、BMP7、BMP8a、BMP10和BMP15基因序列,应用Oligo 6软件,分别设计一对特异性的引物(表1),均由生工生物工程(上海)股份有限公司合成。
表 1 BMP基因的扩增引物Table 1. BMP amplification primers引物名称
Primer name引物序列(5′-3′)
Primer sequence扩增长度
Amplification length/bp参考序列编号
GenBank No.BMP1-F CTTGAAGATGGAGCCGGAG 182 XM038166990 BMP1-R TGTCTCCTTTGCTCAGCCTC BMP2-F CCACGAAGAAGTTTTGGAAG
CTGTTGTTCTCAAAGGCTCC156 XM027453308 BMP2-R BMP3-F ATGAACTTCTGATCGGTGTC 170 XM038178529 BMP3-R GACCCTTGTCAGAGGATTAC BMP4-F CCACCATGAAGAGCACCTGG
TTTATCCGGTGGAAGCCCCT180 XM038179087 BMP4-R BMP5-F CCCAACAGGGATGCAGACTT
ACTTCGACCGTCCCCAGTTT165 XM038177391 BMP5-R BMP6-F CAAACAGATCCTCTGACCTG
TTTACACTGAAACCGTCCTG169 XM027451601 BMP6-R BMP7-F AAAGCATAGACGGGCAAAGC
TTTGGATCGATTCTGGCTCC167 XM005010474 BMP7-R BMP8a-F GCACCCTGCACATCAGCATC
AGCCCATCATCCGTCTCCAC202 XM038167176 BMP8a-R BMP10-F TCCTGGACTTGGAGAACCTG 181 XM038167140 BMP10-R GAATCCCAGCCAATCTCCTT BMP15-F ACGCCGTGGTGCAGAACTTGGT 157 XM027465364 BMP15-R CAGGACTCTGCGATCATGTTCT 1.4 cDNA样品的制备
用总RNA提取试剂盒按操作说明书分别提取鸭DEFs细胞的总RNA,提取的RNA经DNase消化后,用RNA纯化试剂盒纯化,溶于35 μL DEPC水,用于合成cDNA。按20 μL反应体系:总RNA 1 μg,5×Super Mix 4 μL,gDNA Remove 1 μL,加水补至20 μL,按说明书反应程序50 ℃ 5 min、85 ℃ 2 min,合成cDNA后用于PCR扩增及荧光定量PCR检测,剩余的cDNA放置于−20 ℃保存备用。
1.5 标准品的制备
用引物做常规PCR扩增组织样品基因,回收PCR产物,经试剂盒纯化后连接入pMD-18-T载体,转化至DH5α大肠杆菌,提取阳性质粒经PCR鉴定和通过序列测定分析验证后,测定DNA浓度,按公式计算标准品的拷贝数,以10倍系列稀释质粒标准品到下限至10−1 拷贝·μL−1。
1.6 标准曲线的建立
以10倍稀释好的标准品为模板,使用SYBR Green I荧光定量PCR法进行测定。反应体系:SYBR Green Ⅰ Real-time PCR Master Mix 10 μL,上游/下游引物各0.5 μL,模板1 μL,ddH2O 8 μL。
1.7 特异性和敏感性试验
应用1.4制备的cDNA样品,用常规PCR和荧光定量PCR验证所设计的BMP家族蛋白qPCR引物的特异性,对PCR产物进行核酸凝胶电泳及荧光定量PCR产物的熔解曲线进行分析,对扩增产物进行测序分析;将101~103 拷贝·μL−1的阳性标准质粒作为模板,其qPCR的Ct值变异系数均小于1%的样本最低浓度即为检出限。
1.8 重复性试验
分别用所建立的qPCR方法检测半番鸭脾脏组织样品BMP家族蛋白的mRNA表达水平,组间、组内各进行3次重复性试验,并计算变异系数。
1.9 组织样品的BMPs基因荧光定量PCR检测
无菌采集3只雏半番鸭心脏、肝脏、脾脏、肾脏、胸腺、法氏囊、喙和肌肉等组织,用总RNA提取试剂盒提取的组织RNA,用DNase去除基因组DNA后进行反转录合成cDNA,按照已确定的反应条件进行荧光定量PCR,计算各组织中BMPs基因的拷贝数,并进行统计分析。
2. 结果与分析
2.1 标准阳性质粒的制备
由DEFs细胞提取的总RNA经反转录成cDNA后,用表1中的特异性引物对BMP1、BMP2、BMP3、BMP4、BMP5、BMP6、BMP7、BMP8a、BMP10和BMP15基因进行PCR扩增,3%琼脂糖凝胶电泳得到与目的片段大小一致的产物,大小介于156~202 bp(图1)。产物回收后与pMD-18-T载体连接,并转化DH5α感受态细胞。经PCR及测序鉴定,与预期结果一致。重组质粒DNA的测序结果表明,插入片段与原始片段的序列同源性为100%,表明成功构建了质粒标准品。
2.2 荧光定量PCR检测
以10倍梯度稀释的质粒标准品为模板进行qPCR,得到BMP1、BMP2、BMP3、BMP4、BMP5、BMP6、BMP7、BMP8a、BMP10和BMP15的扩增曲线(图2)、标准曲线(图3)及熔解曲线(图4)。结果表明,标准品起始模板浓度与Ct值呈现良好的线性关系,相关系数均达到0.998以上,熔解曲线扩增产物形成单一的特异性熔解峰。
![]() 图 2 BMP基因的SYBR Green I荧光定量PCR扩增曲线A:1~6分别为6.03×108~6.03×103 copies·μL−1,7为DEPC水;B:1~6分别为6.41×108~6.41×103 copies·μL−1,7为DEPC水;C:1~6分别为1.73×108~1.73×103 copies·μL−1,7为DEPC水;D:1~6分别为6.22×107~6.22×102 copies·μL−1,7为DEPC水;E:1~6分别为1.84×108~1.84×103 copies·μL−1,7为DEPC水;F:1~6分别为1.85×107~1.85×102 copies·μL−1,7为DEPC水;G:1~6分别为1.49×107~1.49×102 copies·μL−1,7为DEPC水;H:1~6分别为7.01×107~7.01×102 copies·μL−1,7为DEPC水;I:1~6分别为6.03×108~6.03×103 copies·μL−1,7为DEPC水;J:1~6分别为2.86×107~2.86×102 copies·μL−1,7为DEPC水。Figure 2. Amplification curves of BMPs by SYBR Green I qRT-PCRA: 1–6 represent 6.03×108–6.03×103 copies·μL−1, respectively, 7 represents DEPC water; B: 1–6 represent 6.41×108–6.41×103 copies·μL−1, respectively, 7 represents DEPC water; C: 1–6 represent 1.73×108–1.73×103 copies·μL−1, respectively, 7 represents DEPC water; D: 1–6 represent 6.22×107–6.22×102 copies·μL−1, respectively, 7 represents DEPC water; E: 1–6 represent 1.84×108–1.84×103 copies·μL−1, respectively, 7 represents DEPC water; F: 1–6 represent 1.85×107–1.85×102 copies·μL−1, respectively, 7 represents DEPC water; G: 1–6 represent 1.49×107–1.49×102 copies·μL−1, respectively, 7 represents DEPC water; H: 1–6 represent 7.01×107–7.01×102 copies·μL−1, respectively, 7 represents DEPC water; I: 1–6 represent 6.03×108–6.03×103 copies·μL−1, respectively, 7 represents DEPC water; J: 1–6 represent 2.86×107–2.86×102 copies·μL−1, respectively, 7 represents DEPC water.
图 2 BMP基因的SYBR Green I荧光定量PCR扩增曲线A:1~6分别为6.03×108~6.03×103 copies·μL−1,7为DEPC水;B:1~6分别为6.41×108~6.41×103 copies·μL−1,7为DEPC水;C:1~6分别为1.73×108~1.73×103 copies·μL−1,7为DEPC水;D:1~6分别为6.22×107~6.22×102 copies·μL−1,7为DEPC水;E:1~6分别为1.84×108~1.84×103 copies·μL−1,7为DEPC水;F:1~6分别为1.85×107~1.85×102 copies·μL−1,7为DEPC水;G:1~6分别为1.49×107~1.49×102 copies·μL−1,7为DEPC水;H:1~6分别为7.01×107~7.01×102 copies·μL−1,7为DEPC水;I:1~6分别为6.03×108~6.03×103 copies·μL−1,7为DEPC水;J:1~6分别为2.86×107~2.86×102 copies·μL−1,7为DEPC水。Figure 2. Amplification curves of BMPs by SYBR Green I qRT-PCRA: 1–6 represent 6.03×108–6.03×103 copies·μL−1, respectively, 7 represents DEPC water; B: 1–6 represent 6.41×108–6.41×103 copies·μL−1, respectively, 7 represents DEPC water; C: 1–6 represent 1.73×108–1.73×103 copies·μL−1, respectively, 7 represents DEPC water; D: 1–6 represent 6.22×107–6.22×102 copies·μL−1, respectively, 7 represents DEPC water; E: 1–6 represent 1.84×108–1.84×103 copies·μL−1, respectively, 7 represents DEPC water; F: 1–6 represent 1.85×107–1.85×102 copies·μL−1, respectively, 7 represents DEPC water; G: 1–6 represent 1.49×107–1.49×102 copies·μL−1, respectively, 7 represents DEPC water; H: 1–6 represent 7.01×107–7.01×102 copies·μL−1, respectively, 7 represents DEPC water; I: 1–6 represent 6.03×108–6.03×103 copies·μL−1, respectively, 7 represents DEPC water; J: 1–6 represent 2.86×107–2.86×102 copies·μL−1, respectively, 7 represents DEPC water.2.3 特异性试验
应用所设计的10个BMP家族基因引物对鸭胚成纤维细胞样品进行常规PCR扩增,仅出现单条带;应用引物进行qPCR检测,熔解曲线为单峰,无杂峰(图4),且扩增测序结果与对应目的基因的同源性为100%,表明本研究设计的引物特异性好。
2.4 敏感性检测
取含量为101~103 拷贝·μL−1阳性标准重组质粒作为模板,每个样品组内重复3次,进行qPCR检测,计算各不同浓度样品的Ct值(
¯x±SD )。结果(表2)表明,BMP1、BMP2、BMP3、BMP4、BMP5、BMP6、BMP7、BMP8a、BMP10和BMP15的检测下限分别为6.03×102、6.41×102、1.73×102、6.22×102、1.84×102、1.85×102、1.49×102、7.01×102、1.34×102、2.86×102 拷贝·μL−1。表 2 实时荧光定量PCR敏感性检测Table 2. Sensitivity of qRT-PCR assay基因
Gene质粒浓度
Concentration of
plasmid standard /
(拷贝·μL−1)Ct值(¯x±SD)
Ct value (¯x±SD)变异系数
Coefficient of
variation/%BMP1 6.03×103 26.48±0.05 0.18 6.03×102 29.94±0.10 0.33 6.03×101 33.63±0.56 1.67 BMP2 6.41×103 28.93±0.03 0.11 6.41×102 32.36±0.23 0.71 6.41×101 33.97±0.62 1.83 BMP3 1.73×103 26.11±0.03 0.12 1.73×102 29.40±0.21 0.72 1.73×101 33.10±0.72 2.20 BMP4 6.22×103 29.07±0.10 0.34 6.22×102 32.09±0.10 0.34 6.22×101 33.94±0.55 1.64 BMP5 1.84×103 28.38±0.16 0.58 1.84×102 30.10±0.24 0.82 1.84×101 31.17±0.42 1.36 BMP6 1.85×103 28.45±0.02 0.08 1.85×102 31.92±0.09 0.27 1.85×101 36.35±0.46 1.28 BMP7 1.49×103 29.39±0.07 0.25 1.49×102 32.72±0.10 0.31 1.49×101 36.30±0.58 1.59 BMP8a 7.01×103 26.44±0.04 0.15 7.01×102 30.10±0.07 0.24 7.01×101 33.98±0.48 1.41 BMP10 1.34×104 24.53±0.07 0.31 1.34×102 27.43±0.18 0.68 1.34×101 30.44±0.53 1.74 BMP15 2.86×103 28.11±0.07 0.23 2.86×102 32.14±0.19 0.58 2.86×101 35.93±0.80 2.23 2.5 重复性试验
将采集的半番鸭脾脏用所建立的方法分别对BMP1、BMP2、BMP3、BMP4、BMP5、BMP6、BMP7、BMP8a、BMP10和BMP15进行重复性试验,计算变异系数,结果(表3)表示,组内变异系数为0.06%~0.28%,组间变异系数为0.32%~0.74%,变异系数均小于1%,表明重复性良好。
表 3 荧光定量PCR重复性试验结果Table 3. Reproducibility of qRT-PCR assay基因
Gene组内重复性试验Ct值
The Ct values of intra-assay组间重复性试验Ct值
The Ct values of inter-assay平均值±标准差
Means ± SD变异系数
CV/%平均值±标准差
Means ± SD变异系数
CV/%BMP1 20.19±0.02 0.12 20.04±0.08 0.42 BMP2 26.71±0.03 0.11 26.90±0.19 0.69 BMP3 28.88±0.08 0.28 28.79±0.12 0.41 BMP4 29.20±0.08 0.28 29.32±0.16 0.56 BMP5 31.06±0.06 0.20 31.37±0.23 0.72 BMP6 26.87±0.04 0.13 26.81±0.20 0.74 BMP7 23.40±0.03 0.12 23.52±0.11 0.47 BMP8a 32.14±0.07 0.22 32.11±0.19 0.60 BMP10 27.81±0.07 0.24 27.69±0.18 0.65 BMP15 25.13±0.01 0.06 25.17±0.08 0.32 2.6 组织样品BMPs表达水平的qRT-PCR检测
提取半番鸭中各个组织RNA,反转录后进行荧光定量PCR检测,计算BMPs基因的拷贝值。结果(图5)表明,所检测的基因在心脏、肝脏、脾脏、肾脏、胸腺、法氏囊、上喙、肌肉中均有表达。BMP1、BMP2、BMP5和BMP7基因在心脏中表达量较高,分别为5.5×104、1.1×105、5.1×105和7.7×105 拷贝·μL−1;BMP6和BMP10基因在肝脏中表达量较高,分别为7.5×104、7.6×103 拷贝·μL−1;而BMP3、BMP4、BMP8a和BMP15分别在法氏囊、胸腺、喙和脾脏中表达量较高,分别为1.5×105、2.1×104、1.8×103和3.3×103 拷贝·μL−1。
3. 讨论
目前为止,已确定的BMP家族成员约20个,但对于鸭BMP蛋白功能应用的研究较少。BMPs通过其分子的抗原决定簇与细胞表面的BMP Ⅰ型受体和Ⅱ型受体结合形成异源三聚体复合物,其中BMP配体与BMP Ⅱ型受体结合,使其氨基酸残基发生次磷酸化,激活Ⅱ型受体并磷酸化 Ⅰ 型受体的GS区(Glycine and serine-rich domain),激活 Ⅰ 型受体,并进一步将信号传递给细胞内的Smad蛋白。通过激活受体调节型Smad蛋白(Receptor regulate smad,R-smad)将信号传递至胞浆内,再和共同调节型Smad蛋白(Common mediator smad,Co-smad)结合后转移至细胞核,与靶基因结合诱导BMP蛋白合成[2, 20-21],参与诸如中胚层形成、神经系统分化、软骨、牙齿、骨骼发育以及癌症发生等许多重要的生物学过程。
通过本研究建立的qRT-PCR检测方法进行检测,发现鸭BMPs基因在已检测的8个组织中均有表达并呈现出一定的差异性。总体来看,BMP1~BMP7基因在各组织中拷贝量高于BMP8a、BMP10和BMP15基因;BMP1、BMP2、BMP5和BMP7基因在心脏中检测表达量较高,BMP6和BMP10基因在肝脏中的表达量较高,而BMP3、BMP4、BMP8a和BMP15基因分别在法氏囊、胸腺、喙和脾脏中的检出量最高。Somi等[20]发现通过抑制BMP2、4、5、6、7蛋白的合成,会导致体外培养的鸡心肌细胞的形成数量下降,证明BMP蛋白对鸡心肌形成至关重要;Huang等[22]发现BMP4基因具有调节人CD4+ T细胞中的糖酵解和干扰素γ的产生的机制; Wang等[23]确定BMP6是内皮细胞与肝细胞间串扰的主要调节因子,并进一步影响小鼠体内游离铁离子的调节代谢,BMPs基因还有可能参与调控鸭的繁殖生长、疾病产生和发展等方面,具体有待深入研究探讨。
SYBR Green Ⅰ实时荧光定量PCR技术相较于TaqMan实时RT-PCR的检测方法,成本低、无需设计探针;相较于常规PCR,减少了复杂的加样和电泳步骤,能够有效缩短样品的检测时间。本研究所设计的BMPs引物在常规PCR扩增出156~202 bp目的片段,均为单一条带,且无引物二聚体,荧光定量PCR所检测出的熔解曲线均为单峰,表明设计的引物特异性强;所绘制的标准曲线相关系数R2均大于0.998,组内变异系数为0.06%~0.28%,组间变异系数为0.32%~0.74%,均小于1%,表明该方法建立的标准曲线线性拟合度高,重复性良好,可以快速、精确、简便检测样品中BMPs的mRNA表达量。本研究建立的检测方法还可以通过同步检测BMPs和内参基因,得到BMPs mRNA的相对表达水平,为明确BMP家族蛋白在组织和细胞间的表达差异以及致病过程中BMP家族蛋白的表达机制研究奠定基础。
综上,本研究建立的鸭BMP家族蛋白mRNA转录水平的SYBR Green Ⅰ实时荧光定量RT-PCR检测方法特异性高、敏感性强、重复性好,可研究鸭不同组织和细胞中BMPs的差异,为检测鸭BMPs表达水平提供了技术手段。
-
图 7 高知芽孢杆菌(C. kochii) H产蛋白酶优化前后对4种病原菌的抑菌效果
A:尖孢镰刀菌(F. oxysporum);B:脐橙青霉病菌(P. italicu); C:辣椒疫霉病菌(P. capsici); D:辣椒炭疽病菌(C. capsici); A~D为对照; E~H为优化前; J~M为优化后。
Figure 7. Bacteriostatic effects of C. kochii H culture broth on 4 pathogens before and after process optimization
A: F. oxysporum; B: P. italic; C: P. capsica; D: C. capsica; Lines A-D: control; Lines E-H: before optimization; Lines J-M: after optimization.
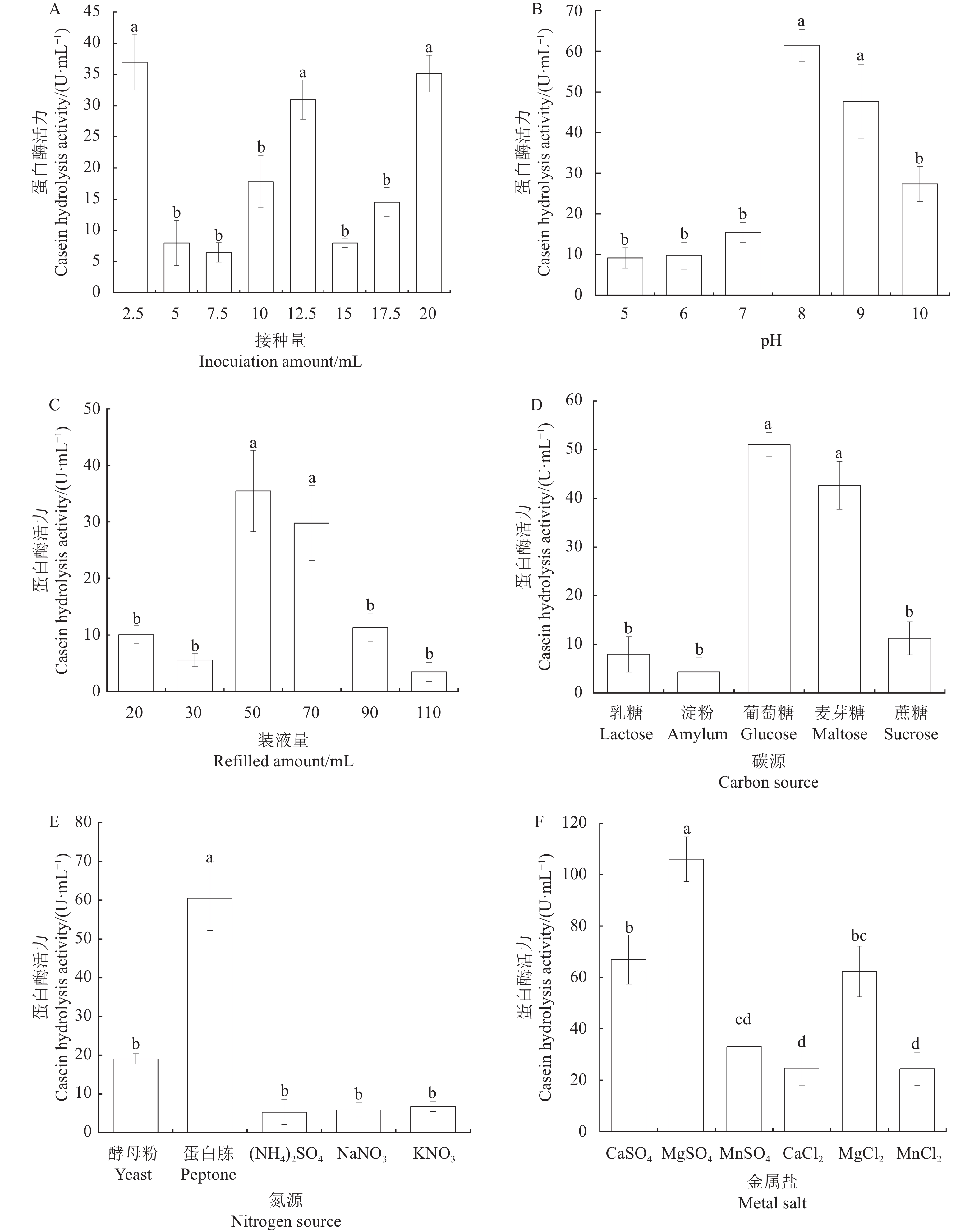
表 1 单因素优化设计
Table 1 Single factor optimization experimental design
接种量
Inocuiation amount/mL装液量
Refilled amount/mLpH 2.5 20 5.0 5.0 30 6.0 7.5 50 7.0 10 70 8.0 12.5 90 9.0 15 110 10.0 17.5 - - 20 - - 表 2 正交试验优化组合试验因子及水平
Table 2 Factors and levels of optimization experiment
水平
LevelA:葡萄糖
Glucose/
(g·L−1)B:蛋白胨
Peptone/
( g·L−1)C:Mg2+/
( g·L−1)D:Ca2+/
( g·L−1)1 10 8 0.1 0.1 2 15 10 0.5 0.5 3 20 12 1.0 1.0 表 3 菌株H生理生化试验
Table 3 Physiological and biochemical test results on C. kochii H
试验名称
Test names试验现象
Experimental phenomenon试验结果
Results甲基红试验 Methylred test 反应液呈黄色 The reaction solution is yellow − 革兰氏染色 Gram staining test 菌体呈紫色 The cell is purple + V-P试验 V-P test 培养液呈非红色 The culture medium is not red − 靛基质试验 Imdole test 反应液呈红色 The reaction solution is red + 触酶试验 Contact enzyme test 有气泡 Bubble + 柠檬酸盐利用试验 Citrate test 培养基呈深蓝色 The culture medium is navy blue + +:阳性;−:阴性。
+: positive; −: negative.表 4 高知芽孢杆菌(C. kochii) H产蛋白酶优化组合正交试验结果
Table 4 Orthogonal optimization of C. kochii H protease-producing process
试验编号
Test No因子 Factor 酶活力
Protease activity/(U·mL−1)葡萄糖
Glucose蛋白胨
PeptoneMg2+ Ca2+ 1 1 1 1 1 215.370 2 1 2 2 2 51.584 3 1 3 3 3 122.418 4 2 1 2 3 15.460 5 2 2 3 1 176.814 6 2 3 1 2 100.002 7 3 1 3 2 360.028 8 3 2 1 3 107.773 9 3 3 2 1 221.348 K1 129.791 196.953 141.048 204.511 - K2 97.426 112.057 96.130 170.538 - K3 229.716 147.923 219.753 81.884 - R 132.291 84.896 123.623 122.627 - 优化组合
Optimization grouping3 1 3 1 - 表 5 蛋白酶抑菌验证
Table 5 Validation on bacteriostasis of C. kochii H protease
病原菌
Pathogen粗酶液抑菌率
Antibacterial rate of
crude enzyme
solution/%粗酶液灭活后抑菌率
Antibacterial rate of
crude enzyme solution
after inactivation/%尖孢镰刀菌
Fusarium oxysporum21.89±1.40 a 6.26±1.23 b 辣椒疫霉病菌
Phytophthora capsici36.67±3.10 a 29.63±3.40 a 脐橙青霉病菌
Penicillium italicu16.90±2.70 a 1.98±1.31 b 辣椒炭疽病菌
Colletotrichum capsici17.57±9.69 a 0.40±0.00 b -
[1] KUDDUS M, PRAMOD W R. Recent development in production and biotechnological applications of cold-active mocrobial protease [J]. Critical Reviews in Microbiology, 2012, 38(4): 330−338. DOI: 10.3109/1040841X.2012.678477
[2] WANG Z, WANG Y, ZHENG L, et al. Isolation and characterization of an antifungal protein from Bacillus licheniformis HS10 [J]. Biochemical and Biophysical Research Communications, 2014, 454(1): 48−52. DOI: 10.1016/j.bbrc.2014.10.031
[3] 周桂旭, 文阳宣, 李新锋, 等. 重组枯草芽孢杆菌产碱性蛋白酶发酵条件优化 [J]. 山西大学学报(自然科学版), 2020, 43(2):405−412. DOI: 10.13451/j.sxu.ns.2019103 ZHOU G X, WEN Y X, LI X F, et al. Optimization of fermentation conditions for recombinant alkaline protease produced by Bacillus subtilis [J]. Journal of Shanxi University (Natural Science Edition), 2020, 43(2): 405−412.(in Chinese) DOI: 10.13451/j.sxu.ns.2019103
[4] 朱泓, 王一明, 林先贵. 一株高温蛋白酶高产菌株产酶条件的优化 [J]. 南京林业大学学报(自然科学版), 2014, 38(1):31−35. ZHU H, WANG Y M, LIN X G. Optimization of fermentation medium and culture condition of a thermostable protease from moderate thermophilic strain Bacillus subtilis BY25 [J]. Journal of Nanjing Forestry University (Natural Science Edition), 2014, 38(1): 31−35.(in Chinese)
[5] 龚志立, 曹誉, 刘平, 等. 产蛋白酶菌株P12产酶条件的优化 [J]. 科技创新与生产力, 2021(4):69−71. DOI: 10.3969/j.issn.1674-9146.2021.04.069 GONG Z L, CAO Y, LIU P, et al. Optimization of enzyme production condition of a protease strain P12 [J]. Sci-Tech Innovation and Productivity, 2021(4): 69−71.(in Chinese) DOI: 10.3969/j.issn.1674-9146.2021.04.069
[6] 韩淑梅, 李欣, 张芝元, 等. 微生物角蛋白酶的特性及其应用研究进展 [J]. 微生物学通报, 2021, 48(11):4315−4326. DOI: 10.13344/j.microbiol.china.210152 HAN S M, LI X, ZHANG Z Y, et al. Research progress on the characteristics and application of microbial keratinase [J]. Microbiology China, 2021, 48(11): 4315−4326.(in Chinese) DOI: 10.13344/j.microbiol.china.210152
[7] HAMED A A, KHEDR M, ABDELRAOF M. Molecular characterization of alkaline protease-coding gene from Bacillus licheniformis MK90 mutants with biofilm inhibitory activity [J]. Egyptian Pharmaceutical Journal, 2019, 18(4): 419−433. DOI: 10.4103/epj.epj_47_19
[8] KABANOV D, KHABIPOVA N, VALEEVA L, et al. Effect of subtilisin-like proteinase of Bacillus pumilus 3-19 on Pseudomonas aeruginosa biofilms [J]. Bionanoscience, 2019, 9(2): 515−520. DOI: 10.1007/s12668-019-00617-z
[9] 张永军, 彭国雄, 方卫国, 等. 球孢白僵菌胞外蛋白酶及类枯草杆菌蛋白酶的诱导 [J]. 应用与环境生物学报, 2000, 6(2):182−186. DOI: 10.3321/j.issn:1006-687X.2000.02.017 ZHANG Y J, PENG G X, FANG W G, et al. Induction of extracellular protease and subtilisin like protease of Beauveria bassiana [J]. Chinese Journal of Applied and Environmental Biology, 2000, 6(2): 182−186.(in Chinese) DOI: 10.3321/j.issn:1006-687X.2000.02.017
[10] FAN H J, LIU Z H, ZHANG R S, et al. Functional analysis of a subtilisin-like serine gene from biocontrol fungus Trichoderma harzianum [J]. Journal of Microbiology, 2014, 52(2): 129−138. DOI: 10.1007/s12275-014-3308-9
[11] 陈瑜, 孔海深. 伯杰鉴定细菌学手册第9版简介[J]. 国外医学(微生物学分册), 1995, 18(6): 32, 48. CHEN Y, KONG H S. Berger’s Handbook of identification bacteriology, 9th Edition[J] Foreign Med., 1995, 18 (6): 32, 48.
[12] 姜艳彬, 王海, 侯东军, 等. 两种快速细菌菌种鉴定方法的比较 [J]. 中国测试, 2010, 36(5):41−44. JIANG Y B, WANG H, HOU D J, et al. Comparison of two rapid bacteria strain identification methods [J]. China Measurement & Test, 2010, 36(5): 41−44.(in Chinese)
[13] 中华人民共和国国家质量监督检验检疫总局. 蛋白酶制剂: GB/T23527-2009[S]. 北京: 中国标准出版社, 2009. [14] 王永红, 李小斌, 徐磊, 等. 产蛋白酶菌株的筛选、鉴定及水解菜粕蛋白能力 [J]. 生物资源, 2018(2):135−140. WANG Y H, LI X B, XU L, et al. Screening and identification of protease producing strains and their ability of hydrolysis of rapeseed protein [J]. Biotic Resources, 2018(2): 135−140.(in Chinese)
[15] 曹慧, 张腾月, 赵龙妹, 等. 土壤中高产蛋白酶菌株产酶条件及酶学性质 [J]. 微生物学通报, 2020, 47(7):2072−2081. DOI: 10.13344/j.microbiol.china.200135 CAO H, ZHANG T Y, ZHAO L M, et al. Identification and characterization of a high protease-producing strain from soil [J]. Microbiology China, 2020, 47(7): 2072−2081.(in Chinese) DOI: 10.13344/j.microbiol.china.200135
[16] 张红岩, 张妮, 杨梦莹, 等. 拟蕈状芽孢杆菌Gxun-30产角蛋白酶液体发酵条件优化 [J]. 食品与发酵工业, 2021, 47(4):136−143. DOI: 10.13995/j.cnki.11-1802/ts.025196 ZHANG H Y, ZHANG N, YANG M Y, et al. Optimization of liquid fermentation conditions of keratinase produced by Bacillus paramycoides Gxun-30 [J]. Food and Fermentation Industries, 2021, 47(4): 136−143.(in Chinese) DOI: 10.13995/j.cnki.11-1802/ts.025196
[17] 杨城, 姚善泾, 杨志坚, 等. 一株产酸性蛋白酶菌株的筛选、鉴定及发酵条件优化 [J]. 农业生物技术学报, 2019, 27(2):371−380. YANG C, YAO S J, YANG Z J, et al. Screening, identification and fermentation optimization of a acidprotease strain [J]. Journal of Agricultural Biotechnology, 2019, 27(2): 371−380.(in Chinese)
[18] 朱祥杰, 王震, 苑志欣, 等. 海洋芽孢杆菌N11-8产蛋白酶的发酵条件优化 [J]. 渔业科学进展, 2018, 39(6):155−163. ZHU X J, WANG Z, YUAN Z X, et al. Optimization of fermentation conditions of Bacillus sp. N11-8 on the production of protease PBN11-8 [J]. Progress in Fishery Sciences, 2018, 39(6): 155−163.(in Chinese)
[19] 侯泽林. 从土壤中筛选碱性蛋白酶产生菌及产酶条件优化研究[D]. 哈尔滨: 东北农业大学, 2021. HOU Z L. Screening alkaline protease-producing bacteria from soil and optimization of enzyme-producing conditions[D]. Harbin: Northeast Agricultural University, 2021. (in Chinese)
[20] 周魏, 曾嵩玉, 余金凤, 等. 一株地衣芽胞杆菌产碱性蛋白酶条件优化 [J]. 微生物学通报, 2022, 49(7):2753−2766. DOI: 10.13344/j.microbiol.china.211107 ZHOU W, ZENG S Y, YU J F, et al. Optimization of alkaline protease production by a strain of Bacillus licheniformis [J]. Microbiology China, 2022, 49(7): 2753−2766.(in Chinese) DOI: 10.13344/j.microbiol.china.211107
[21] 刘新风, 牛春华, 刘香英, 等. 枯草芽孢杆菌BSG1产蛋白酶发酵条件优化 [J]. 食品工业, 2013, 34(7):1−4. LIU X F, NIU C H, LIU X Y, et al. The Bacillus subtilis BSG1 producing protease optimization of fermentation conditions [J]. The Food Industry, 2013, 34(7): 1−4.(in Chinese)
[22] 曹春红, 李爽, 王海燕, 等. 解淀粉芽孢杆菌YF03产蛋白酶发酵培养基及发酵条件的优化 [J]. 饲料工业, 2020(10):23−29. CAO C H, LI S, WANG H Y, et al. The Bacillus amyloliquefaciens YF03 producing protease optimization of fermentation medium and fermentation conditions [J]. Feed Industry, 2020(10): 23−29.(in Chinese)
[23] HADDAR A, HMIDET N. Alkaline proteases produced by Bacillus licheniforms PR1 grown on shrimp wastes: application in chitin extraction, chicken feather-degradation and as a dehairing agent [J]. Biotechnology and Bioprocess Engineering, 2011(16): 669−678.
[24] 张晓云, 李宝庆, 郭庆港, 等. 枯草芽孢杆菌CAB-1抑菌蛋白对黄瓜白粉病的防治作用 [J]. 中国生物防治学报, 2012, 28(3):375−380. DOI: 10.3969/j.issn.2095-039X.2012.03.012 ZHANG X Y, LI B Q, GUO Q G, et al. Inhibitive effect of antifungal protein produced by Bacillus subtilis CAB-I against sphaerothecafuliginea in cucumber [J]. Chinese Journal of Biological Control, 2012, 28(3): 375−380.(in Chinese) DOI: 10.3969/j.issn.2095-039X.2012.03.012
[25] 陈志杰, 谢江辉, 陈宇丰, 等. 一株植物病原拮抗细菌的分离筛选及拮抗物质 [J]. 生态学杂志, 2018, 37(5):1595−1604. CHEN Z J, XIE J H, CHEN Y F, et al. Isolation and screening of a plant pathogen-antagonistic bacterium and antagonistic substance [J]. Chinese Journal of Ecology, 2018, 37(5): 1595−1604.(in Chinese)
[26] 彭帅. 地衣芽孢杆菌W10枯草杆菌蛋白酶Sp1抗菌和诱导植物抗病性机理研究[D]. 扬州: 扬州大学, 2021. PENG S. Study on the mechanism of antimicrobial activity and plant disease resistance induced by Bacillus licheniformis W10 subtilisin Sp1[D]. Yangzhou: Yangzhou University, 2021. (in Chinese)
[27] 彭兵, 张树斌, 贾宇, 等. 枯草芽孢杆菌菌株A抗菌蛋白的分离纯化及抗真菌机理 [J]. 中国农业科学, 2011, 44(1):67−74. DOI: 10.3864/j.issn.0578-1752.2011.01.008 PENG B, ZHANG S B, JIA Y, et al. Purification and antifungal mechanism of a kind of antifungal protein from strain A of Bacillus subtilis [J]. Scientia Agricultura Sinica, 2011, 44(1): 67−74.(in Chinese) DOI: 10.3864/j.issn.0578-1752.2011.01.008




 下载:
下载: