Identification and VP1 Sequencing of Duck Hepatitis A Virus Type 3
-
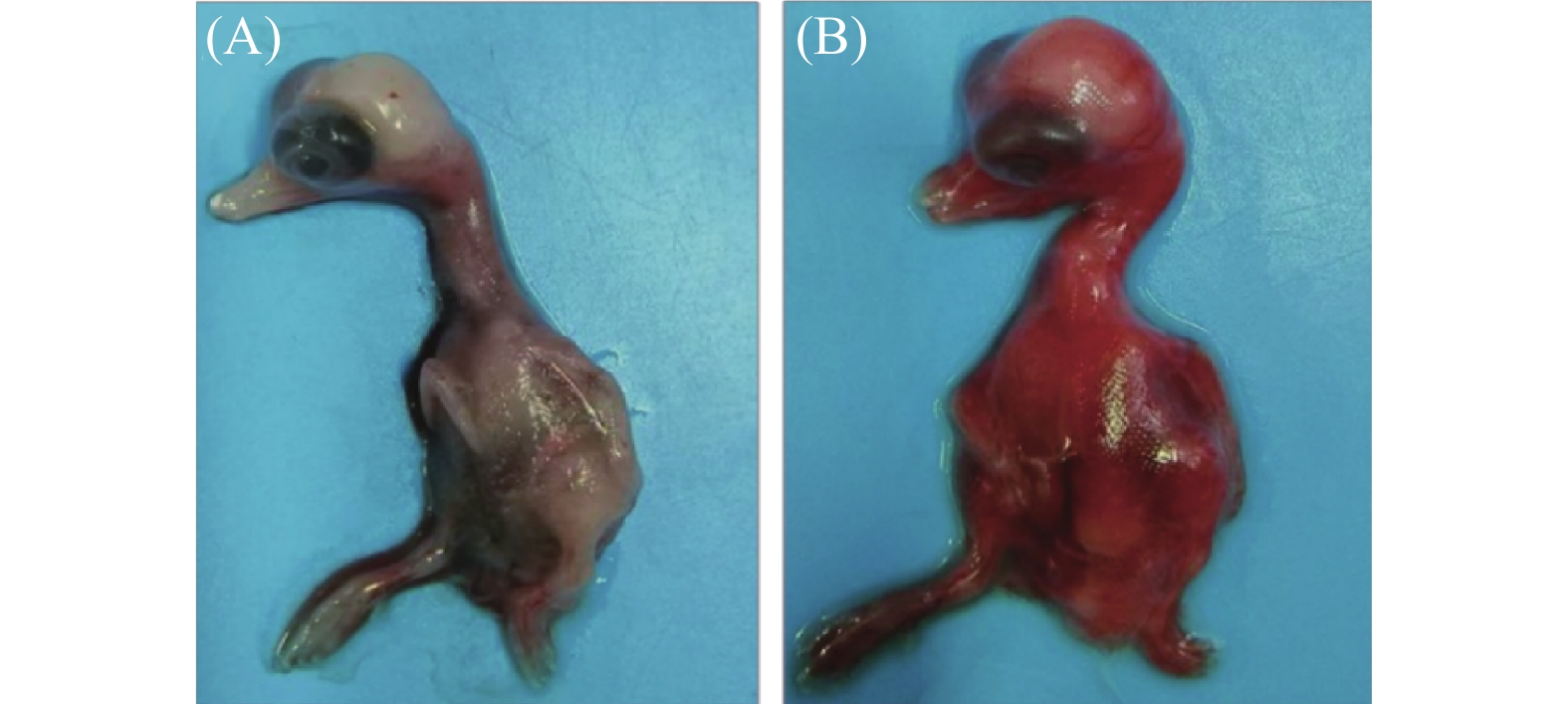
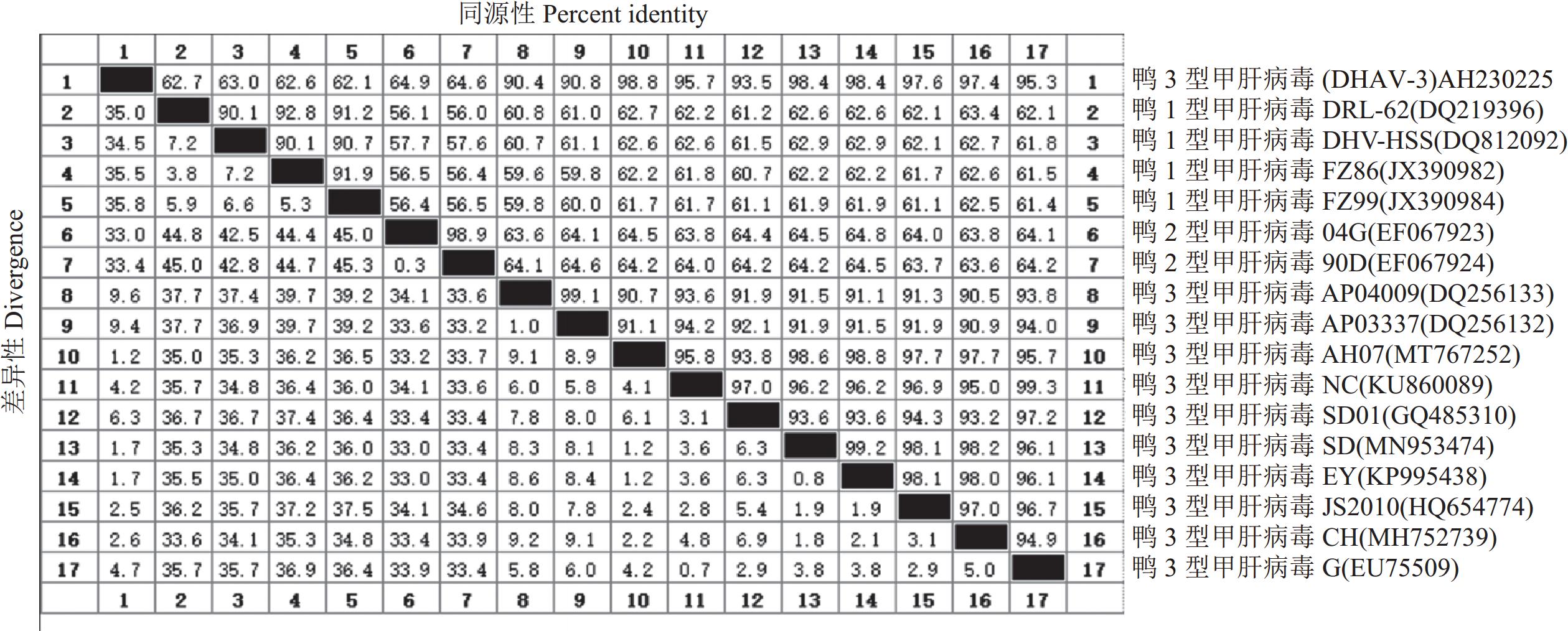
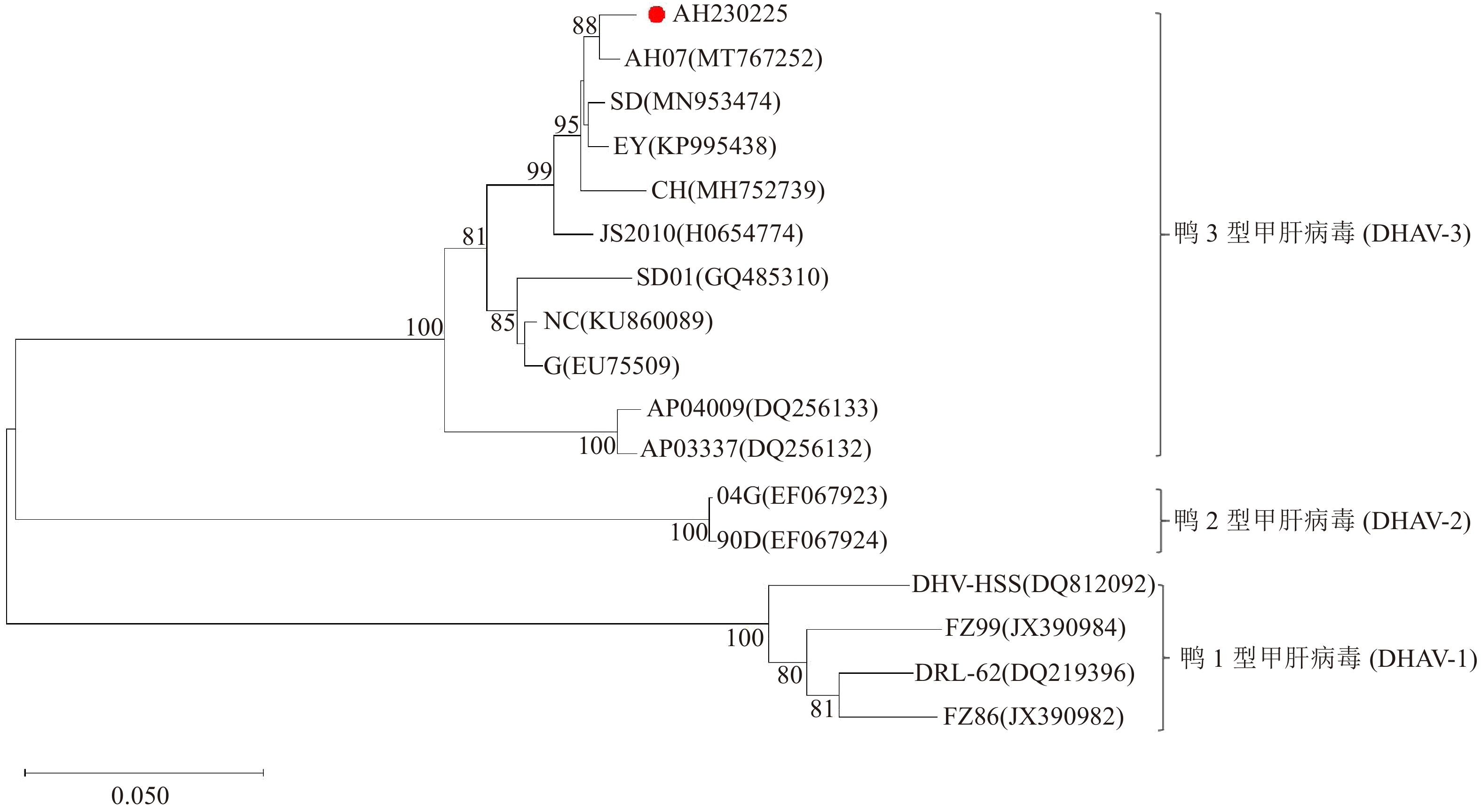
摘要:目的 探明引起安徽某鸭场雏鸭肝脏出血和大量死亡的病原及其遗传进化特征。方法 对安徽省某鸭场的病死雏鸭中采集的出血肝脏开展鸭已知病原核酸检测、病原分离鉴定和动物回归试验,在明确其病原为鸭3型甲肝病毒(Duck hepatitis A virus type 3, DHAV-3)的基础上分析其VP1基因序列分子特征。结果 细菌分离结果显示,未分离到细菌;经病毒核酸(RT-)PCR检测结果显示,鸭3型甲肝病毒(DHAV-3)核酸阳性,未检测出其他已知引起鸭肝出血的病毒核酸。将该阳性样品经鸭胚进行病毒分离与传代,发现接种后鸭胚发生死亡,胚体全身出血,对第5代尿囊液经RT-PCR检测为DHAV-3,将其命名为AH230225。经测定,该分离株的鸭胚半数致死量(Effective lethal dose 50, ELD50)为10−4.17/0.1 mL。动物回归试验表明,该毒株对樱桃谷雏鸭的致死率为80%,且攻毒死亡鸭肝脏和肾脏的剖检病变与临床典型病变相近。对该分离毒的VP1基因核苷酸序列进行同源性分析,显示AH230225株的VP1基因核苷酸序列与AH07株DHAV-3(安徽分离株)的同源性最高,为98.8%,与GenBank登录的10株DHAV-3分离株VP1基因核苷酸序列同源性为90.4%~98.8%,而与DHAV-1和DHAV-2的VP1基因核苷酸序列同源性分别为62.1%~63.0%、64.6%~64.9%;基于VP1蛋白氨基酸序列的遗传进化显示,该分离株与AH07株DHAV-3处于同一小进化分支上,亲缘关系最近;而与SD01株、G株和韩国株(AP-04009、AP-03337)等亲缘关系较远,即远离DHAV-1和DHAV-2进化分支。结论 引起安徽某鸭场雏鸭肝脏出血和大量死亡的病原为鸭3型甲肝病毒DHAV-3,同时明确了该毒株VP1基因的分子特征及遗传进化规律,为深入研究DHAV-3的致病机制和制定防控措施提供科学依据。Abstract:Objective Pathogen that caused duck liver hemorrhage in Anhui Province was identified and its genetics studied.Method A suspected virus strain was isolated from the ducks suffered from liver hemorrhage at a poultry farm in Anhui Province. Pathogenic nucleic acid and an animal regression test were employed to identify the culprit. VP1 of the confirmed duck hepatitis A virus type 3 (DHAV-3) were sequenced and analyzed using biological software.Results No bacterial pathogens were isolated from the culture of the diseased duck liver tissue. However, the specimens were tested positive for DHAV-3 but free of other viruses commonly known for duck liver hemorrhages by RT-PCR. The duck embryos inoculated with the isolate died with massive hemorrhages, and the 5th generation allantoic fluid detected presence of DHAV-3 by RT-PCR. The isolate was subsequently code-named AH230225 and determined to have the effective lethal dose 50 (ELD50) of 10−4.17/0.1 mL. In an animal regression test, the Cherry Valley ducklings had a mortality rate of 80%. The dissected lesions in the liver and kidneys of the dead ducks were similar to the typical clinical specimen. The sequenced VP1 nucleotides of AH230225 showed the greatest homology of 98.8% with the DHAV-3 of Anhui isolate, AH07. Its homologies with the 10 strains of DHAV-3 listed in GenBank were 90.4%~98.8%, with DHAV-1, 62.1%–63.0%, and with DHAV-2, 64.6%–64.9%. It appeared that the VP1 of AH230225 was genetically most closely related to that of AH07 but farther from those of SD01, G, or Korean AP-04009 and AP-03337. In other words, it was distant from DHAV-1 and DHAV-2 on the evolutionary branch.Conclusion The virus that caused the liver hemorrhage on ducks at the farm in Anhui was identified to be DHAV-3 with VP1 closely related genetically to that of AH07.
-
Keywords:
- Duck hepatitis A virus type 3 /
- isolation and identification /
- VP1
-
0. 引言
【研究意义】水稻光温敏核不育基因的发现与利用是选育两系杂交水稻的重要环节,利用基因编辑技术突变水稻光温敏核不育基因,对定向创制水稻光温敏核不育系具有重要意义。【前人研究进展】农垦58S是我国研究人员最早发现的一个水稻光温敏核不育系[1]。以农垦58S为不育基因供体,育种者选育了一系列光温敏核不育系应用于两系杂交稻生产[2]。张启发研究团队研究表明,pms3是决定农垦58S育性的光敏雄性核不育基因[3-4],它的转录本是一条长度为1 236 bp的长链非编码RNA,命名为长日特异性雄性不育相关RNA(long-day-specific-male-fertility-associated RNA,LDMAR)[5]。在农垦58S中,由一个C变为G的单碱基突变,使这条LDMAR的甲基化程度产生明显差异,造成长日照条件下农垦58S中的LDMAR转录量降低,促使发育中的花药提前进入程序化死亡,导致花粉不能正常发育,产生了水稻农垦58S光敏雄性核不育表型[5]。在短日条件下,水稻花粉粒的正常发育并不需要LDMAR来维持,所以农垦58S育性可以恢复[5]。庄楚雄课题组以温敏核不育系材料培矮64S为研究对象,发现ptms12-1(pms3)决定了培矮64S温敏不育特征。ptms12-1是一个非编码RNA基因,它的原始转录本在经过2次加工剪切后,最终形成了一个21 nt的小RNAosa-smR5864w[6]。水稻品种培矮64S的小RNA序列为CAU UGU UUG UCU ACC AUC CAU,而温敏不育水稻培矮64S在该小RNA序列第11个碱基处存在一个由C变为G的单碱基突变(CAU UGU UUG UGU ACC AUC CAU),形成了osa-smR5864m。在高温条件下,这个单碱基突变一方面可能影响了小RNA的表达水平,另一方面也有可能影响了小RNA与下游靶基因的结合能力而最终导致温敏雄性不育特征[6]。以上两项研究结果表明,pms3位点的同一个SNP在粳稻农垦58S中产生了光敏不育性,而在籼稻培矮64S内产生了温敏不育性,说明了水稻光温敏核不育性遗传机理的复杂性。由于osa-smR5864w是一非编码小RNA,其编码序列上的任何新改变都有可能影响其正常表达量或改变其对下游基因的调控功能,从而使突变体表现类似农垦58S的光温敏核不育特征。近年来以TALEN和CRISPR/Cas-9为代表的基因编辑技术已广泛应用于水稻基因定向突变,快速有效地获得了定向改良性状[7-12]。【本研究切入点】利用基因编辑技术对编码osa-smR5864w的序列进行定向突变,可能创制出新的光温敏核不育复等位基因,但目前尚未见到相关报道。【拟解决的关键问题】由于小RNA osa-smR5864w上的原始突变位置附近无理想的CRISPR/Cas-9编辑所需的NGG结构,因此本研究利用TALEN方法对编码该小RNA的基因组区间进行定点突变。构建了1个靶向水稻PMS3的TALEN载体,以籼稻品种明恢86和粳稻品种日本晴品种为遗传突变对象,期望通过定向突变PMS3基因,获得不同类型的小RNA osa-smR5864序列突变体,分析不同突变体的雄性育性光温反应变化规律,旨在为基于定向编辑水稻PMS3基因的方法培育水稻两系不育系材料提供科学依据。
1. 材料与方法
1.1 试验材料
粳稻品种日本晴,由福建省农业遗传工程重点实验室保存。明恢86,由三明市农业科学院提供。大肠杆菌Escherichia coli感受态DH5α和根癌农杆菌Agrobacterium tumefaciens感受态LBA4404由福建省农业遗传工程重点实验室保存。TALEN载体构建试剂盒由上海斯丹赛生物技术有限公司提供。
1.2 试验方法
1.2.1 载体构建及水稻遗传转化
选择5′-GGT GAA GCA AAG AAG-3′及5′-ACT CTT GAT GGA T-3′序列作为TALEN载体的左右臂,设计的基因编辑靶点位于小RNA osa-smR5864w之上。利用上海斯丹赛生物技术有限公司提供的TALEN载体构建试剂盒,参照说明书中的方法,构建TALEN载体。将构建好的质粒通过热激转化到DH5α大肠杆菌感受态细胞。挑取3个正常生长的单克隆培养,于铂尚生物技术(福州)有限公司测序,正向测序引物序列为305F:5′-CTC CCC TTC AGC TGG ACA C-3′,反向测序引物序列为306R:5′-AGC TGG GCC ACG ATT GAC-3′。将测序结果中的核酸序列翻译成氨基酸形式后进行比对,验证翻译的TALE蛋白是否正确,选取1个序列正确的质粒命名为TALEN-PMS3,将该质粒载体电击导入LBA4404感受态细胞中,通过农杆菌介导的方法[13]转化日本晴和明恢86的愈伤组织,获得T0代植株。
1.2.2 转基因植株鉴定
使用CTAB法[14]从水稻叶片中提取基因组DNA。以T0代各单株的基因组DNA为模板,通过上游引物HptF和下游引物HptR(表 1)PCR扩增潮霉素磷酸转移酶基因(标记基因目的条带大小为419 bp),以扩增出目的条带的为转基因阳性株。以T1和T2代各单株的基因组DNA为模板,通过HptF/HptR进行PCR扩增潮霉素磷酸转移酶基因,无目的条带的单株即为分离出的非转基因植株。
表 1 试验所用引物Table 1. Primers employed引物
Primer序列(5′- 3′)
Sequence(5′- 3′)试验目的
ApplicationHptF AAGCTGCATCATCGAAATTG 转基因阳性株检测 HptR TCGTTATGTTTATCGGCACT 305F CTCCCCTTCAGCTGGACAC TALEN质粒验证 306R AGCTGGGCCACGATTGAC LDF1 TAAGAACTGCTGCTCCAAAT PMS3基因扩增、测序 LDR1 CGGCTCCGTTATAGATAGAC 1.2.3 靶位点突变检测
以水稻PMS3基因组序列为参考,设计正向引物LDF1和反向引物LDR1(表 1),PCR扩增T0、T1和T2代各植株的PMS3基因,PCR扩增产物预计410 bp。PCR产物于铂尚生物技术公司(福州)进行测序,通过比对测序结果鉴定PMS3突变体。
1.2.4 突变体育性观察及鉴定
T2代pms3突变体及野生型对照在福建省农业科学院生物技术研究所福州寿山基地作中稻种植,明恢86来源的材料于5月20播种,日本晴来源的材料于6月20日播种,秧苗于25 d秧龄时移栽于大田,每份材料种植100株,采取正常田间管理。水稻抽穗时,摘取即将开花的小穗上、中、下各2个颖花制片,用1% I2+0.1% IK对花粉进行染色,每份材料制取10个样本,利用显微镜观察并计算花粉黑染率。套袋10个单穗,25 d后调查自交结实率。
2. 结果与分析
2.1 TALEN系统介导的PMS3序列特异性突变分析
构建了TALEN-PMS3载体,通过农杆菌介导的方法将该载体转化日本晴和明恢86的愈伤组织。共获25个转基因T0代克隆,其中日本晴获10个克隆,明恢86获15个克隆。对每个克隆靶位点序列的测序结果显示,在日本晴中有4株产生了突变,明恢86有5株产生了突变,突变率分别为40.0%和33.3%。
通过T0代植株自交,收获了上述含有靶序列突变的9株水稻T1代种子。在T1代中,通过对单株潮霉素磷酸转移酶基因及突变位点的检测,分离出非转基因纯合突变单株。各种纯合突变体序列如图 1和表 2所示。以日本晴为背景,获得了3种纯合突变类型(插入1 bp,缺失1 bp和缺失4 bp),以明恢86为背景,获得了5种纯合突变类型(插入12 bp,缺失3 bp,缺失4 bp和缺失62 bp,以及缺失82 bp外加插入23 bp)。以上突变体序列分析表明,通过TALEN方法,在日本晴和明恢86背景下都成功获得了PMS3序列的定向突变。
![]() 图 1 T1代pms3纯合突变体序列注:WT表示野生型序列; N-pms3-#和M-pms3-#分别代表日本晴和明恢86背景中的突变植株。在野生型序列中,osa-smR5864的编码序列标注为灰底字母,其中带下划线的碱基G为农垦58S的原始突变位点。黑色三角形所处位置为缺失突变发生的位置,空心三角形处为插入突变发生的位置。Figure 1. Sequencing chromatograms of mutations in T1 homozygous pms3 mutant linesNote: WT indicates wild-type sequence; N-pms3-# and M-pms3-# represent the mutant plants in Nipponbare and MH86 backgrounds, respectively. In the wild type sequence, the osa-smR5864 sequence is marked in gray letters, the underlined G is the original mutation site of Nongken 58S.The deletion and insertion sites are indicated by black triangle and hollow triangle, respectively.表 2 T1代pms3纯合突变类型Table 2. Homozygous mutation types of T1 pms3 lines
图 1 T1代pms3纯合突变体序列注:WT表示野生型序列; N-pms3-#和M-pms3-#分别代表日本晴和明恢86背景中的突变植株。在野生型序列中,osa-smR5864的编码序列标注为灰底字母,其中带下划线的碱基G为农垦58S的原始突变位点。黑色三角形所处位置为缺失突变发生的位置,空心三角形处为插入突变发生的位置。Figure 1. Sequencing chromatograms of mutations in T1 homozygous pms3 mutant linesNote: WT indicates wild-type sequence; N-pms3-# and M-pms3-# represent the mutant plants in Nipponbare and MH86 backgrounds, respectively. In the wild type sequence, the osa-smR5864 sequence is marked in gray letters, the underlined G is the original mutation site of Nongken 58S.The deletion and insertion sites are indicated by black triangle and hollow triangle, respectively.表 2 T1代pms3纯合突变类型Table 2. Homozygous mutation types of T1 pms3 lines品种背景
Background株系号
Line突变序列
Mutation sequences突变类型
Mutation types日本晴
NipponbareN-pms3-#1 ATGGATGGTAGACAA-CAATG -1 N-pms3-#2 ATGGATGGTAGA----CAATG -4 N-pms3-#3 ATTGGATGGTAGACAAACAATG +1 N-pms3-#4 ATGGATGGTAGACAA-CAATG -1 明恢86
MH86M-pms3-#1 ATGGATGGATCTAGAGTATCTGAA +12 M-pms3-#2 ATGGATGGTAGA-----------… -82 & +23 M-pms3-#3 ATGGATGGTAGAC---CAATG -3 M-pms3-#4 ATGGATGGTAGA----CAATG -4 M-pms3-#5 …--------------------TG -62 注:短划线和灰底字母分别表示缺失和插入突变,…表示已省略的序列。带下划线的G碱基为农垦58S的原始突变位点。+:插入,-:缺失,具体数字表示所涉及的核苷酸数。
Note:Deletion, insertion and omitted sequences are indicated by dashes, gray letters and dots, respectively. The underlined G is original mutation site of Nongken 58S. +: insertion, -: deletion. Data indicate number of nucleotides involved.2.2 pms3纯合突变体花粉育性正常
在福州作中稻种植时,所有日本晴和明恢86 pms3突变体及野生型亲本于8月15~20日开始抽穗。在开花期对N-pms3-#1、N-pms3-#2、N-pms3-#3、M-pms3-#1、M-pms3-#2、M-pms3-#5的T2代纯合突变体花药外型进行观察并利用碘染法对花粉的育性进行统计,结果列于表 3。分析结果显示,所有pms3突变体的花药外型正常,能够正常开裂并散粉,花粉粒经过碘-碘化钾染色后,表现为正常染色的可育花粉,突变体与野生型花粉黑染率均为90%左右,无显著差异(图 2、表 3)。而光温敏核不育系培矮64S表现为花药瘦小不开裂,花粉粒经过碘-碘化钾染色后,镜检结果显示全部为典败型花粉,无黑染花粉粒。以上结果表明,本试验通过定向突变日本晴及明恢86的PMS3基因,所获得的pms3突变体花粉在福州夏季自然长日高温条件下表现正常可育,并不能转变为不育花粉。
表 3 T2代pms3纯合突变体花粉育性及结实率Table 3. Pollen fertility and seed setting rates of T2 pms3 mutant lines材料
Lines花粉黑染率
Stained pollens rate/%每穗结实率
Seed setting rates of individual panicles/%平均结实率
Average seed setting rates/%明恢86MH86 90.3±6.2 a 95.0 86.1 93.6 94.1 89.7 78.9 83.6 82.3 76.8 91.2 87.1±6.6 a M-pms3-#1 89.9±5.1 a 88.1 92.7 91.0 89.5 85.3 90.2 77.5 81.6 80.8 85.4 86.2±5.0 a M-pms3-#2 91.5±6.1 a 91.2 89.3 90.6 88.5 79.9 91.4 87.3 82.2 78.4 86.7 86.6±4.8 a M-pms3-#5 90.5±5.8 a 90.8 83.4 84.5 78.8 91.3 92.8 76.3 87.2 80.3 87.6 85.3±5.6 a 日本晴Nipponbare 92.1±6.8 a 97.8 87.6 85.5 93.0 91.4 82.4 88.9 89.6 88.2 88.6 89.3±4.2 a N-pms3-#1 91.8±5.5 a 95.3 89.2 86.3 91.1 93.9 81.5 87.8 86.5 90.1 84.3 88.6±4.2 a N-pms3-#2 92.0±4.8 a 86.2 90.2 93.1 86.9 90.8 89 92.3 89.6 93.5 86.0 89.8±2.8 a N-pms3-#3 91.9±6.0 a 89.0 91.5 88.7 92.1 85.2 90.8 87.4 90.1 90.2 91.0 89.6±2.1 a 培矮64S P64S 0 0 0 0 0 0 0 0 0 0 0 0 注:平均花粉黑染率为10穗黑染率的平均值±标准差。同列数据后无相同小写字母表示在0.05水平差异显著(P < 0.05)。
Note:The average pollen stained rates were the mean ± standard deviation calculated from 10 panicles. The average seed setting rates were the mean ± standard deviation calculated from 10 panicles and different lowercase letters behind the number in the sane column indicated significant difterences(P < 0.05).![]() 图 2 福州自然长日高温下pms3突变体花粉育性正常注:标尺长度上4图为1 mm,下4图为100 μm。Figure 2. Anther morphology and pollen fertility of pms3 mutants showing similarities to those of wild types under natural long-day-high-temperature conditions in FuzhouNote: Scale length for the upper four graphs is 1 mm, for the lower four graphs is 100 μm.
图 2 福州自然长日高温下pms3突变体花粉育性正常注:标尺长度上4图为1 mm,下4图为100 μm。Figure 2. Anther morphology and pollen fertility of pms3 mutants showing similarities to those of wild types under natural long-day-high-temperature conditions in FuzhouNote: Scale length for the upper four graphs is 1 mm, for the lower four graphs is 100 μm.2.3 pms3纯合突变体自交结实率正常
抽穗开花期对T2代纯合突变体N-pms3-#1、N-pms3-#2、N-pms3-#3、M-pms3-#1、M-pms3-#2和M-pms3-#5进行单穗套袋,种子成熟后分单穗统计自交结实率,调查结果显示明恢86的平均结实率为(87.1±6.6)%,3个明恢86来源的pms3突变体结实率约为86%,与明恢86无显著差异,而雄性不育系培矮64S的结实率为0(表 3)。所有日本晴pms3突变体的结实率约为89%,与野生型日本晴也无显著差异。以上结实率统计结果表明,本试验通过TALEN方法虽然在日本晴和明恢86的osa-smR5864小RNA编码序列上产生了多种类型的突变,但是这些突变体在福州夏季长日高温条件下,并不能表现出类似农垦58S或培矮64S那样的光温敏雄性核不育特征。
3. 讨论与结论
诸多研究者对农垦58S的光温敏核不育基因做了大量研究,基于不同的研究材料,认为不同的水稻杂交组合中引起光敏核不育的遗传基因不同[15-17]。位于12号染色体上的pms3是形成农垦58S光敏核不育的原始突变位点[3-4]。水稻PMS3的转录产物是一条非编码RNA,它的原始转录本在经过2次加工剪切后,最终形成了一个21nt的小RNA(osa-smR5864w)[6]。该小RNA序列第11个碱基处一个由C变为G的单碱基突变,形成了osa-smR5864m,其序列为5′-CAU UGU UUG UGU ACC AUC CAU-3′,这一突变的小RNA决定了农垦58S和培矮64S的光温敏雄性不育特性[6]。
本项研究利用TALEN基因编辑技术,在粳稻品种日本晴和籼稻品种明恢86的osa-smR5864m小RNA编码序列上共产生了7种类型的插入缺失突变,有些突变类型涉及的插入或缺失碱基数较多(表 2,图 1)使osa-smR5864m序列产生了严重改变。例如,M-pms3-#5缺失了62个碱基,几乎导致编码osa-smR5864m的序列完全缺失。然而,这些突变体在福州夏季长日高温条件下,并不能表现出类似农垦58S或培矮64S那样的光温敏雄性核不育特征,这一结果与本研究的预期不符。
基于前人的研究及本试验的结果,推测有两个因素决定了这种现象。首先,可能只有原始突变处由C变为G的单碱基突变类型才能产生光温敏雄性不育表型,但因为本研究并未获得这种突变类型,所以定向突变获得的7种pms3突变体都未表现出光温敏雄性核不育特性。其次,研究者已从农垦58S中克隆出PMS1[16-17]与PMS3两个光温敏核不育基因,它们都编码21nt的小RNA,这两种小RNA在诱导水稻产生光温敏核不育性状上或许具有互作关系,当PMS1位点为野生型时,单纯突变PMS3位点可能不足以诱导形成水稻光温敏核不育表型。以上两种假设还有待进一步的实验予以解释验证。
本研究通过定向突变粳稻日本晴和籼稻明恢86的PMS3基因,使小RNA osa-smR5864的编码序列发生严重改变,但突变体并不能产生水稻光温敏雄性核不育表型,这一结果说明了PMS3位点调控水稻光温敏核不育分子机制的复杂性,具体机理有待进一步实验予以解析。
-
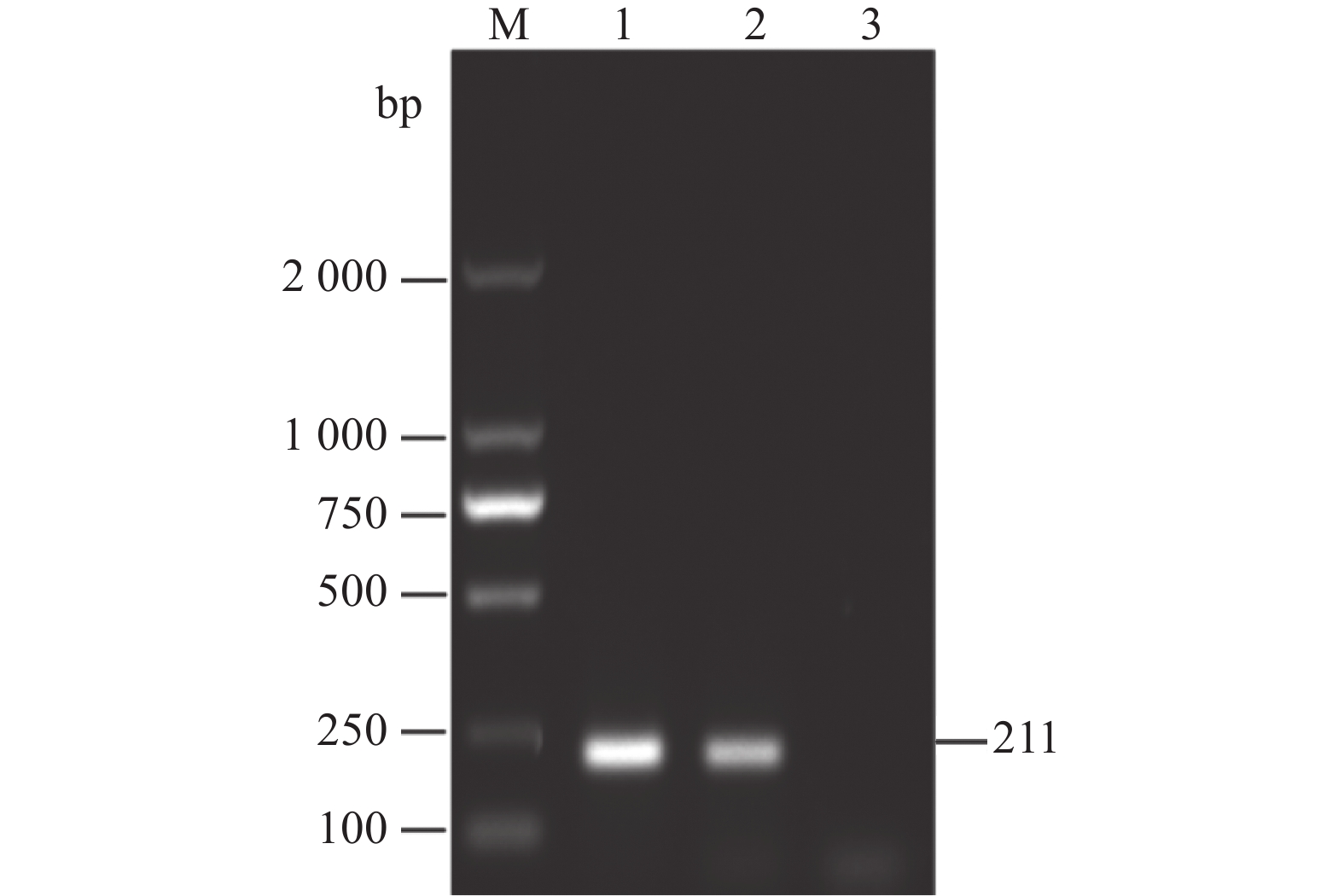
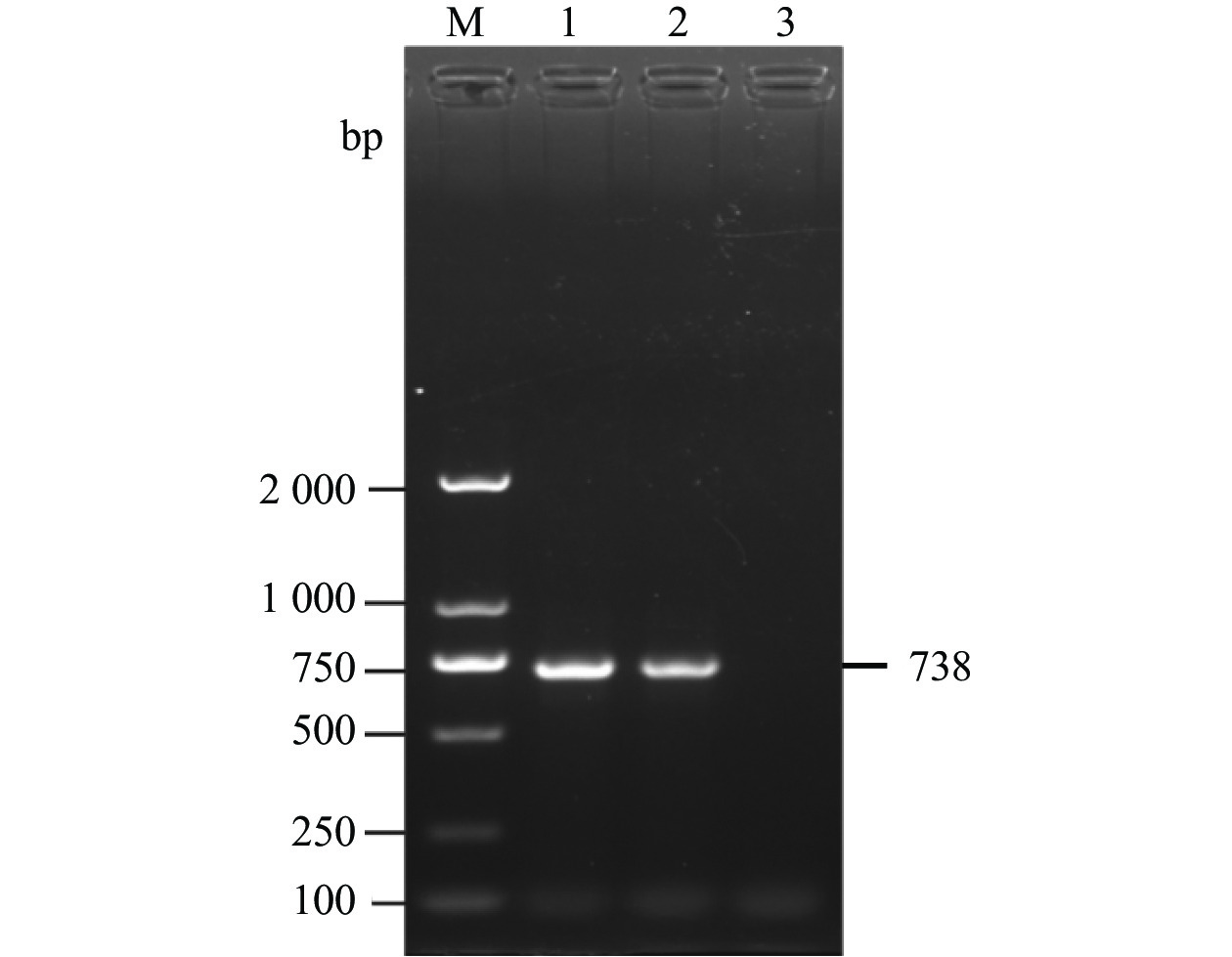
图 1 临床样品RT-PCR检测
M为2000 DNA Maker,1为鸭3型甲肝病毒,2为鸭3型腺病毒,3为禽流感病毒,4为番鸭细小病毒,5为鹅细小病毒,6为番鸭呼肠孤病毒,7为鸭1型甲肝病毒,8为鸭星状病毒,9为阴性对照。
Figure 1. RT-PCR detection on clinical samples
M: 2000 DNA marker; 1: DHAV-3; 2: duck adenovirus type 3 (DAdV-3); 3: avian influenza virus (AIV); 4: Muscovy duck parvovirus (MDPV); 5: goose parvovirus (GPV); 6: Muscovy duck reovirus (MDRV); 7: DHAV-1; 8: duck astrovirus (DAstV); 9: control.
表 1 RT-PCR扩增引物
Table 1 Primers for RT-PCR amplification
引物名称
Primers引物序列
Sequences(5′-3′)退火温度
Annealing temperature/ ℃片段长度
Product size/bpDHAV-3 VP1 F:ATGGCCGCCAATAACCAGGGTGATTC 55 738 R:TTCAATTTCCAAATGGAGCTCAAAGGCAAGTG DHAV-3 F:CACAAACAGCGTAAACGCCA 52 211 R:GCAAGCCTTGGGGATTTTGG 表 2 DHAV参考毒株
Table 2 Reference DHAV strains
登录号
Locus毒株
Virus血清型
Serotype分离时间
Time分离地
IsolateJX390982 FZ86 DHAV-1 1986年 中国福建
Fujian, ChinaDQ219396 DRL-62 DHAV-1 1962年 美国
USAJX390984 FZ99 DHAV-1 1999年 中国福建
Fujian, ChinaDQ812092 DHV-HSS DHAV-1 1995年 韩国
KoreaEF067924 90D DHAV-2 1990年 中国台湾
Taiwan, ChinaEF067923 04G DHAV-2 2004年 中国台湾
Taiwan, ChinaDQ256133 AP-04009 DHAV-3 2004年 韩国
KoreaDQ256132 AP-03337 DHAV-3 2003年 韩国
KoreaEU75509 G DHAV-3 2002年 中国广西
Guangxi, ChinaKU860089 NC DHAV-3 - - GQ485310 SD01 DHAV-3 2008年 中国山东
Shandong, ChinaHQ654774 JS2010 DHAV-3 2010年 中国江苏
Jiangsu, ChinaMH752739 CH DHAV-3 2015年 中国四川
Sichuan, ChinaKP995438 EY DHAV-3 2014年 中国山东
Shandong, ChinaMN953474 SD DHAV-3 2011年 中国山东
Shandong, ChinaMT767252 AH07 DHAV-3 2018年 中国安徽
Anhui, China表 3 樱桃谷鸭动物回归试验结果
Table 3 Animal regression test results on Cherry Valley ducks
组别
Groups数量
Number/
羽攻毒后7 d内的死亡鸭数量
Number of dead ducklings during
7 days post infectious/羽死亡率
Mortality/%1 2 3 4 5 6 7 AH230225 5 0 0 2 1 1 0 0 80 (4/5) 对照组CK 5 0 0 0 0 0 0 0 0(0/5) -
[1] 苏敬良, 张国中, 黄瑜, 等. 血清3型鸭甲型肝炎病毒弱毒疫苗株培育及免疫原性研究 [J]. 中国兽医杂志, 2009, 45(12):11−14. DOI: 10.3969/j.issn.0529-6005.2009.12.003 SU J L, ZHANG G Z, HUANG Y, et al. Development of attenuated duck hepatitis A virus serotype 3 strain and evaluation of its antigencity [J]. Chinese Journal of Veterinary Medicine, 2009, 45(12): 11−14. (in Chinese) DOI: 10.3969/j.issn.0529-6005.2009.12.003
[2] ZHANG Y Y, WU S, LIU W B, et al. Current status and future direction of duck hepatitis A virus vaccines [J]. Avian Pathology, 2023, 52(2): 89−99. DOI: 10.1080/03079457.2022.2162367
[3] FEHÉR E, JAKAB S, BALI K, et al. Genomic epidemiology and evolution of duck hepatitis A virus [J]. Viruses, 2021, 13(8): 1592. DOI: 10.3390/v13081592
[4] TSENG C H, TSAI H J. Molecular characterization of a new serotype of duck hepatitis virus [J]. Virus Research, 2007, 126(1/2): 19−31.
[5] RAJENDRAN R, SRINIVASAN J, NATARAJAN J, et al. First report of Duck Hepatitis A virus genotype 2 in India [J]. Veterinary Research Communications, 2023, 47(3): 1231−1241. DOI: 10.1007/s11259-022-10063-0
[6] 苏敬良, 黄瑜, 贺荣莲, 等. 新型鸭肝炎病毒的分离及初步鉴定 [J]. 中国兽医科技, 2002, 32(1):15−16. DOI: 10.3969/j.issn.1673-4696.2002.01.005 SU J L, HUANG Y, HE R L, et al. Isolation and preliminary identification of a new duck hepatitis virus [J]. Chinese Journal of Veterinary Science and Technology, 2002, 32(1): 15−16. (in Chinese) DOI: 10.3969/j.issn.1673-4696.2002.01.005
[7] 胡薛英, 蔡双双, 谷长勤, 等. 新型鸭肝炎病毒感染雏鸭血液生化指标的动态变化 [J]. 中国兽医学报, 2005, 25(6):628−631. DOI: 10.3969/j.issn.1005-4545.2005.06.021 HU X Y, CAI S S, GU C Q, et al. Dynamic changes of biochemical indexes of ducklings infected with new type duck hepatitis virus [J]. Chinese Journal of Veterinary, 2005, 25(6): 628−631. (in Chinese) DOI: 10.3969/j.issn.1005-4545.2005.06.021
[8] 施少华, 程龙飞, 傅光华, 等. 鸭肝炎病毒新血清型基因组序列分析 [J]. 微生物学报, 2009, 49(3):309−315. DOI: 10.3321/j.issn:0001-6209.2009.03.005 SHI S H, CHENG L F, FU G H, et al. Genomic sequence of a new serotype duck hepatitis virus [J]. Acta Microbiologica Sinica, 2009, 49(3): 309−315. (in Chinese) DOI: 10.3321/j.issn:0001-6209.2009.03.005
[9] KIM M C, KWON Y K, JOH S J, et al. Recent Korean isolates of duck hepatitis virus reveal the presence of a new geno- and serotype when compared to duck hepatitis virus type 1 type strains [J]. Archives of Virology, 2007, 152(11): 2059−2072. DOI: 10.1007/s00705-007-1023-0
[10] DING C Y, ZHANG D B. Molecular analysis of duck hepatitis virus type 1 [J]. Virology, 2007, 361(1): 9−17. DOI: 10.1016/j.virol.2007.01.007
[11] KIM M C, KWON Y K, JOH S J, et al. Molecular analysis of duck hepatitis virus type 1 reveals a novel lineage close to the genus Parechovirus in the family Picornaviridae[J]. Journal of General Virology, 2006, 87(Pt 11): 3307-3316.
[12] LIU G Q, WANG F, NI Z, et al. Genetic diversity of the VP1 gene of duck hepatitis virus type I (DHV-I) isolates from Southeast China is related to isolate attenuation [J]. Virus Research, 2008, 137(1): 137−141. DOI: 10.1016/j.virusres.2008.04.030
[13] FU Y, PAN M, WANG X Y, et al. Complete sequence of a duck astrovirus associated with fatal hepatitis in ducklings[J]. Journal of General Virology, 2009, 90(Pt 5): 1104-1108.
[14] LI X J, ZHAO R, LIN W, et al. Evidence of VP1 of duck hepatitis A type 1 virus as a target of neutralizing antibodies and involving receptor-binding activity [J]. Virus Research, 2017, 227: 240−244. DOI: 10.1016/j.virusres.2016.10.018
[15] 傅秋玲, 傅光华, 陈红梅, 等. 胰腺炎型鸭1型甲肝病毒结构蛋白VP1基因的克隆和表达 [J]. 福建农业学报, 2014, 29(5):409−412. FU Q L, FU G H, CHEN H M, et al. Cloning and prokaryotic expression of the VP1 gene of pancreotropic duck hepatitis type 1 virus [J]. Fujian Journal of Agricultural Sciences, 2014, 29(5): 409−412. (in Chinese)
[16] 傅秋玲, 傅光华, 陈翠腾, 等. 半番鸭源鸭1型甲肝病毒亚型的分离鉴定及其VP1基因分析 [J]. 福建农业学报, 2017, 32(8):813−817. FU Q L, FU G H, CHEN C T, et al. Identification and sequencing of duck hepatitis A virus 1 subtype a isolated from mule ducklings [J]. Fujian Journal of Agricultural Sciences, 2017, 32(8): 813−817. (in Chinese)
[17] LIU R C, CHEN C T, HUANG Y, et al. Microbiological identification and analysis of waterfowl livers collected from backyard farms in Southern China [J]. Journal of Veterinary Medical Science, 2018, 80(4): 667−671. DOI: 10.1292/jvms.17-0452
[18] 程龙飞, 刘荣昌, 傅光华, 等. 鸭3型腺病毒的分离鉴定及其fiber基因分析 [J]. 中国家禽, 2019, 41(11):47−50. CHENG L F, LIU R C, FU G H, et al. Isolation and identification of duck adenovirus 3 and sequence analysis of fiber gene [J]. China Poultry, 2019, 41(11): 47−50. (in Chinese)
[19] 刘家森, 姜骞, 司昌德, 等. 番鸭细小病毒与鹅细小病毒PCR鉴别诊断方法的建立 [J]. 中国兽医科学, 2007, 37(6):469−472. LIU J S, JIANG Q, SI C D, et al. Establishment of PCR assay for differentiation of Muscovy duck parvovirus from goose parvovirus [J]. Veterinary Science in China, 2007, 37(6): 469−472. (in Chinese)
[20] 胡奇林, 林锋强, 陈少莺, 等. 应用RT-PCR技术检测番鸭呼肠孤病毒 [J]. 中国兽医学报, 2004, 24(3):231−232. HU Q L, LIN F Q, CHEN S Y, et al. Detection of Muscovy duck reovirus by RT-PCR [J]. Chinese Journal of Veterinary, 2004, 24(3): 231−232. (in Chinese)
[21] LIN Y, YANG J, HE D L, et al. Differently expression analysis and function prediction of long non-coding RNAs in duck embryo fibroblast cells infected by duck tembusu virus [J]. Frontiers in Immunology, 2020, 11: 1729. DOI: 10.3389/fimmu.2020.01729
[22] DOAN H T T, LE X T K, DO R T, et al. Molecular genotyping of duck hepatitis A viruses (DHAV) in Vietnam [J]. Journal of Infection in Developing Countries, 2016, 10(9): 988−995. DOI: 10.3855/jidc.7239
[23] HASSAN T I R, EID A A M, GHANEM I A I, et al. First report of duck hepatitis A virus 3 from duckling flocks of Egypt [J]. Avian Diseases, 2020, 64(3): 269−276.
[24] ROHAIM M A, NAGGAR R F E, ABDELSABOUR M A, et al. Insights into the genetic evolution of duck hepatitis A virus in Egypt [J]. Animals, 2021, 11(9): 2741. DOI: 10.3390/ani11092741
[25] YEHIA N, ERFAN A M, OMAR S E, et al. Dual circulation of duck hepatitis A virus genotypes 1 and 3 in Egypt [J]. Avian Diseases, 2021, 65(1): 1−9.
[26] WEN X, ZHU D, CHENG A, et al. Molecular epidemiology of duck hepatitis a virus types 1 and 3 in China, 2010-2015 [J]. Transboundary and Emerging Diseases, 2018, 65(1): 10−15. DOI: 10.1111/tbed.12741
[27] 徐倩, 陈琳琳, 张瑞华, 等. 鸭甲肝病毒3型2012年山东分离株VP1基因的序列分析 [J]. 病毒学报, 2013, 29(5):522−528. XU Q, CHEN L L, ZHANG R H, et al. Sequence analysis of VP1 gene of the duck hepatitis A virus type 3 strains isolated from Shandong Province of China in 2012 [J]. Chinese Journal of Virology, 2013, 29(5): 522−528. (in Chinese)
[28] ZHANG R, XIA L, CHEN J, et al. Molecular epidemiology and genetic diversity of duck hepatitis A virus type 3 in Shandong province of China, 2012–2014 [J]. Acta Virologica, 2017, 61(4): 463−472. DOI: 10.4149/av_2017_409
[29] YANG C T, SHAH P T, BAHOUSSI A N, et al. Duck hepatitis a virus: Full-length genome-based phylogenetic and phylogeographic view during 1986–2020 [J]. Virus Research, 2023, 336: 199216. DOI: 10.1016/j.virusres.2023.199216
[30] NIU Y J, MA H Y, DING Y H, et al. The pathogenicity of duck hepatitis A virus types 1 and 3 on ducklings [J]. Poultry Science, 2019, 98(12): 6333−6339. DOI: 10.3382/ps/pez455
[31] LIU R C, SHI S H, HUANG Y, et al. Comparative pathogenicity of different subtypes of duck hepatitis A virus in Pekin ducklings [J]. Veterinary Microbiology, 2019, 228: 181−187. DOI: 10.1016/j.vetmic.2018.11.030
[32] 管飘萍, Enkhbayar Munkhbayar, 黄紫贝, 等. 22株鸭甲型肝炎病毒的分离鉴定及其VP1基因的序列分析 [J]. 中国家禽, 2021, 43(1):105−109. GUAN P P, MUNKHBAYAR E, HUANG Z B, et al. Isolation, identification and sequening of VP1 gene of 22 isolates of duck hepatitis A virus [J]. China Poultry, 2021, 43(1): 105−109. (in Chinese)
[33] GAO J M, CHEN J H, SI X K, et al. Genetic variation of the VP1 gene of the virulent duck hepatitis A virus type 1 (DHAV-1) isolates in Shandong province of China [J]. Virologica Sinica, 2012, 27(4): 248−253. DOI: 10.1007/s12250-012-3255-8
-
期刊类型引用(3)
1. 杨大兵,胡亮,杜雪树,万丙良,夏明元,戚华雄,李进波. CRISPR/Cas9基因编辑技术创制水稻雄性不育系的研究进展. 中国农业科技导报(中英文). 2025(03): 24-34 .  百度学术
百度学术
2. 唐杰,龙湍,吴春瑜,李新鹏,曾翔,吴永忠,黄培劲. 水稻光温敏雄性不育突变体tms3650的鉴定和基因定位. 中国水稻科学. 2023(01): 45-54 .  百度学术
百度学术
3. 谭瑗瑗,汪庆,富昊伟,张渭章,吴三玲,舒庆尧. 利用花培与辐照诱变培育粳型两系不育系江79S. 核农学报. 2022(06): 1073-1079 .  百度学术
百度学术
其他类型引用(3)




 下载:
下载: