Identification and Expression Analysis of ETR Gene Family in Camellia sinensis
-
摘要:目的 鉴定并分析CsETR基因,预测其潜在分子功能,以期从分子水平了解乙烯受体在茶树生长过程中应对胁迫的作用机制。方法 采用生物信息学分析方法鉴定ETR基因家族成员并预测其潜在分子功能,采用实时荧光定量分析法分析其在不同胁迫处理下的表达水平。结果 从茶树基因组中确定了6个茶树ETR基因家族成员,均包含N端跨膜区、GAF区和组氨酸(His)激酶结构域;系统发育树分析显示其为两组,与乙烯受体的两个亚家族分别对应;茶树ETR基因家族中每个成员含有1~12个外显子。ETR家族基因在茶树果实和茎中表达量较高,且该家族表现出明显的组织表达特异性。实时荧光定量分析结果显示:低温处理下,6个基因的表达水平上调,其中ERS1-1基因的表达水平上调最为明显;在植物生长调节剂ABA、JA和GA处理下大部分基因表达水平上调,其中ERS1-3基因上调最为明显,而ETR2-2基因在ABA处理下表达水平呈缓慢下调趋势。结论 共鉴定出6个CsETR基因家族成员,并预测分析了其潜在的分子功能,该基因家族成员在果实和茎中表达量较高,且低温能诱导CsETR基因的表达,其中ERS1-3基因在ABA、MeJA和GA的处理下表达量显著上调。Abstract:Objective CsETRs were identified and analyzed to predict the potential molecular functions involving the mechanism of ethylene receptor in response to stress in tea plants.Method Bioinformatics was used to identify members in the ETR family and predict their potential molecular functions. Real-time fluorescence quantitative analysis was employed for the expressions of the genes under stresses.Result Six ETRs were identified from the genome of tea plants. They all consisted of N-terminal transmembrane region, GAF region, and histidine (His) kinase domain. Phylogenetic tree analysis divided them into two groups. Each member of the ETR family contained 1-12 exons. Significantly differentiated in tissues, the expressions of ETRs were high in the fruits and stems. The fluorescence quantitative expressions of the 6 genes, especially ERS1-1, were upregulated to varying degrees when exposed to low temperature. Under the stress of plant growth regulators ABA, JA, or GA, most of the genes were upregulated, especially ERS1-3, but ETR2-2 downregulated slowly when treated by ABA.Conclusion On tea plants, 6 CsETRs in the family were identified with their potential molecular functions predicted and analyzed in this study. CsETRs were highly expressed in the fruits and stems that could be induced by low-temperature stress. Whereas ABA, MeJA, or GA could significantly upregulate the expression of ERS1-3.
-
Keywords:
- Camellia sinensis /
- ETR gene /
- plant growth regulators /
- abiotic stress /
- expression analysis
-
0. 引言
【研究意义】茶树Camellia sinensis(L.)O. Kuntze是我国重要的无酒精饮料作物[1],在生长过程中容易受到低温、干旱、洪涝等胁迫,从而影响茶树的正常生长发育,降低茶叶的产量和质量。植物通过调节乙烯敏感性来对环境胁迫产生反应。乙烯受体ETR(Ethylene receptor)是乙烯信号转导途径的第一个元件,乙烯受体基因表达对调控乙烯信号传递,从而调节植物生长起着重要作用。因此,鉴定并分析乙烯受体基因,挖掘其相关的功能,以期从分子水平了解乙烯受体在茶树生长过程中应对胁迫的作用机制。【前人研究进展】乙烯是通过其与受体的结合来被植物感知的。受体位于细胞内质网膜上,起着负调控乙烯反应的作用[2]。目前,植物乙烯信号转导通路探明:在CU+的参与下,乙烯分子结合乙烯受体,致使CTR1(Constitutive triple response1),失活而减弱下游的EIN2(Ethylene Insensitive2)蛋白的磷酸化,EIN2蛋白c端被切割,c端移动到细胞核使其中的EIN3(Ethylene insensitive3)等因子感受到乙烯信号,促进下游的ERFs(Ethylene response factors)表达,激活乙烯反应[3]。在模式植物拟南芥中,研究者们发现了5种不同类型的乙烯受体,分别为:ETR1、ERS1、EIN4、ETR2和ERS2。每个受体都包含有N端跨膜区、GAF区和组氨酸(His)激酶结构域,ETR1、EIN4和ETR2受体中还包含一个接收域[4]。根据His激酶结构域中是否存在保守元素,将受体分为2个亚科:亚科1(ETR1和ERS1)和亚科2(ETR2、ERS2和EIN4)。前人已在对香蕉[5]、芒果[6]、木薯[7]、丹参[8]等的研究中证实了乙烯受体在果实成熟和花生长等组织生长发育方面的作用以及对逆境胁迫的抵御功能。茶树基因组学的巨大进步有效促进了研究人员对茶叶品质及适制性的理解,2017年云抗10号茶树基因组发表,开启了茶树基因组学研究序幕[9],2018年中国小叶种茶树舒茶早完成测序,舒茶早采用二代测序和三代PacBio序列补洞的策略获得了更高质量的茶树基因组[10]。科学家们已经分离出2个茶叶品种:中国小叶种茶树CSS(Camellia sinensis vars. sinensis)[11]和中国大叶种茶树CSA(Camellia sinensis vars. assamica)[9]的基因组序列,尤其是3个中国种(舒茶早[12]、碧云[13]和龙井43[11])的高精度染色体水平基因组序列已经被科学家们研究获得。但至今鲜有有关茶树乙烯受体基因方面的研究报告。【本研究切入点】前人研究结果表明乙烯受体基因对植物生长发育有内部调控作用。但至今鲜有茶树乙烯受体基因的有关研究报道。【拟解决的关键问题】本研究通过对茶树ETR基因家族进行生物信息学分析,预测其潜在分子功能,明确乙烯受体在茶树发育过程中的动态变化,初步了解家族各成员功能,筛选主要参与茶树组织发育过程ETR基因家族成员,利用前期基因家族的研究及qRT-PCR技术研究在非生物和外源激素胁迫下ETR基因家族各成员相对表达量变化,为进一步探明茶树乙烯受体各成员功能奠定基础。
1. 材料与方法
1.1 供试材料
试验所用材料于福建农林大学茶学福建省高校重点实验室采集。供试原料为相同栽培条件下生长状态良好的二年生铁观音盆栽茶树。原料于2019年4月参照陈丹(2017)等[14]的方法进行了以下处理:
1)低温处理:将在适宜温度(24±2) ℃下生长的原料植株移到人工气候箱中发育,温度设置为4 ℃,湿度为60%~70%,光照时间14 h,黑暗时间10 h。取低温处理后0、4、24、48 h的顶端第2、3片茶树成熟混合叶。
2)植物生长调节剂处理:制备新鲜的GA、MeJA和ABA溶液,提取100 μmol·L−1喷洒于不同植株[15],取各处理后0、6、24、48 h的顶端第2、3片茶树成熟混合叶。
将以上试验材料每个处理取3次重复,用锡纸包裹标记,液氮速冻后存于−80 ℃冰箱中。
1.2 试验方法
1.2.1 CsETR基因家族成员鉴定
首先在茶树基因组数据库TPIA(http://tpia.teaplant.org)[16]中下载茶树ETR基因蛋白序列,使用SMART(http://smart.embl-heidelberg.de/)[17]和CDD(https://www.ncbi.nlm.nih.gov/cdd/)[18]在线网站查找鉴定到的潜在基因的ETR保守结构域,与完整的ETR基因结构域进行比对,以筛选准确的基因家族成员。采用ExPASY(http://www.expasy.org/)[19]和WoLF PSORT(https://wolfpsort.hgc.jp/)[20]在线网站分析CsETR家族成员的蛋白质理化性质并预测其亚细胞定位。
1.2.2 CsETR基因家族系统发育树构建
采用MEGA7.0软件对CsETR基因进行保守序列分析,并于TAIR(https://www.arabidopsis.org/)网站和NCBI(https://www.ncbi.nlm.nih.gov/genbank/)网站下载拟南芥和番茄的乙烯受体基因序列,使用MEGA7.0软件构建系统进化树[21]。
1.2.3 CsETR基因成员结构和保守结构域分析
从TPIA数据库中下载CsETR基因的外显子和内含子数据文件,利用GSDS(http://gsds.cbi.pku.edu.cn/)[22]在线网站制作CsETR家族成员的基因结构图,包含外显子和内含子数目。使用MEME5.0.2(http://meme-suit.org/tools/meme)在线软件分析CsETR基因蛋白序列的保守基序[23]。
1.2.4 CsETR基因的顺式作用元件分析
从TPIA数据库中提取CsETR基因起始位置上游1000 bp序列,利用Editra软件查找启动子顺式作用元件,并于TBtools软件进行分析[24]。
1.2.5 实时荧光定量PCR分析
茶树叶片总RNA使用北京天根多糖多酚植物总RNA提取试剂盒提取,参照北京全式金试剂盒合成茶树乙烯受体cDNA用于qRT-PCR[10]。CsETR家族成员引物设计在primer3plus(http://www.primer3plus.com/)在线网站上进行(表1),内参基因选用CsGAPDH(登录号GE651107)。qRT-PCR反应在荧光定量PCR仪上进行。反应体系及程序参照实验室前期方法,使用2−ΔΔCt方法计算基因相对表达量[25],并使用TBtools绘制热图。
表 1 茶树CsETR家族成员引物序列Table 1. Primer sequence of CsETRs in tea plants基因名称
Gene name上游引物
Forward primers
(5′→3′)下游引物
Reverse primer
(5′→3′)ERS1-1 AATGGCCTGCTGATGAGCTT CACAGAAGATCCAGCAGGCA ERS1-2 GAGGATGGCAGCCTTCAACT AGTACATCCCTTGCCAAGCC ERS1-3 GGCACCAGATTTACCTGCCT GCTGAGCTTTCTGCAACACC EIN4 AATTGTGCCGTTTGGATGCC GAGTTTCTTGCCTGGCTTGC ETR2-1 GCAAATCGCTTGATCGGCAT CCATCATTGCGCTCTGCTTC ETR2-2 ATAAAGGAAGCCGCTTGCCT TCCCGGTTCAGAGATGCCTA CsGAPDH TTGGCATCGTTGAGGGTCT CAGTGGGAACAGGAAAGC 2. 结果与分析
2.1 CsETR基因家族成员的鉴定
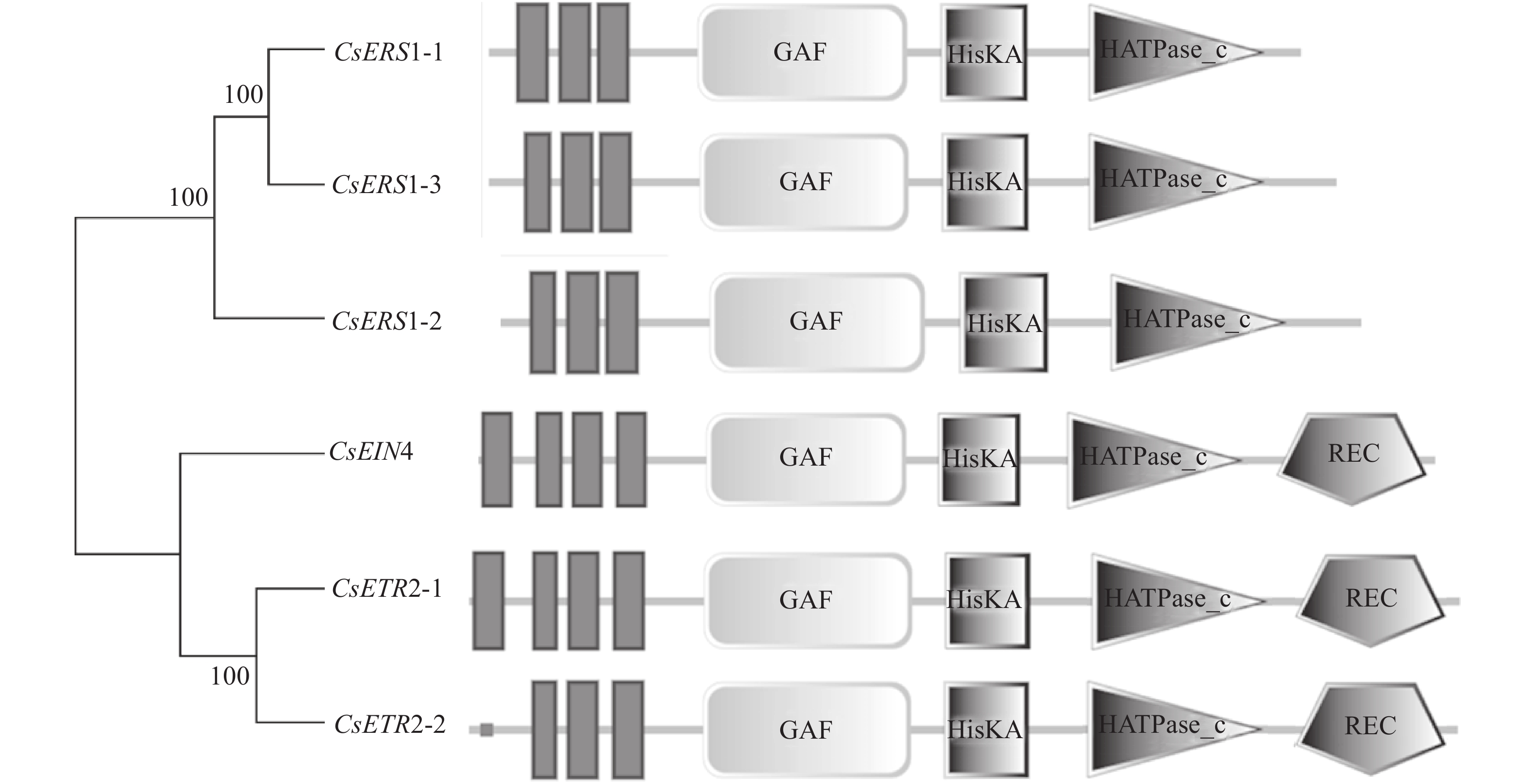
为准确鉴定CsETR家族成员,本研究采用了Blast比对方法,并将鉴定到的潜在CsETR家族成员提交至SMART和CDD网站再次进行鉴定,最终获得6个CsETR家族成员,分别命名为CsERS1-1、CsERS1-2、CsERS1-3、CsEIN4、CsETR2-1、CsETR2-2(图1)。采用ExPASY和WoLF PSORT在线网站分析6个家族成员各项因子的理化性质(表2)。通过蛋白质理化分析后得出结果,CsETR的ORF氨基酸长度为614 aa(CsERS1-1)~763 aa(CsETR2-1),分子量为68.63~85.56 kD,等电点大小为6.09(CsERS1-1)~7.57(CsETR2-2),亲水性为0.014~0.250,表明CsETR基因蛋白为碱性疏水蛋白;通过亚细胞定位预测分析后发现,CsETR基因蛋白均位于质膜上。
表 2 茶树CsETR基因家族的序列特征Table 2. Characteristics of CsETR sequences in tea plants基因
Gene name基因组编号
Genome ID编码序列长度
CDS/bp开放阅读框长度
ORF/aa分子量
MW/kD等电点
pI平均亲水性
Grand average of hydropathicity亚细胞定位
Subcellular localizationCsERS1-1 TEA017521 1845 614 68.63 6.09 0.162 质膜 Plasma membrane CsERS1-2 TEA032252 1923 640 71.71 7.02 0.144 质膜 Plasma membrane CsERS1-3 TEA012203 1932 643 71.89 6.67 0.250 质膜 Plasma membrane CsEIN4 TEA025082 2292 763 85.56 7.27 0.014 质膜 Plasma membrane CsETR2-1 TEA002824 2292 763 84.73 6.32 0.077 质膜 Plasma membrane CsETR2-2 TEA020178 2292 763 85.56 7.57 0.054 质膜 Plasma membrane 2.2 CsETR家族成员的系统发育和分类分析
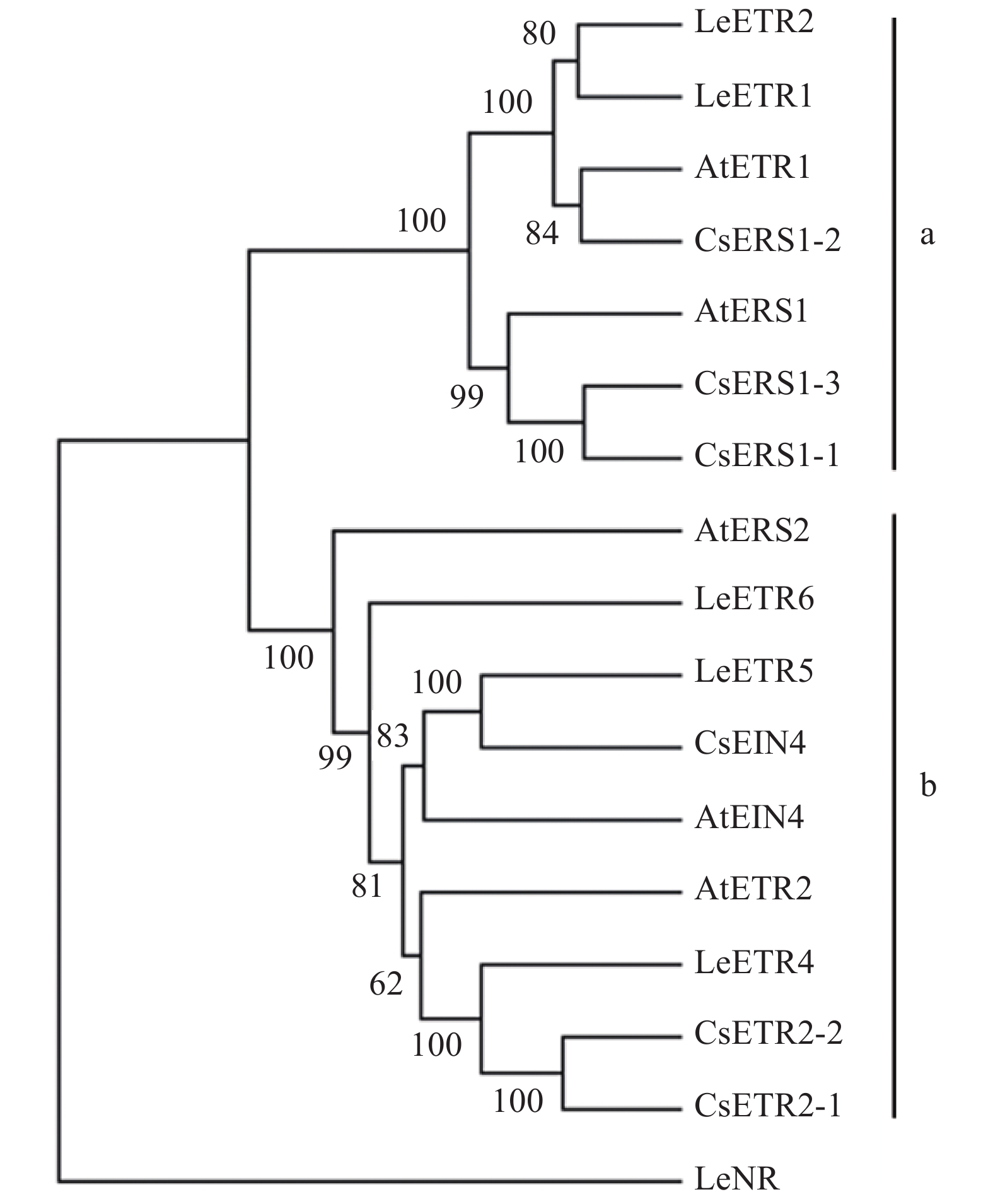
为了解ETR蛋白之间的进化关系,本研究选取了3个典型物种的17个ETR蛋白用来构建系统进化树。共采用6个茶树乙烯受体基因、6个番茄乙烯受体基因和5个拟南芥乙烯受体基因来构建系统发育树(图2)。ETR家族总共可分为2个亚家族,分别为ETR家族和ERS家族。另外,番茄的变异受体基因Nr单独形成一组,与其他2个亚家族功能有所区别。
2.3 CsETR家族成员结构和保守结构域分析
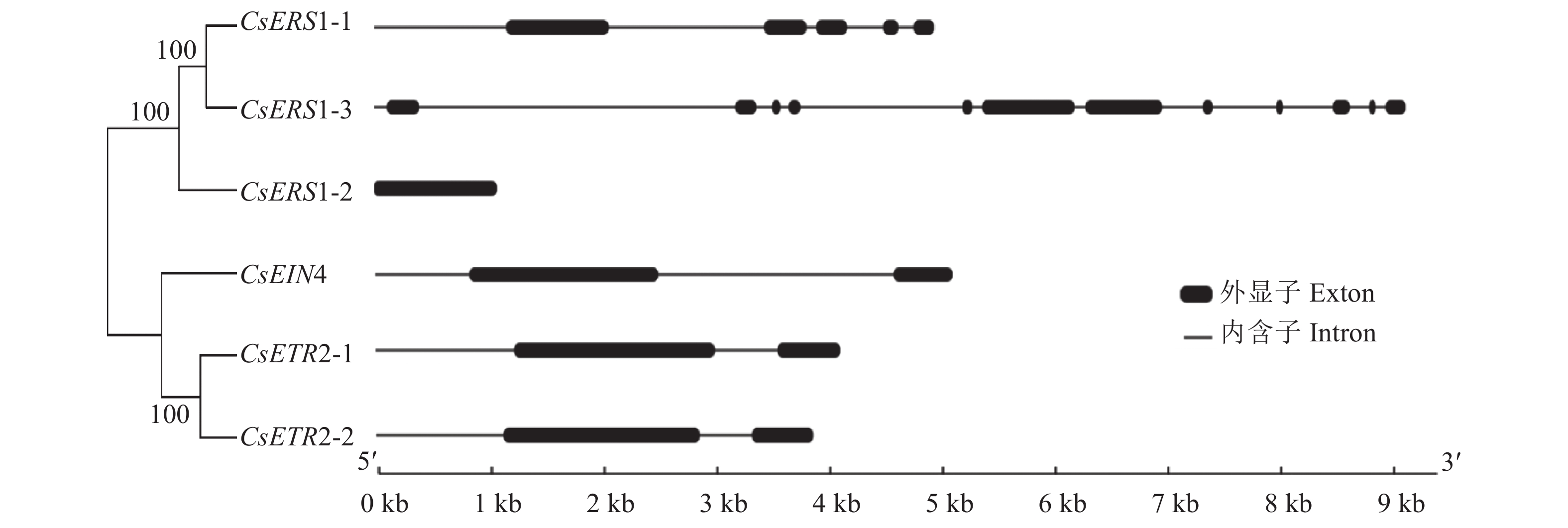
为分析CsETR基因家族成员结构的多样性,本文通过在GSDS网站上制作基因结构图来比较乙烯受体家族的内含子/外显子数量(图3),通过比较这6个成员的基因结构发现,外显子的数量从1(ERS1-2)至12(ERS1-3)个不等,差别较大,其中ERS1-3基因序列最长,超过了10000 bp,ERS1-2除外显子外,无内含子的存在,其他成员内含子均被外显子中断隔开。除此之外,发现ETR亚家族的基因序列分布相似,外显子内含子数量均为2个,而ERS亚家族基因序列分布不均。
利用分析网站MEME预测发现了ETR家族的6个保守基序(图4)和基因的保守基序分布及长度,结果显示所有家族成员都有相同的保守基序,且2个亚家族之间的分布在分布区域及长度上都十分相似。说明ETR家族成员基因功能较为保守,2个亚家族分别有着相似的遗传功能。
2.4 CsETR基因家族启动子顺势元件分析
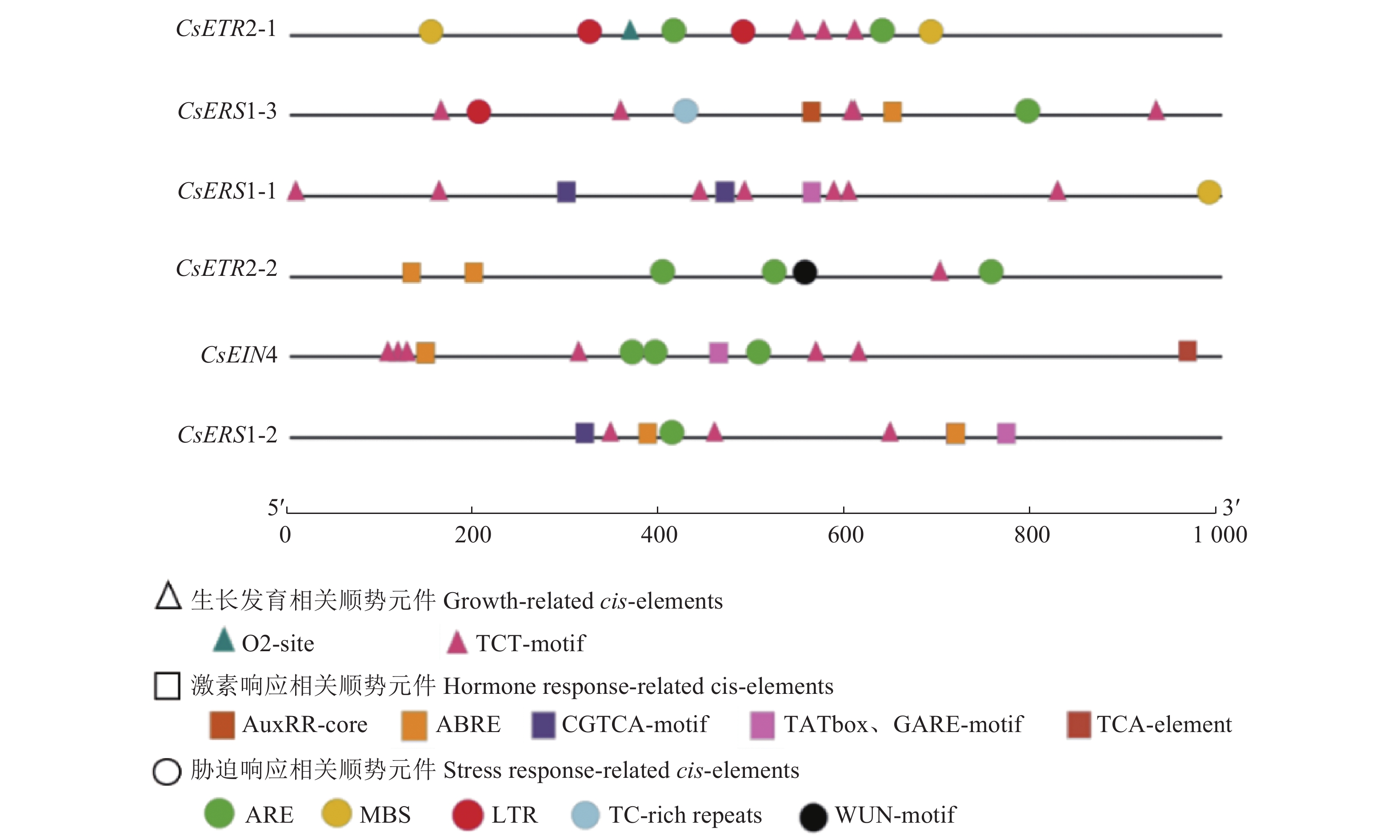
启动子顺势元件对于调控基因表达发挥着重要作用。CsETR基因启动子顺式作用元件使用Plant-CARE在线工具分析(图5)。最终获得3类顺势元件,与茶树生长相关的顺势元件:玉米醇溶蛋白代谢调控(O2-site,1个)、光响应(TCT-motif,25个);胁迫相应元件:厌氧响应(ARE,10个)、干旱响应(MBS,3个)、低温响应(LTR,3个)、防御和压力响应(TC-rich repeats,1个)、创伤响应(WUN-motif,1个)以及激素响应元件:包括生长素(AuxRR-core,1个)、脱落酸(ABmotif,1个)以及激素响应元件:包括生长素(AuxRR-TATbox、GARE-motif,3个)、水杨酸(TCA-element,1个)。由图可知,在ETR家族中6个成员均含有大量顺势元件,其中CsETR2-1含有1个O2-site,CsETR1-3含有1个AuxRR-core,CsETR2-2含有1个WUN-motif。以上结果表明,CsETR家族在茶树的生长发育、激素和外界胁迫响应中起着重要作用。
2.5 CsETR家族成员在不同组织中的表达模式
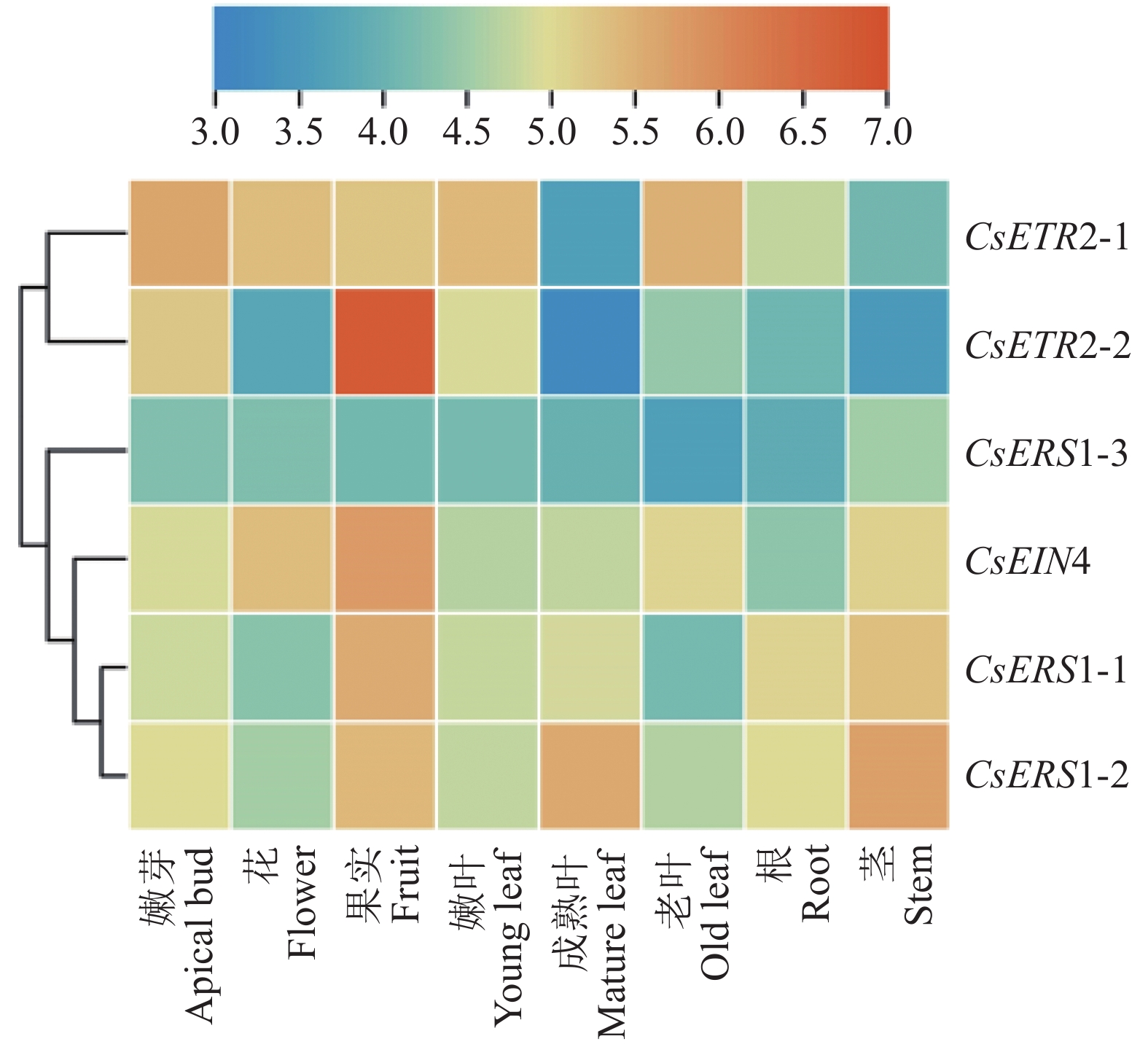
为研究CsETR基因家族对茶树组织生长发育的功能,从TPIA数据库中下载8种茶树组织表达数据,利用TBtools软件分析其在不同组织中的表达模式(图6)。根据结果可分析出:ETR家族在果实中的表达最为明显,茎和顶芽次之,在根和老叶部分表达相对较低,表明CsETR家族对茶树的果实发育起着重要作用。分析各个基因家族成员的组织表达量发现,CsETR家族有着较为明显的组织表达特异性,其中CsERS1-1、CsEIN4和CsETR2-2基因在果实中表达量较高,说明这3个基因在乙烯对果实成熟的影响过程中扮演着重要角色,而CsERS1-2、CsERS1-1在茎组织有较高表达,CsETR2-2在成熟叶中表达量较低。
2.6 CsETR家族在4种处理下的表达模式
为探究CsETR基因家族在激素和胁迫下的生理反应,鉴定外界胁迫和激素影响下的乙烯受体表达,本研究使用荧光定量技术测定了CsETR家族6个成员在低温(4 ℃)、赤霉素、脱落酸、茉莉酸处理下的相对表达量(图7)。在对低温4 ℃诱导下的试验材料进行PCR分析后得出其不同时段的基因表达量,发现CsETR基因家族的6个成员表达量均呈现上调趋势,其中CsERS1-1表达量上调幅度最大,CsERS1-3较小,大部分基因在低温诱导48 h后的表达量最高。分别对GA、MeJA、ABA处理后试验材料基因表达量进行分析,在GA的影响下,除去EIN4基因的表达量略有波动外,大部分ETR基因家族成员表达量均呈上调趋势,其中CsERS1-3的上调幅度最大,而CsERS1-1在6~48 h的基因表达量差别不大。在JA影响下的ETR基因表达量均呈现上调趋势,且CsERS1-3的上调幅度亦位于首位。在ABA的影响下,除CsETR2-2基因的表达量呈下调趋势外,其他家族成员都呈上调趋势,其中CsERS1-3的上调幅度依旧稳居首位。然而,ETR基因家族成员在ABA处理6 h后的表达量变化均难以测定,但内参处理下的基因表达量变化正常,推测其原因是ABA处理6 h对CsETR基因的影响不明显。
3. 讨论与结论
前人研究表明,ETR基因对植物生长,尤其是在成花及果实成熟阶段发挥着重要的调节作用。目前已经在拟南芥中鉴定到5个ETR成员[10]、番茄中鉴定出6个ETR成员[26]、甜瓜中鉴定出3个家族成员[27]、水稻中分离出5个基因[28]、烟草中鉴定出4个ETR家族成员[29]。在对ETR基因的进化史研究发现,ETR家族成员中,类ETR1受体首先进化,可能伴随着茶藻进化过程。乙烯反应在从藻类向陆地环境过渡期间可能很重要,对乙烯应对外界环境的进化研究有贡献[30]。然而,目前尚无对茶树ETR家族的研究。本研究在茶树基因组数据的基础上,根据其蛋白质结构鉴定出了6个ETR成员,分析结果介于拟南芥和番茄之间。通过对模式植物拟南芥ETR基因家族的分析发现,ETR基因分为2个亚家族,茶树ETR基因在这2个亚家族中均有分布,其中亚家族1中ERS基因有3个。其中,CsETR2-1和CsETR2-2与LeETR4同源性最为接近;CsERS1-1和CsERS1-3与AtERS1同源性最近,表明它们可能行使了相同的功能。这些不同的主要序列,存在区域和表现出激酶活性的类型,可能表明受体间的功能差异性。在分析ETR家族结构之后,发现ETR基因外显子-内含子高度保守,外显子数目为1~12个,同一组间的外显子数目相似,说明同一组基因间有着功能的相似之处。而在MEME网站上分析CsETR蛋白质的基序发现,6个基因都存在相同的6个基序,且相同组的距离几乎相同,说明2个亚家族有着相似的遗传功能。
乙烯受体是乙烯信号转导通路中的第一步,对乙烯引起的植物生理反应有着重要的作用。研究表明,ETR基因对于调节植物果实成熟和花衰老有着关键作用。不同的乙烯受体,即使结构相似,在同一或不同植物中对于植物生长阶段的表达水平与模式都有差异,如在同一株月季中,亚家族1的2个成员RhETR1和RhETR3结构相似,但在切花开放过程中,2个基因的表达却完全相反,RhETR1上调,RhETR3则下调。在2个不同植物番茄和甜瓜中,番茄LeETR1与甜瓜Cm-ETR1同源,在果实成熟时,LeETR1表达不受乙烯合成影响,而Cm-ETR1的表达随内源乙烯的合成同比变化[31]。ETR基因负调控乙烯信号转导,即上调ETR基因的表达,植物的乙烯敏感性就降低,反之升高。因此,可以通过调节ETR基因的表达来调控植物对乙烯的敏感性,如在水稻培育过程中通过上调ETR2基因的表达来降低转基因植株的敏感性,从而延迟水稻开花[32]。本研究发现CsETR家族在茶树果实中高表达,尤其是CsERS1-1、CsEIN4、CsETR2-2,说明这些基因对于调控茶果成熟起着重要作用,其次,ERS亚家族对茶树茎高表达,说明这些基因在茶树茎的发育中发挥作用。
研究基因启动子有助于了解植物基因表达模式的差异及其分子调控机制。前人研究发现,许多ETR基因自身含有响应非生物胁迫的顺式作用元件,如在番茄中SlETR6基因含有LTR、MBS、ARE、TCT-motif、ERF(乙烯应答)等顺式作用元件[33]。甘蔗的受体基因SoERS1中也含有干旱诱导的MYB结合位点和LTR元件,说明该基因的表达可能受干旱和高温等因素的调控[34]。同样,我们在对茶树的启动子研究当中,也发现了每个成员所携带的顺式作用元件,如ARE、TCT-motif、LTR、ABRE、AuxRR-core、CGTCA-motif等,大部分都与非生物胁迫和激素有关。在对低温及植物生长调节剂处理下的茶叶经PCR分析后发现,CsETR家族所有成员在低温胁迫下的表达量均呈上调趋势,这与启动子顺势元件分析结果相匹配。在GA、ABA、JA的诱导下,大部分家族成员基因表达量都呈上调趋势,而EIN4基因在GA处理6 h后达到最高值,说明EIN4基因在GA处理前期正调控反应最大,而ETR2-2基因在ABA处理下表达量呈下调趋势,可能是ETR2-2基因在ABA的影响下表达收到抑制。在CsETR家族基因中,CsERS1-3在植物生长调节剂的影响下表达量上调幅度最大,说明ERS1-3在对植物生长调节剂的响应中可能发挥重要的正调控作用。
本研究首次开展了对茶树全基因组数据库中CsETR基因家族的鉴定分析,鉴定出6个家族成员,分为2个亚科,对其保守结构域、启动子顺势元件分析以及在低温、外源激素胁迫下的反应进行分析后得出初步结论:茶树乙烯受体基因家族与茶树的组织生长发育、非生物胁迫和外源激素影响有着密切的关系,后续还需进行细致的试验具体验证这些基因的生物学功能,并为茶树新品种选育提供参考。
-
表 1 茶树CsETR家族成员引物序列
Table 1 Primer sequence of CsETRs in tea plants
基因名称
Gene name上游引物
Forward primers
(5′→3′)下游引物
Reverse primer
(5′→3′)ERS1-1 AATGGCCTGCTGATGAGCTT CACAGAAGATCCAGCAGGCA ERS1-2 GAGGATGGCAGCCTTCAACT AGTACATCCCTTGCCAAGCC ERS1-3 GGCACCAGATTTACCTGCCT GCTGAGCTTTCTGCAACACC EIN4 AATTGTGCCGTTTGGATGCC GAGTTTCTTGCCTGGCTTGC ETR2-1 GCAAATCGCTTGATCGGCAT CCATCATTGCGCTCTGCTTC ETR2-2 ATAAAGGAAGCCGCTTGCCT TCCCGGTTCAGAGATGCCTA CsGAPDH TTGGCATCGTTGAGGGTCT CAGTGGGAACAGGAAAGC 表 2 茶树CsETR基因家族的序列特征
Table 2 Characteristics of CsETR sequences in tea plants
基因
Gene name基因组编号
Genome ID编码序列长度
CDS/bp开放阅读框长度
ORF/aa分子量
MW/kD等电点
pI平均亲水性
Grand average of hydropathicity亚细胞定位
Subcellular localizationCsERS1-1 TEA017521 1845 614 68.63 6.09 0.162 质膜 Plasma membrane CsERS1-2 TEA032252 1923 640 71.71 7.02 0.144 质膜 Plasma membrane CsERS1-3 TEA012203 1932 643 71.89 6.67 0.250 质膜 Plasma membrane CsEIN4 TEA025082 2292 763 85.56 7.27 0.014 质膜 Plasma membrane CsETR2-1 TEA002824 2292 763 84.73 6.32 0.077 质膜 Plasma membrane CsETR2-2 TEA020178 2292 763 85.56 7.57 0.054 质膜 Plasma membrane -
[1] HAZRA A, DASGUPTA N, SENGUPTA C, et al. Next generation crop improvement program: Progress and prospect in tea (Camellia sinensis (L.) O. Kuntze) [J]. Annals of Agrarian Science, 2018, 16(2): 128−135.
[2] HUA J, MEYEROWITZ E M. Ethylene responses are negatively regulated by a receptor gene family in Arabidopsis thaliana [J]. Cell, 1998, 94(2): 261−271. DOI: 10.1016/S0092-8674(00)81425-7
[3] WEN X, ZHANG C L, JI Y S, et al. Activation of ethylene signaling is mediated by nuclear translocation of the cleaved EIN2 carboxyl Terminus [J]. Cell Research, 2012, 22(11): 1613−1616. DOI: 10.1038/cr.2012.145
[4] HUA J, SAKAI H, NOURIZADEH S, et al. EIN4 and ERS2 are members of the putative ethylene receptor gene family in Arabidopsis [J]. The Plant Cell, 1998, 10(8): 1321−1332. DOI: 10.1105/tpc.10.8.1321
[5] 冯斗, 张春发, 张颖. 香蕉乙烯受体基因cDNA的克隆及其表达分析 [J]. 热带作物学报, 2004, 25(1):6−10. DOI: 10.3969/j.issn.1000-2561.2004.01.002 FENG D, ZHANG C F, ZHANG Y. Ublished by Evert (2001). Slot Northern hybridization with the labeled probe of PGCHI indicated [J]. Chinese Journal of Tropical Crops, 2004, 25(1): 6−10.(in Chinese) DOI: 10.3969/j.issn.1000-2561.2004.01.002
[6] 李丽, 孙健, 易萍, 等. 芒果乙烯受体ETR1和ERS1基因的克隆及序列分析 [J]. 南方农业学报, 2018, 49(6):1053−1060. DOI: 10.3969/j.issn.2095-1191.2018.06.02 LI L, SUN J, YI P, et al. Cloning and sequence analysis of ethylene receptor ETR1 and ERS1 genes from Mango [J]. Journal of Southern Agriculture, 2018, 49(6): 1053−1060.(in Chinese) DOI: 10.3969/j.issn.2095-1191.2018.06.02
[7] 陈志晟, 颜彦, 赵思涵, 等. 木薯ETR1基因克隆及表达分析 [J]. 南方农业学报, 2020, 51(1):19−26. DOI: 10.3969/j.issn.2095-1191.2020.01.003 CHEN Z S, YAN Y, ZHAO S H, et al. Cloning and expression analysis of MeETR1 gene in cassava [J]. Journal of Southern Agriculture, 2020, 51(1): 19−26.(in Chinese) DOI: 10.3969/j.issn.2095-1191.2020.01.003
[8] 王睿, 吴佳文, 毛仁俊, 陈国梁, 白朕卿. 丹参乙烯受体SmETR1基因的克隆及序列分析[J/OL]. 基因组学与应用生物学: 1−9[2021-05-06]. http://kns.cnki.net/kcms/detail/45.1369.Q.20210317.1532.002.html. WANG R, WU J W, ZHAO S H, et al. Cloning and sequence analysis of ethylene receptor SmETR1 gene from Salvia miltiorrhiza[J/OL]. Genomics and Applied Biology, 1-9[2021-05-06]. http://kns.cnki.net/kcms/detail/45.1369.Q.20210317.1532.002.html.
[9] XIA E H, ZHANG H B, SHENG J, et al. The tea tree genome provides insights into tea flavor and independent evolution of caffeine biosynthesis [J]. Molecular Plant, 2017, 10(6): 866−877. DOI: 10.1016/j.molp.2017.04.002
[10] WEI C L, YANG H, WANG S B, et al. Draft genome sequence of Camellia sinensis var. sinensis provides insights into the evolution of the tea genome and tea quality [J]. PNAS, 2018, 115(18): E4151−E4158. DOI: 10.1073/pnas.1719622115
[11] WANG X C, FENG H, CHANG Y X, et al. Population sequencing enhances understanding of tea plant evolution [J]. Nature Communications, 2020, 11: 4447. DOI: 10.1038/s41467-020-18228-8
[12] CHEN J D, ZHENG C, MA J Q, et al. The chromosome-scale genome reveals the evolution and diversification after the recent tetraploidization event in tea plant [J]. Horticulture Research, 2020, 7: 63. DOI: 10.1038/s41438-020-0288-2
[13] ZHANG Q J, LI W, LI K, et al. The chromosome-level reference genome of tea tree unveils recent bursts of non-autonomous LTR retrotransposons in driving genome size evolution [J]. Molecular Plant, 2020, 13(7): 935−938. DOI: 10.1016/j.molp.2020.04.009
[14] 陈丹, 俞滢, 岳川, 等. 茶树△12-脂肪酸去饱和酶基因FAD2和FAD6的克隆与表达分析 [J]. 茶叶科学, 2017, 37(6):541−550. DOI: 10.3969/j.issn.1000-369X.2017.06.001 CHEN D, YU Y, YUE C, et al. Cloning and expression analysis of △12-fatty acid desaturase in tea plants [J]. Journal of Tea Science, 2017, 37(6): 541−550.(in Chinese) DOI: 10.3969/j.issn.1000-369X.2017.06.001
[15] 王鹏杰, 郑玉成, 林浥, 等. 茶树GRF基因家族的全基因组鉴定及表达分析 [J]. 西北植物学报, 2019, 39(3):413−421. WANG P J, ZHENG Y C, LIN Y, et al. Genome-wide identification and expression analysis of GRF gene family in Camellia sinensis [J]. Acta Botanica Boreali-Occidentalia Sinica, 2019, 39(3): 413−421.(in Chinese)
[16] XIA E H, LI F D, TONG W, et al. Tea Plant Information Archive: A comprehensive genomics and bioinformatics platform for tea plant [J]. Plant Biotechnology Journal, 2019, 17(10): 1938−1953. DOI: 10.1111/pbi.13111
[17] SCHULTZ J, MILPETZ F, BORK P, et al. SMART, a simple modular architecture research tool: Identification of signaling domains [J]. PNAS, 1998, 95(11): 5857−5864. DOI: 10.1073/pnas.95.11.5857
[18] MARCHLER-BAUER A, DERBYSHIRE M K, GONZALES N R, et al. CDD: NCBI's conserved domain database [J]. Nucleic Acids Research, 2015, 43(Database issue): D222−D226.
[19] DUVAUD S, GABELLA C, LISACEK F, et al. Expasy, the Swiss bioinformatics resource portal, as designed by its users [J]. Nucleic Acids Research, 2021, 49(W1): W216−W227. DOI: 10.1093/nar/gkab225
[20] HORTON P, PARK K J, OBAYASHI T, et al. WoLF PSORT: Protein localization predictor [J]. Nucleic Acids Research, 2007, 35(Web Server issue): W585−W587.
[21] NEWMAN L, DUFFUS A L J, LEE C. Using the free program MEGA to build phylogenetic trees from molecular data [J]. The American Biology Teacher, 2016, 78(7): 608−612. DOI: 10.1525/abt.2016.78.7.608
[22] HU B, JIN J P, GUO A Y, et al. GSDS 2.0: An upgraded gene feature visualization server [J]. Bioinformatics, 2015, 31(8): 1296−1297. DOI: 10.1093/bioinformatics/btu817
[23] BAILEY T L, BODEN M, BUSKE F A, et al. MEME Suite: Tools for motif discovery and searching [J]. Nucleic Acids Research, 2009, 37(Suppl_2): W202−W208.
[24] 陈雪津, 王鹏杰, 郑玉成, 等. 茶树BES1转录因子全基因组鉴定与分析 [J]. 西北植物学报, 2019, 39(5):876−885. CHEN X J, WANG P J, ZHENG Y C, et al. Genome-wide identification and analysis of BES1 transcription factor family in Camellia sinensis [J]. Acta Botanica Boreali-Occidentalia Sinica, 2019, 39(5): 876−885.(in Chinese)
[25] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCT method [J]. Methods, 2001, 25(4): 402−408. DOI: 10.1006/meth.2001.1262
[26] TIEMAN D M, KLEE H J. Differential expression of two novel members of the tomato ethylene-receptor family [J]. Plant Physiology, 1999, 120(1): 165−172. DOI: 10.1104/pp.120.1.165
[27] 郭呈宇, 柳俊, 李园磊, 等. 甜瓜果实成熟相关基因家族的全基因组鉴定及分析 [J]. 华北农学报, 2019, 34(2):35−43. DOI: 10.7668/hbnxb.201750903 GUO C Y, LIU J, LI Y L, et al. Genome-wide identification and analysis of gene families related to fruit ripening of melon [J]. Acta Agriculturae Boreali-Sinica, 2019, 34(2): 35−43.(in Chinese) DOI: 10.7668/hbnxb.201750903
[28] WATANABE H, SAIGUSA M, SHU H S, et al. Cloning of a cDNA encoding an ETR2-like protein (Os-ERL1) from deep water rice (Oryza sativa L.) and increase in its mRNA level by submergence, ethylene, and gibberellin treatments [J]. Journal of Experimental Botany, 2004, 55(399): 1145−1148. DOI: 10.1093/jxb/erh110
[29] XIE C, ZHANG Z G, ZHANG J S, et al. Spatial expression and characterization of a putative ethylene receptor protein NTHK1 in tobacco [J]. Plant and Cell Physiology, 2002, 43(7): 810−815. DOI: 10.1093/pcp/pcf095
[30] GALLIE D R. Ethylene receptors in plants-why so much complexity? [J]. F1000Prime Reports, 2015, 7: 39.
[31] ZHOU D B, KALAITZIS P, MATTOO A K, et al. The mRNA for an ETR1 homologue in tomato is constitutively expressed in vegetative and reproductive tissues [J]. Plant Molecular Biology, 1996, 30(6): 1331−1338. DOI: 10.1007/BF00019564
[32] WURIYANGHAN H, ZHANG B, CAO W H, et al. The ethylene receptor ETR2 delays floral transition and affects starch accumulation in rice [J]. The Plant Cell, 2009, 21(5): 1473−1494. DOI: 10.1105/tpc.108.065391
[33] 苏丽艳. 番茄SlETR6基因的克隆及非生物胁迫下的表达分析 [J]. 华北农学报, 2019, 34(1):19−25. DOI: 10.7668/hbnxb.201751117 SU L Y. Cloning and expression analysis of ethylene receptor gene SlETR6 in Solanum lycopersicum under abiotic stress [J]. Acta Agriculturae Boreali-Sinica, 2019, 34(1): 19−25.(in Chinese) DOI: 10.7668/hbnxb.201751117
[34] 黄静丽. 甘蔗乙烯受体基因SoERS1表达、转化及启动子序列的研究[D]. 南宁: 广西大学, 2013. HUANG J L. Study on sugarcane ethylene receptor gene SoERS1Expression, genetic transformation and promoter sequences[D]. Nanning: Guangxi University, 2013. (in Chinese)
-
期刊类型引用(1)
1. 青亚林,侯炳豪,张宇航,曾珊珊,高婷,叶乃兴. 茶树GATA基因家族的全基因组鉴定及表达分析. 江苏农业科学. 2024(09): 42-50 .  百度学术
百度学术
其他类型引用(0)




 下载:
下载: